+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of succinate receptor bound to succinate acid coupling MiniGsq | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of angiotensin metabolic process / renin secretion into blood stream / positive regulation of chemotaxis / macrophage activation involved in immune response / Class A/1 (Rhodopsin-like receptors) / PKA activation in glucagon signalling / hair follicle placode formation / developmental growth / D1 dopamine receptor binding / intracellular transport ...regulation of angiotensin metabolic process / renin secretion into blood stream / positive regulation of chemotaxis / macrophage activation involved in immune response / Class A/1 (Rhodopsin-like receptors) / PKA activation in glucagon signalling / hair follicle placode formation / developmental growth / D1 dopamine receptor binding / intracellular transport / vascular endothelial cell response to laminar fluid shear stress / renal water homeostasis / Hedgehog 'off' state / adenylate cyclase-activating adrenergic receptor signaling pathway / energy homeostasis / activation of adenylate cyclase activity / regulation of insulin secretion / cellular response to glucagon stimulus / adenylate cyclase activator activity / trans-Golgi network membrane / negative regulation of inflammatory response to antigenic stimulus / electron transport chain / G protein-coupled receptor activity / bone development / G-protein beta/gamma-subunit complex binding / platelet aggregation / cognition / Olfactory Signaling Pathway / Activation of the phototransduction cascade / G beta:gamma signalling through PLC beta / Presynaptic function of Kainate receptors / Thromboxane signalling through TP receptor / G protein-coupled acetylcholine receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / G-protein activation / response to calcium ion / Activation of G protein gated Potassium channels / Inhibition of voltage gated Ca2+ channels via Gbeta/gamma subunits / Prostacyclin signalling through prostacyclin receptor / positive regulation of inflammatory response / G beta:gamma signalling through CDC42 / Glucagon signaling in metabolic regulation / G beta:gamma signalling through BTK / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) / ADP signalling through P2Y purinoceptor 12 / Sensory perception of sweet, bitter, and umami (glutamate) taste / photoreceptor disc membrane / Glucagon-type ligand receptors / Adrenaline,noradrenaline inhibits insulin secretion / Vasopressin regulates renal water homeostasis via Aquaporins / G alpha (z) signalling events / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / cellular response to catecholamine stimulus / ADORA2B mediated anti-inflammatory cytokines production / ADP signalling through P2Y purinoceptor 1 / G beta:gamma signalling through PI3Kgamma / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / adenylate cyclase-activating dopamine receptor signaling pathway / sensory perception of smell / GPER1 signaling / Inactivation, recovery and regulation of the phototransduction cascade / cellular response to prostaglandin E stimulus / glucose homeostasis / G-protein beta-subunit binding / heterotrimeric G-protein complex / G alpha (12/13) signalling events / sensory perception of taste / extracellular vesicle / signaling receptor complex adaptor activity / signaling receptor activity / Thrombin signalling through proteinase activated receptors (PARs) / positive regulation of cold-induced thermogenesis / G protein activity / GTPase binding / retina development in camera-type eye / Ca2+ pathway / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / fibroblast proliferation / G alpha (i) signalling events / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / G alpha (s) signalling events / phospholipase C-activating G protein-coupled receptor signaling pathway / G alpha (q) signalling events / Ras protein signal transduction / periplasmic space / Extra-nuclear estrogen signaling / electron transfer activity / cell population proliferation / G protein-coupled receptor signaling pathway / iron ion binding / lysosomal membrane / GTPase activity / synapse / heme binding / protein-containing complex binding / GTP binding / signal transduction / extracellular exosome / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Oplophorus gracilirostris (甲殻類) Oplophorus gracilirostris (甲殻類) | |||||||||
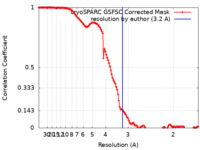
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Wang TX / Tang WQ / Li FH / Wang JY | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2024 ジャーナル: Cell Res / 年: 2024タイトル: Molecular activation and G protein coupling selectivity of human succinate receptor SUCR1. 著者: Tianxin Wang / Wenqin Tang / Xiaolei Zhu / Zhenyu Lv / Jiayan Chen / Yongze Li / Xiaoyu Sun / Haoyu Lv / Quanchang Gu / Fahui Li / Jiangyun Wang /  | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36486.map.gz emd_36486.map.gz | 167.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36486-v30.xml emd-36486-v30.xml emd-36486.xml emd-36486.xml | 17.9 KB 17.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36486_fsc.xml emd_36486_fsc.xml | 13.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36486.png emd_36486.png | 29.3 KB | ||
| Filedesc metadata |  emd-36486.cif.gz emd-36486.cif.gz | 6.4 KB | ||
| その他 |  emd_36486_half_map_1.map.gz emd_36486_half_map_1.map.gz emd_36486_half_map_2.map.gz emd_36486_half_map_2.map.gz | 164.9 MB 164.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36486 http://ftp.pdbj.org/pub/emdb/structures/EMD-36486 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36486 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36486 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jppMC  8jpnC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36486.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36486.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_36486_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
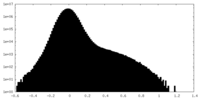
| 投影像・断面図 |
| ||||||||||||
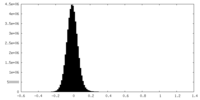
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36486_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
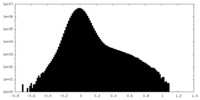
| 投影像・断面図 |
| ||||||||||||
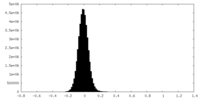
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GPCR complex
| 全体 | 名称: GPCR complex |
|---|---|
| 要素 |
|
-超分子 #1: GPCR complex
| 超分子 | 名称: GPCR complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Guanine nucleotide-binding protein G(s) subunit alpha isoforms short
| 分子 | 名称: Guanine nucleotide-binding protein G(s) subunit alpha isoforms short タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.245139 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGCLGNSKTE DQRNEEKAQR EANKKIEKQL QKDKQVYRAT HRLLLLGADN SGKSTIVKQM RILHGGSGGS GGTSGIFETK FQVDKVNFH MFDVGGQRDE RRKWIQCFND VTAIIFVVDS SDYNRLQEAL NLFKSIWNNR WLRTISVILF LNKQDLLAEK V LAGKSKIE ...文字列: MGCLGNSKTE DQRNEEKAQR EANKKIEKQL QKDKQVYRAT HRLLLLGADN SGKSTIVKQM RILHGGSGGS GGTSGIFETK FQVDKVNFH MFDVGGQRDE RRKWIQCFND VTAIIFVVDS SDYNRLQEAL NLFKSIWNNR WLRTISVILF LNKQDLLAEK V LAGKSKIE DYFPEFARYT TPEDATPEPG EDPRVTRAKY FIRDEFLRIS TASGDGRHYC YPHFTCAVDT ENARRIFNDC KD IILQMNL REYNLV UniProtKB: Guanine nucleotide-binding protein G(s) subunit alpha isoforms short |
-分子 #2: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1
| 分子 | 名称: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 41.055867 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MHHHHHHGSL LQSELDQLRQ EAEQLKNQIR DARKACADAT LSQITNNIDP VGRIQMRTRR TLRGHLAKIY AMHWGTDSRL LVSASQDGK LIIWDSYTTN KVHAIPLRSS WVMTCAYAPS GNYVACGGLD NICSIYNLKT REGNVRVSRE LAGHTGYLSC C RFLDDNQI ...文字列: MHHHHHHGSL LQSELDQLRQ EAEQLKNQIR DARKACADAT LSQITNNIDP VGRIQMRTRR TLRGHLAKIY AMHWGTDSRL LVSASQDGK LIIWDSYTTN KVHAIPLRSS WVMTCAYAPS GNYVACGGLD NICSIYNLKT REGNVRVSRE LAGHTGYLSC C RFLDDNQI VTSSGDTTCA LWDIETGQQT TTFTGHTGDV MSLSLAPDTR LFVSGACDAS AKLWDVREGM CRQTFTGHES DI NAICFFP NGNAFATGSD DATCRLFDLR ADQELMTYSH DNIICGITSV SFSKSGRLLL AGYDDFNCNV WDALKADRAG VLA GHDNRV SCLGVTDDGM AVATGSWDSF LKIWNGSSGG GGSGGGGSSG VSGWRLFKKI S UniProtKB: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1 |
-分子 #3: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2
| 分子 | 名称: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 7.861143 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MASNNTASIA QARKLVEQLK MEANIDRIKV SKAAADLMAY CEAHAKEDPL LTPVPASENP FREKKFFCAI L UniProtKB: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2 |
-分子 #4: Soluble cytochrome b562,Succinate receptor 1,Oplophorus-luciferin...
| 分子 | 名称: Soluble cytochrome b562,Succinate receptor 1,Oplophorus-luciferin 2-monooxygenase catalytic subunit タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Oplophorus gracilirostris (甲殻類) Oplophorus gracilirostris (甲殻類) |
| 分子量 | 理論値: 74.993492 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MKTIIALSYI FCLVFADYKD DDDAGRAHHH HHHHHHHENL YFQSGAPADL EDNWETLNDN LKVIEKADNA AQVKDALTKM RAAALDAQK ATPPKLEDKS PDSPEMKDFR HGFDILVGQI DDALKLANEG KVKEAQAAAE QLKTTRNAYI QKYLLEVLFQ G PEFMLGIM ...文字列: MKTIIALSYI FCLVFADYKD DDDAGRAHHH HHHHHHHENL YFQSGAPADL EDNWETLNDN LKVIEKADNA AQVKDALTKM RAAALDAQK ATPPKLEDKS PDSPEMKDFR HGFDILVGQI DDALKLANEG KVKEAQAAAE QLKTTRNAYI QKYLLEVLFQ G PEFMLGIM AWNATCKNWL AAEAALEKYY LSIFYGIEFV VGVLGNTIVV YGYIFSLKNW NSSNIYLFNL SVSDLAFLCT LP MLIRSYA NGNWIYGDVL CISNRYVLHA NLYTSILFLT FISIDRYLII KYPFREHLLQ KKEFAILISL AIWVLVTLEL LPI LPLINP VITDNGTTCN DFASSGDPNY NLIYSMCLTL LGFLIPLFVM CFFYYKIALF LKQRNRQVAT ALPLEKPLNL VIMA VVIFS VLFTPYHVMR NVRIASRLGS WKQYQCTQVV INSFYIVTRP LAFLNSVINP VFYFLLGDHF RDMLMNQLRH NFKSL TSFS RWAHELLLSF REKAAAVFTL EDFVGDWEQT AAYNLDQVLE QGGVSSLLQN LAVSVTPIQR IVRSGENALK IDIHVI IPY EGLSADQMAQ IEEVFKVVYP VDDHHFKVIL PYGTLVIDGV TPNMLNYFGR PYEGIAVFDG KKITVTGTLW NGNKIID ER LITPDGSMLF RVTINS UniProtKB: Soluble cytochrome b562, Succinate receptor 1 |
-分子 #5: SUCCINIC ACID
| 分子 | 名称: SUCCINIC ACID / タイプ: ligand / ID: 5 / コピー数: 1 / 式: SIN |
|---|---|
| 分子量 | 理論値: 118.088 Da |
| Chemical component information |  ChemComp-SIN: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)