+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human sodium-dependent vitamin C transporter 1 bound to L-ascorbic acid in an inward-open state | |||||||||
 マップデータ マップデータ | FSC-weighted, sharpened and masked map by PostProcess | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Transporter / Membrane protein / Ascorbic acid / Vitamin C / Sodium / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nucleobase transport / L-ascorbate:sodium symporter activity / L-ascorbic acid transmembrane transporter activity / L-ascorbic acid transmembrane transport / nucleobase transmembrane transporter activity / dehydroascorbic acid transmembrane transporter activity / intracellular organelle / sodium ion transmembrane transporter activity / dehydroascorbic acid transport / Vitamin C (ascorbate) metabolism ...nucleobase transport / L-ascorbate:sodium symporter activity / L-ascorbic acid transmembrane transporter activity / L-ascorbic acid transmembrane transport / nucleobase transmembrane transporter activity / dehydroascorbic acid transmembrane transporter activity / intracellular organelle / sodium ion transmembrane transporter activity / dehydroascorbic acid transport / Vitamin C (ascorbate) metabolism / L-ascorbic acid metabolic process / urate transmembrane transporter activity / sodium ion transport / lung development / basal plasma membrane / brain development / response to toxic substance / apical plasma membrane / extracellular exosome / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
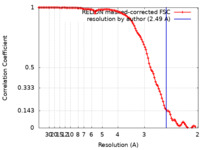
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.49 Å | |||||||||
 データ登録者 データ登録者 | Kobayashi TA / Kusakizako T / Nureki O | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Dimeric transport mechanism of human vitamin C transporter SVCT1. 著者: Takaaki A Kobayashi / Hiroto Shimada / Fumiya K Sano / Yuzuru Itoh / Sawako Enoki / Yasushi Okada / Tsukasa Kusakizako / Osamu Nureki /  要旨: Vitamin C plays important roles as a cofactor in many enzymatic reactions and as an antioxidant against oxidative stress. As some mammals including humans cannot synthesize vitamin C de novo from ...Vitamin C plays important roles as a cofactor in many enzymatic reactions and as an antioxidant against oxidative stress. As some mammals including humans cannot synthesize vitamin C de novo from glucose, its uptake from dietary sources is essential, and is mediated by the sodium-dependent vitamin C transporter 1 (SVCT1). Despite its physiological significance in maintaining vitamin C homeostasis, the structural basis of the substrate transport mechanism remained unclear. Here, we report the cryo-EM structures of human SVCT1 in different states at 2.5-3.5 Å resolutions. The binding manner of vitamin C together with two sodium ions reveals the counter ion-dependent substrate recognition mechanism. Furthermore, comparisons of the inward-open and occluded structures support a transport mechanism combining elevator and distinct rotational motions. Our results demonstrate the molecular mechanism of vitamin C transport with its underlying conformational cycle, potentially leading to future industrial and medical applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36201.map.gz emd_36201.map.gz | 2.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36201-v30.xml emd-36201-v30.xml emd-36201.xml emd-36201.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36201_fsc.xml emd_36201_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36201.png emd_36201.png | 112.5 KB | ||
| マスクデータ |  emd_36201_msk_1.map emd_36201_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-36201.cif.gz emd-36201.cif.gz | 6.9 KB | ||
| その他 |  emd_36201_half_map_1.map.gz emd_36201_half_map_1.map.gz emd_36201_half_map_2.map.gz emd_36201_half_map_2.map.gz | 6.7 MB 6.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36201 http://ftp.pdbj.org/pub/emdb/structures/EMD-36201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36201 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36201_validation.pdf.gz emd_36201_validation.pdf.gz | 825.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36201_full_validation.pdf.gz emd_36201_full_validation.pdf.gz | 825.2 KB | 表示 | |
| XML形式データ |  emd_36201_validation.xml.gz emd_36201_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_36201_validation.cif.gz emd_36201_validation.cif.gz | 17.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36201 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36201 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36201 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36201 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36201.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36201.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | FSC-weighted, sharpened and masked map by PostProcess | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.996 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_36201_msk_1.map emd_36201_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36201_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36201_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Human SVCT1 dimer in a substrate-bound inward-open state
+超分子 #1: Human SVCT1 dimer in a substrate-bound inward-open state
+分子 #1: Solute carrier family 23 member 1
+分子 #2: ASCORBIC ACID
+分子 #3: SODIUM ION
+分子 #4: Lauryl Maltose Neopentyl Glycol
+分子 #5: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
+分子 #6: CHOLESTEROL
+分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
+分子 #8: PALMITIC ACID
+分子 #9: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 25 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 3753 / 平均電子線量: 49.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)