+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Vibrio campbellii alpha-hemolysin | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Vibrio campbellii alpha-hemolysin / Pore-forming toxins (PFTs) / Calcium-dependent oligomerization / Membrane association / Structure-function relationship / TOXIN | |||||||||
| 機能・相同性 | :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Vibrio campbellii (バクテリア) Vibrio campbellii (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.06 Å | |||||||||
 データ登録者 データ登録者 | Wang CH / Yeh MK / Ho MC / Lin SM | |||||||||
| 資金援助 |  台湾, 2件 台湾, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural basis for calcium-stimulating pore formation of Vibrio α-hemolysin. 著者: Yu-Chuan Chiu / Min-Chi Yeh / Chun-Hsiung Wang / Yu-An Chen / Hsiang Chang / Han-You Lin / Meng-Chiao Ho / Shih-Ming Lin /  要旨: Vibrio α-hemolysins (αHLs) are β-pore-forming toxins secreted by Vibrio pathogens, crucial for the facilitation of bacterial infections through host cell lysis. These toxins are produced as ...Vibrio α-hemolysins (αHLs) are β-pore-forming toxins secreted by Vibrio pathogens, crucial for the facilitation of bacterial infections through host cell lysis. These toxins are produced as inactive precursors, requiring proteolytic maturation and membrane association for activation within host tissues. Here, we investigate Vibrio campbellii αHL (VcαHL), and establish that its hemolytic activity is significantly stimulated by calcium ions, with an EC that aligns with physiological calcium concentrations. Furthermore, we illustrate the vital contribution of calcium ions to the oligomerization of VcαHL on membranes. Using X-ray crystallography and cryo-electron microscopy, we decipher both the immature and assembled structures of VcαHL and elucidate the conformational changes corresponding to toxin assembly. We also identify a calcium-binding module that is integral for VcαHL's calcium-dependent activation. These findings provide insights into the regulatory mechanisms of VcαHL and have the potential to inform the development of targeted therapeutic strategies against Vibrio infections. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36150.map.gz emd_36150.map.gz | 944.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36150-v30.xml emd-36150-v30.xml emd-36150.xml emd-36150.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36150_fsc.xml emd_36150_fsc.xml | 23 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36150.png emd_36150.png | 200.4 KB | ||
| Filedesc metadata |  emd-36150.cif.gz emd-36150.cif.gz | 5.9 KB | ||
| その他 |  emd_36150_additional_1.map.gz emd_36150_additional_1.map.gz emd_36150_half_map_1.map.gz emd_36150_half_map_1.map.gz emd_36150_half_map_2.map.gz emd_36150_half_map_2.map.gz | 499 MB 928.9 MB 928.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36150 http://ftp.pdbj.org/pub/emdb/structures/EMD-36150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36150 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36150_validation.pdf.gz emd_36150_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36150_full_validation.pdf.gz emd_36150_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_36150_validation.xml.gz emd_36150_validation.xml.gz | 30.8 KB | 表示 | |
| CIF形式データ |  emd_36150_validation.cif.gz emd_36150_validation.cif.gz | 40.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36150 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36150 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jc7MC  8jbqC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36150.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36150.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.5305 Å | ||||||||||||||||||||||||||||||||||||
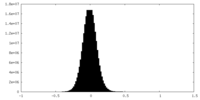
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: unsharpen map
| ファイル | emd_36150_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpen map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
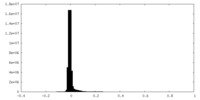
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36150_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
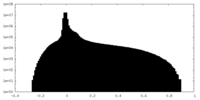
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36150_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
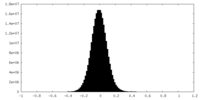
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : heptameric ring-shaped complex of Vibrio campbellii alpha-hemolysin
| 全体 | 名称: heptameric ring-shaped complex of Vibrio campbellii alpha-hemolysin |
|---|---|
| 要素 |
|
-超分子 #1: heptameric ring-shaped complex of Vibrio campbellii alpha-hemolysin
| 超分子 | 名称: heptameric ring-shaped complex of Vibrio campbellii alpha-hemolysin タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Vibrio campbellii (バクテリア) Vibrio campbellii (バクテリア) |
-分子 #1: Hemolysin
| 分子 | 名称: Hemolysin / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vibrio campbellii (バクテリア) Vibrio campbellii (バクテリア) |
| 分子量 | 理論値: 80.643578 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNINEPSGEA ANIISQAADS HAMKYYNAAD WQAEDNALPS LAELRDLVIN QQKSVLFDFS QNSDADGQAE MQAQFRKTYG VGFANQFIF ITEHKGELLF TPFEHSEEVD PKLLEAPLTT RSGLKSTAPT NSETSTLPHV AFYISVNRPI SDEECTFDNS W LWKDEKGS ...文字列: MNINEPSGEA ANIISQAADS HAMKYYNAAD WQAEDNALPS LAELRDLVIN QQKSVLFDFS QNSDADGQAE MQAQFRKTYG VGFANQFIF ITEHKGELLF TPFEHSEEVD PKLLEAPLTT RSGLKSTAPT NSETSTLPHV AFYISVNRPI SDEECTFDNS W LWKDEKGS RPFCKDANIS LIYRVNLERS LQYGIVGSAT PNAKIVRISL DDDSSGAGIH LNDQLSYRRF GASYTTLDAY FR EWSTDAI AQDYRFVFKT SNNKAEILET FPIDNLNVKY EKRKQSGFEL GVTGGAEVSE DGPKAKLEAR ASITQSRWLT YNT QDYRVE RNAKNAQTVS FTWNRQEYAT AESLLNRSTD ALWVDTYPVD VNRISPLSYA SFVPKMDVIY KASDTETGST DFII DSSVN IRPIYNGAYK HYYVVGAHQS YHGFENSPRR RITKSASFTV DWDHPVFTGG RPVNLQLASF NNRCVQVDAQ SRLTA NTCD DQQSAQSFIY DQLGRYVSAS NTELCLDGAA LDVLQTCNQN LTQRWEWRKN TDELTNVYSG ESLGHDKQTG ELGLYA SSN DAVSLRTITA YTNVFNVQKS SPILGYTQGK MNQQSVGQNY RLYVREGSAI DALGTASDLL VGGNGGSLTS VDLSGVK SI TATSGDFQYG GQQLVALTFT YQDGRQQMVG SKAHVTNAHE DRFDLPDAAK ITQLNIWADD WLVKGVQFDL NLEHHHHH H UniProtKB: UNIPROTKB: A0A344KRS4 |
-分子 #2: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 2 / コピー数: 7 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 7 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 591 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)