+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of SARS CoV-2 RBD and Aptamer complex | |||||||||
 マップデータ マップデータ | Protein:RBD_Fab_AM032_AM047, EM: Titan krios 300keV, Dose:50, Defocus:-0.8 to -2.0, Magnification: 105k, movies: 11743 plus 9597, Map:from 3DFlex reconstruction | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Aptamer / SARS CoV-2 RBD / Inhibitor / NapdU / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / entry receptor-mediated virion attachment to host cell / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / membrane / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
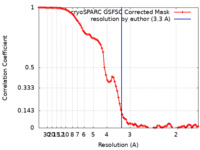
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Rahman MS / Jang SK / Lee JO | |||||||||
| 資金援助 |  韓国, 1件 韓国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Molecules / 年: 2023 ジャーナル: Molecules / 年: 2023タイトル: Structure-Guided Development of Bivalent Aptamers Blocking SARS-CoV-2 Infection. 著者: Md Shafiqur Rahman / Min Jung Han / Sang Won Kim / Seong Mu Kang / Bo Ri Kim / Heesun Kim / Chang Jun Lee / Jung Eun Noh / Hanseong Kim / Jie-Oh Lee / Sung Key Jang /  要旨: Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) has caused devastation to human society through its high virulence, infectivity, and genomic mutations, which reduced the efficacy of ...Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) has caused devastation to human society through its high virulence, infectivity, and genomic mutations, which reduced the efficacy of vaccines. Here, we report the development of aptamers that effectively interfere with SARS-CoV-2 infection by targeting its spike protein, which plays a pivotal role in host cell entry of the virus through interaction with the viral receptor angiotensin-converting enzyme 2 (ACE2). To develop highly effective aptamers and to understand their mechanism in inhibiting viral infection, we determined the three-dimensional (3D) structures of aptamer/receptor-binding domain (RBD) complexes using cryogenic electron microscopy (cryo-EM). Moreover, we developed bivalent aptamers targeting two distinct regions of the RBD in the spike protein that directly interact with ACE2. One aptamer interferes with the binding of ACE2 by blocking the ACE2-binding site in RBD, and the other aptamer allosterically inhibits ACE2 by binding to a distinct face of RBD. Using the 3D structures of aptamer-RBD complexes, we minimized and optimized these aptamers. By combining the optimized aptamers, we developed a bivalent aptamer that showed a stronger inhibitory effect on virus infection than the component aptamers. This study confirms that the structure-based aptamer-design approach has a high potential in developing antiviral drugs against SARS-CoV-2 and other viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35930.map.gz emd_35930.map.gz | 58.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35930-v30.xml emd-35930-v30.xml emd-35930.xml emd-35930.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35930_fsc.xml emd_35930_fsc.xml | 9.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35930.png emd_35930.png | 36.6 KB | ||
| その他 |  emd_35930_half_map_1.map.gz emd_35930_half_map_1.map.gz emd_35930_half_map_2.map.gz emd_35930_half_map_2.map.gz | 3.4 MB 3.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35930 http://ftp.pdbj.org/pub/emdb/structures/EMD-35930 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35930 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35930 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35930_validation.pdf.gz emd_35930_validation.pdf.gz | 537.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35930_full_validation.pdf.gz emd_35930_full_validation.pdf.gz | 537.3 KB | 表示 | |
| XML形式データ |  emd_35930_validation.xml.gz emd_35930_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_35930_validation.cif.gz emd_35930_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35930 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35930 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35930 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35930 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8j1qMC  8j26C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35930.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35930.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Protein:RBD_Fab_AM032_AM047, EM: Titan krios 300keV, Dose:50, Defocus:-0.8 to -2.0, Magnification: 105k, movies: 11743 plus 9597, Map:from 3DFlex reconstruction | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A
| ファイル | emd_35930_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_35930_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RBD_Fab_AM032_AM047 complex
| 全体 | 名称: RBD_Fab_AM032_AM047 complex |
|---|---|
| 要素 |
|
-超分子 #1: RBD_Fab_AM032_AM047 complex
| 超分子 | 名称: RBD_Fab_AM032_AM047 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  株: Wuhan |
| 分子量 | 理論値: 124 KDa |
-分子 #1: AM032-0
| 分子 | 名称: AM032-0 / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 26.51775 KDa |
| 配列 | 文字列: (DT)(DA)(DT)(DC)(DG)(DA)(DG)(DC)(DG)(DT) (DC)(DC)(DT)(DG)(DC)(DC)(DT)(DT)(DT)(DG) (DC)(DG)(DG)(DA)(DG)(DG)(85Y)(DG)(DA) (DA)(DC)(DC)(85Y)(85Y)(DA)(DC)(DG)(85Y) (85Y)(DC)(DA)(DA)(DC)(85Y) ...文字列: (DT)(DA)(DT)(DC)(DG)(DA)(DG)(DC)(DG)(DT) (DC)(DC)(DT)(DG)(DC)(DC)(DT)(DT)(DT)(DG) (DC)(DG)(DG)(DA)(DG)(DG)(85Y)(DG)(DA) (DA)(DC)(DC)(85Y)(85Y)(DA)(DC)(DG)(85Y) (85Y)(DC)(DA)(DA)(DC)(85Y)(DG)(DG)(DA)(85Y) (DC)(DG)(85Y)(DG)(85Y)(DG)(85Y)(DA) (DG) (DG)(85Y)(DA)(DC)(DA)(DC)(DC)(DG)(DA)(DC) (DA)(DG)(DC)(DC)(DA)(DC)(DC)(DC) (DA) (DG)(DA)(DA)(DA) |
-分子 #2: AM047-0
| 分子 | 名称: AM047-0 / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 18.034312 KDa |
| 配列 | 文字列: (DC)(DT)(DG)(85Y)(DC)(DG)(DG)(85Y)(DA)(DA) (DG)(DG)(DG)(DA)(DG)(DC)(85Y)(DA)(DG) (85Y)(DG)(85Y)(DA)(85Y)(DG)(DC)(DC)(DC)(DC) (85Y)(DG)(DA)(DG)(85Y)(85Y)(DG)(DG) (DG) (DG)(DA)(85Y)(DA)(85Y) ...文字列: (DC)(DT)(DG)(85Y)(DC)(DG)(DG)(85Y)(DA)(DA) (DG)(DG)(DG)(DA)(DG)(DC)(85Y)(DA)(DG) (85Y)(DG)(85Y)(DA)(85Y)(DG)(DC)(DC)(DC)(DC) (85Y)(DG)(DA)(DG)(85Y)(85Y)(DG)(DG) (DG) (DG)(DA)(85Y)(DA)(85Y)(DC)(DG)(DC)(DC)(DG) (DA)(DC)(DA)(DG) |
-分子 #3: Fab Light chain (REGN10987)
| 分子 | 名称: Fab Light chain (REGN10987) / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.90933 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: QSALTQPASV SGSPGQSITI SCTGTSSDVG GYNYVSWYQQ HPGKAPKLMI YDVSKRPSGV SNRFSGSKSG NTASLTISGL QSEDEADYY CNSLTSISTW VFGGGTKLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP ...文字列: QSALTQPASV SGSPGQSITI SCTGTSSDVG GYNYVSWYQQ HPGKAPKLMI YDVSKRPSGV SNRFSGSKSG NTASLTISGL QSEDEADYY CNSLTSISTW VFGGGTKLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP SKQSNNKYAA SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECSHH HHHHHH |
-分子 #4: Fab heavy chain (REGN10987)
| 分子 | 名称: Fab heavy chain (REGN10987) / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.705879 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: QVQLVESGGG VVQPGRSLRL SCAASGFTFS NYAMYWVRQA PGKGLEWVAV ISYDGSNKYY ADSVKGRFTI SRDNSKNTLY LQMNSLRTE DTAVYYCASG SDYGDYLLVY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS ...文字列: QVQLVESGGG VVQPGRSLRL SCAASGFTFS NYAMYWVRQA PGKGLEWVAV ISYDGSNKYY ADSVKGRFTI SRDNSKNTLY LQMNSLRTE DTAVYYCASG SDYGDYLLVY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV EPKSCDKSNS LVPRGSPSRL EE ELRRRLT E |
-分子 #5: Spike protein S1
| 分子 | 名称: Spike protein S1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: Wuhan |
| 分子量 | 理論値: 28.415693 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RVQPTESIVR FPNITNLCPF GEVFNATRFA SVYAWNRKRI SNCVADYSVL YNSASFSTFK CYGVSPTKLN DLCFTNVYAD SFVIRGDEV RQIAPGQTGK IADYNYKLPD DFTGCVIAWN SNNLDSKVGG NYNYLYRLFR KSNLKPFERD ISTEIYQAGS T PCNGVEGF ...文字列: RVQPTESIVR FPNITNLCPF GEVFNATRFA SVYAWNRKRI SNCVADYSVL YNSASFSTFK CYGVSPTKLN DLCFTNVYAD SFVIRGDEV RQIAPGQTGK IADYNYKLPD DFTGCVIAWN SNNLDSKVGG NYNYLYRLFR KSNLKPFERD ISTEIYQAGS T PCNGVEGF NCYFPLQSYG FQPTNGVGYQ PYRVVVLSFE LLHAPATVCG PKKSTNLVKN KCVNFENLYF QGAAAGGSHH HH HHGGSDY KDDDDK |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 1 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.74 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 1mM MgCl2, 0.15% amphipol A8-35 and 0.003% cymal-6 additive added during sample preparation | |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.034 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: 2.5 ul of samples placed on grid before plunge frozen with 5s blot time. | |||||||||
| 詳細 | Aptamer AM032 and AM047 mixed with RBD_Fab complex at 1:1.2 molar ratio and incubate 1hr on ice before preparing grids |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 21340 / 平均露光時間: 4.56 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 105000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.73 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | (Chain: PDB, experimental model, PDB, experimental model, PDB, experimental model) |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8j1q: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X