[日本語] English
 万見
万見- EMDB-35827: Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cas9 complex / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA-RNA complex | |||||||||
| 生物種 |  Chryseobacterium (バクテリア) Chryseobacterium (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / 解像度: 2.46 Å | |||||||||
 データ登録者 データ登録者 | Zhang S / Lin S / Liu JJG | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Pro-CRISPR PcrIIC1-associated Cas9 system for enhanced bacterial immunity. 著者: Shouyue Zhang / Ao Sun / Jing-Mei Qian / Shuo Lin / Wenjing Xing / Yun Yang / Han-Zhou Zhu / Xin-Yi Zhou / Yan-Shuo Guo / Yun Liu / Yu Meng / Shu-Lin Jin / Wenhao Song / Cheng-Ping Li / ...著者: Shouyue Zhang / Ao Sun / Jing-Mei Qian / Shuo Lin / Wenjing Xing / Yun Yang / Han-Zhou Zhu / Xin-Yi Zhou / Yan-Shuo Guo / Yun Liu / Yu Meng / Shu-Lin Jin / Wenhao Song / Cheng-Ping Li / Zhaofu Li / Shuai Jin / Jian-Hua Wang / Meng-Qiu Dong / Caixia Gao / Chunlai Chen / Yang Bai / Jun-Jie Gogo Liu /  要旨: The CRISPR system is an adaptive immune system found in prokaryotes that defends host cells against the invasion of foreign DNA. As part of the ongoing struggle between phages and the bacterial ...The CRISPR system is an adaptive immune system found in prokaryotes that defends host cells against the invasion of foreign DNA. As part of the ongoing struggle between phages and the bacterial immune system, the CRISPR system has evolved into various types, each with distinct functionalities. Type II Cas9 is the most extensively studied of these systems and has diverse subtypes. It remains uncertain whether members of this family can evolve additional mechanisms to counter viral invasions. Here we identify 2,062 complete Cas9 loci, predict the structures of their associated proteins and reveal three structural growth trajectories for type II-C Cas9. We found that novel associated genes (NAGs) tended to be present within the loci of larger II-C Cas9s. Further investigation revealed that CbCas9 from Chryseobacterium species contains a novel β-REC2 domain, and forms a heterotetrameric complex with an NAG-encoded CRISPR-Cas-system-promoting (pro-CRISPR) protein of II-C Cas9 (PcrIIC1). The CbCas9-PcrIIC1 complex exhibits enhanced DNA binding and cleavage activity, broader compatibility for protospacer adjacent motif sequences, increased tolerance for mismatches and improved anti-phage immunity, compared with stand-alone CbCas9. Overall, our work sheds light on the diversity and 'growth evolutionary' trajectories of II-C Cas9 proteins at the structural level, and identifies many NAGs-such as PcrIIC1, which serves as a pro-CRISPR factor to enhance CRISPR-mediated immunity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35827.map.gz emd_35827.map.gz | 91.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35827-v30.xml emd-35827-v30.xml emd-35827.xml emd-35827.xml | 22.2 KB 22.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35827.png emd_35827.png | 24.3 KB | ||
| Filedesc metadata |  emd-35827.cif.gz emd-35827.cif.gz | 7.5 KB | ||
| その他 |  emd_35827_half_map_1.map.gz emd_35827_half_map_1.map.gz emd_35827_half_map_2.map.gz emd_35827_half_map_2.map.gz | 95.6 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35827 http://ftp.pdbj.org/pub/emdb/structures/EMD-35827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35827 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35827_validation.pdf.gz emd_35827_validation.pdf.gz | 605.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35827_full_validation.pdf.gz emd_35827_full_validation.pdf.gz | 605.4 KB | 表示 | |
| XML形式データ |  emd_35827_validation.xml.gz emd_35827_validation.xml.gz | 12.9 KB | 表示 | |
| CIF形式データ |  emd_35827_validation.cif.gz emd_35827_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35827 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35827 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35827.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35827.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0825 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35827_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
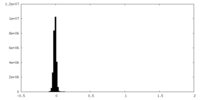
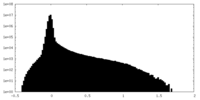
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35827_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
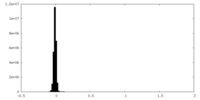
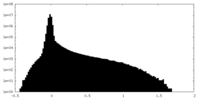
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate
| 全体 | 名称: Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate |
|---|---|
| 要素 |
|
-超分子 #1: Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate
| 超分子 | 名称: Structure of CbCas9 bound to 20-nucleotide complementary DNA substrate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Chryseobacterium (バクテリア) Chryseobacterium (バクテリア) |
| 分子量 | 理論値: 228.04 KDa |
-分子 #1: deadCbCas9
| 分子 | 名称: deadCbCas9 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chryseobacterium (バクテリア) Chryseobacterium (バクテリア) |
| 分子量 | 理論値: 170.029109 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIKNILGLAL GTNSIGWALV KQDFENKQGE ILGMGSRIIP MSQDILGDFG KGNSVSQTAE RTKYRSVRRL RERFLLRRER LHRVLYILN FLPEHYASQI DFEKRLGKFK VETEPKLVWK NTDGQFSFLF QNSFNEMLED FKAAGQELKI PYDWTIYHLR K KAISQKIE ...文字列: MIKNILGLAL GTNSIGWALV KQDFENKQGE ILGMGSRIIP MSQDILGDFG KGNSVSQTAE RTKYRSVRRL RERFLLRRER LHRVLYILN FLPEHYASQI DFEKRLGKFK VETEPKLVWK NTDGQFSFLF QNSFNEMLED FKAAGQELKI PYDWTIYHLR K KAISQKIE KEELAWILLN FNHKRGYYQL RGEDFEEEKD KTFVRLKVDR IVDSGENVKG KILYDVYFEN GWKYDKQVVK TE DWVDRTK EFIVSESILK NGETKRTFKA VDSEKDWIAI KTKTEQEIEH SHKTVGTYIY ETLLQNPKQK IKGKLVRTIE RKF YKEELR QILEKQKEFH QELQSDDLYN DCIRELYRNN EVHQLTLRKK DFVHLFMEDI IFYQRPLRSQ KSSVSNCTLE FRKY KGENG AEHTQYLKAI PKSNPYYQEF RLWQWIFNLN LYTKDNDENV TKVFLNTTQD FENLFEFLNT RKEVDQKALL KHFKL NEKT HRWNFVEDKK YPCNETKTMI SSRLDKVENI SDDFLTRDIE QKIWHIIYSV NDKVEYEKAL KSFARKHHLD ESSFFE AFR KFPPFKSEYG SFSEKAIKKL LPLMRLGKYW NYAEIDKYSR ERIQKIITGE YDENIKDKVR EKSVHLTIEN DFQGLQL WL AQYIVYGRHS EASMIGKWNS ANDLEVFLKD FKQHSLRNPI VEQVITETLR VVKDIWLKYG NGTKDFFNEI HIELGREM K LPADDRKKLT NQITENENTN LRIKALLAEM MNDHSVENVR PFSPMQQEIL KIYEDGVLKS DIEIEDDILK ISKTAQPSS SDLKRYKLWL EQKYKSPYTG QIIPLNKLFT PEYEIEAIIP QSRYFDDSFS NKIICESAVN KLKDNYIGLG FIKQFGGTII ELGFGKSVK VFDTEEYEDF VKKHYANNRG KRNKLLMEDI PEKMIERQLN DTRYISKYIS GILSNIVRVE DGSDEGVNSK N IVPGNGKI TTQLKQDWGL NDVWNDLILP RFERMNQLTN SKDFTAWNEN HQKFLPTVPI EFSKGFSKKR IDHRHHALDA LV IACATTD HVNLLNNQSA KSDTKRYDLK KKLMKFEKVV YHHTQTGEKI EREIPKQFLK PWEKFTVDAK HNLESIIVSF KQN LRVINK ATNYYEKYVE KDGTKNKERV EQAGTNWAIR KPMHKDTVSG KVDLPWVKVP KGKILTATRK SLDSSFDLKS IGSI TDTGI QKILKNYLAF KDGNPELAFS PEGIDDLNKN IEKYNDGKPH QPINKVRVFE LGSKFQVGQT GNKKGKYVEA AKGTN LFFA VYEDEKGKRS YETIPLNEVI ERQKQGLTSV PLENEKGSRL LFDLSPNDLV YVPEIDENID SNFVFSNLNK EKISRI YKV EKTSGTECYF VRQDIAYLIK QYDAKTKIGE LESQNKLQVT MTDDRIRITD TCVKINCDRL GNINFITKEK IKQIFNE FR |
-分子 #2: TS
| 分子 | 名称: TS / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Chryseobacterium (バクテリア) Chryseobacterium (バクテリア) |
| 分子量 | 理論値: 8.450413 KDa |
| 配列 | 文字列: (DC)(DG)(DT)(DT)(DT)(DT)(DG)(DT)(DC)(DT) (DC)(DG)(DG)(DC)(DT)(DC)(DC)(DC)(DC)(DG) (DA)(DC)(DA)(DT)(DT)(DC)(DT)(DC) |
-分子 #4: NTS
| 分子 | 名称: NTS / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Chryseobacterium (バクテリア) Chryseobacterium (バクテリア) |
| 分子量 | 理論値: 8.76267 KDa |
| 配列 | 文字列: (DG)(DA)(DG)(DA)(DA)(DT)(DG)(DT)(DC)(DG) (DG)(DG)(DG)(DA)(DG)(DC)(DC)(DG)(DA)(DG) (DA)(DC)(DA)(DA)(DA)(DA)(DC)(DG) |
-分子 #3: sgRNA
| 分子 | 名称: sgRNA / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Chryseobacterium (バクテリア) Chryseobacterium (バクテリア) |
| 分子量 | 理論値: 40.936227 KDa |
| 配列 | 文字列: GAGAAUGUCG GGGAGCCGAG GUUGUGAAUU GCUUUCAAAA AUUAUUGAGA AAUAAUUUUG AAAAGCAAUU CACAAUAAGG AUUAUUCCG UUGUGAAAAC AUUCAAGGCG GGGCAACUCG CCUUUUUU |
-実験情報
-構造解析
 解析 解析 | 単粒子再構成法 |
|---|---|
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 1.0 uM / 構成要素 - 式: NaCl / 構成要素 - 名称: sodium chloride 詳細: 150mM NaCl, 20mM Hepes (pH=7.5), 5mM MgCl2, 1mM Tcep, 0.1% Glycerol |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 温度因子: 88 |
|---|---|
| 得られたモデル |  PDB-8iyq: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)