+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Crt-SPARTA-gRNA-tDNA dimer (conformation-2) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ago / DNA/RNA / Dimer / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Thermoflavifilum thermophilum (バクテリア) / synthetic construct (人工物) Thermoflavifilum thermophilum (バクテリア) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Gao X / Shang K / Zhu K / Wang L / Mu Z / Fu X / Yu X / Qin B / Zhu H / Ding W / Cui S | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Nucleic-acid-triggered NADase activation of a short prokaryotic Argonaute. 著者: Xiaopan Gao / Kun Shang / Kaixiang Zhu / Linyue Wang / Zhixia Mu / Xingke Fu / Xia Yu / Bo Qin / Hongtao Zhu / Wei Ding / Sheng Cui /  要旨: Argonaute (Ago) proteins mediate RNA- or DNA-guided inhibition of nucleic acids. Although the mechanisms used by eukaryotic Ago proteins and long prokaryotic Ago proteins (pAgos) are known, that used ...Argonaute (Ago) proteins mediate RNA- or DNA-guided inhibition of nucleic acids. Although the mechanisms used by eukaryotic Ago proteins and long prokaryotic Ago proteins (pAgos) are known, that used by short pAgos remains elusive. Here we determined the cryo-electron microscopy structures of a short pAgo and the associated TIR-APAZ proteins (SPARTA) from Crenotalea thermophila (Crt): a free-state Crt-SPARTA; a guide RNA-target DNA-loaded Crt-SPARTA; two Crt-SPARTA dimers with distinct TIR organization; and a Crt-SPARTA tetramer. These structures reveal that Crt-SPARTA is composed of a bilobal-fold Ago lobe that connects with a TIR lobe. Whereas the Crt-Ago contains a MID and a PIWI domain, Crt-TIR-APAZ has a TIR domain, an N-like domain, a linker domain and a trigger domain. The bound RNA-DNA duplex adopts a B-form conformation that is recognized by base-specific contacts. Nucleic acid binding causes conformational changes because the trigger domain acts as a 'roadblock' that prevents the guide RNA 5' ends and the target DNA 3' ends from reaching their canonical pockets; this disorders the MID domain and promotes Crt-SPARTA dimerization. Two RNA-DNA-loaded Crt-SPARTA dimers form a tetramer through their TIR domains. Four Crt-TIR domains assemble into two parallel head-to-tail-organized TIR dimers, indicating an NADase-active conformation, which is supported by our mutagenesis study. Our results reveal the structural basis of short-pAgo-mediated defence against invading nucleic acids, and provide insights for optimizing the detection of SPARTA-based programmable DNA sequences. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35702.map.gz emd_35702.map.gz | 31.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35702-v30.xml emd-35702-v30.xml emd-35702.xml emd-35702.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35702.png emd_35702.png | 142.9 KB | ||
| Filedesc metadata |  emd-35702.cif.gz emd-35702.cif.gz | 6.7 KB | ||
| その他 |  emd_35702_additional_1.map.gz emd_35702_additional_1.map.gz emd_35702_half_map_1.map.gz emd_35702_half_map_1.map.gz emd_35702_half_map_2.map.gz emd_35702_half_map_2.map.gz | 56.9 MB 59.2 MB 59.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35702 http://ftp.pdbj.org/pub/emdb/structures/EMD-35702 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35702 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35702 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35702_validation.pdf.gz emd_35702_validation.pdf.gz | 693.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35702_full_validation.pdf.gz emd_35702_full_validation.pdf.gz | 693.1 KB | 表示 | |
| XML形式データ |  emd_35702_validation.xml.gz emd_35702_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_35702_validation.cif.gz emd_35702_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35702 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35702 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35702 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35702 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35702.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35702.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
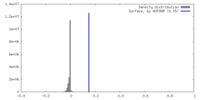
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_35702_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
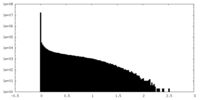
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35702_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
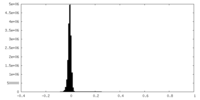
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35702_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The dimer of prokaryotic Argonaute System with DNA and RNA
| 全体 | 名称: The dimer of prokaryotic Argonaute System with DNA and RNA |
|---|---|
| 要素 |
|
-超分子 #1: The dimer of prokaryotic Argonaute System with DNA and RNA
| 超分子 | 名称: The dimer of prokaryotic Argonaute System with DNA and RNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Thermoflavifilum thermophilum (バクテリア) Thermoflavifilum thermophilum (バクテリア) |
-分子 #1: Piwi domain-containing protein
| 分子 | 名称: Piwi domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermoflavifilum thermophilum (バクテリア) Thermoflavifilum thermophilum (バクテリア) |
| 分子量 | 理論値: 58.304848 KDa |
| 組換発現 | 生物種: Expression vector pET-mod (その他) |
| 配列 | 文字列: MKELIYIEEP SILFAHGQKC TDPRDGLALF GPLNQIYGIK SGVVGTQKGL QIFKSYLDKI QKPIYNHNNI TRPMFPGFEA VFGCKWESQ NIVFKEITDE EIRRYLFNAS THKRTYDLVT LFNDKIITAN KNDEERVDVW FVIVPEEIYK YCRPNSVLPN E LVQTKSLI ...文字列: MKELIYIEEP SILFAHGQKC TDPRDGLALF GPLNQIYGIK SGVVGTQKGL QIFKSYLDKI QKPIYNHNNI TRPMFPGFEA VFGCKWESQ NIVFKEITDE EIRRYLFNAS THKRTYDLVT LFNDKIITAN KNDEERVDVW FVIVPEEIYK YCRPNSVLPN E LVQTKSLI SKSKAKSFRY TPTLFEEFNK KLKEVEKEAK TYNYDAQFHD QLKARLLEHT IPTQILREST LAWRDFKNTF GA PIRDFSK IEGHLAWTIS TAAYYKAGGK PWKLGDIRPG VCYLGLVYKK IEKSKNPQNA CCAAQMFLDN GDGTVFKGEV GPW YNPEKG EYHLKPKEAK ALLTQALESY KEQNKSYPKE VFIHARTRFN DEEWNAFNEV TPKNTNLVGV TITKSKPLKL YKTE GAFPI MRGNAYIVDE KKAFLWTLGF VPKLQSTLSM EVPNPIFIEI NKGEAEIQQV LKDILALTKL NYNACIYADG EPVTL RFAN KIGEILTAST EIKTPPLAFK YYI UniProtKB: Piwi domain-containing protein |
-分子 #2: TIR domain-containing protein
| 分子 | 名称: TIR domain-containing protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermoflavifilum thermophilum (バクテリア) Thermoflavifilum thermophilum (バクテリア) |
| 分子量 | 理論値: 53.256734 KDa |
| 組換発現 | 生物種: Expression vector pET-mod (その他) |
| 配列 | 文字列: MRNKIFISHA TPEDDDFTRW LSLKLIGLGY EVWCDILFLD KGVDFWSTIE KEIRENTCKF LIVSSTAGNK REGVLKELAV ATKVKKHLQ DDMFIIPLAI DENLSYDDIN IEIVRLNAID FKKSWAKGLQ DLLDAFEKQN VPKKPPDHSK SNLLYQQIFL H DKQAIEKE ...文字列: MRNKIFISHA TPEDDDFTRW LSLKLIGLGY EVWCDILFLD KGVDFWSTIE KEIRENTCKF LIVSSTAGNK REGVLKELAV ATKVKKHLQ DDMFIIPLAI DENLSYDDIN IEIVRLNAID FKKSWAKGLQ DLLDAFEKQN VPKKPPDHSK SNLLYQQIFL H DKQAIEKE ETYDSNWFPI ISFPNELRFH RYDWRLPKQF DVRTLAFPAI RYKEYLCTFA WEYDFIHQLP KTETYNGQES IR ISTSDIL SGRYDTDFIR NYECQRLIVQ LINKAFELRM KDKNVREYQM SKTFAYWIEK GKLEKDKFEK IKLVGKQKNK YWH FGISAA GKLYPSPVLM VSSHIIFTMD GINLIKSKSI QHSSRRKQGK NWWNDKWREK LLAFIRFLSD DQNAIYLNVG SEEK ILISN KPLKFFGKMS YVTPSEVTLE EESVLADINN FEEDTEDLDE LEDIE UniProtKB: TIR domain-containing protein |
-分子 #3: DNA (45-mer)
| 分子 | 名称: DNA (45-mer) / タイプ: dna / ID: 3 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.721869 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DC)(DG)(DA)(DC)(DG)(DG)(DC) (DC)(DA)(DG)(DT)(DG)(DC)(DC)(DA)(DA)(DG) (DC)(DA)(DA)(DA)(DC)(DT)(DA)(DT)(DA) (DC)(DA)(DA)(DC)(DC)(DT)(DA)(DC)(DT)(DA) (DC) (DC)(DT)(DC)(DA)(DT) |
-分子 #4: RNA (5'-R(P*UP*GP*AP*GP*GP*UP*AP*GP*UP*AP*GP*GP*UP*UP*GP*UP*AP*UP...
| 分子 | 名称: RNA (5'-R(P*UP*GP*AP*GP*GP*UP*AP*GP*UP*AP*GP*GP*UP*UP*GP*UP*AP*UP*AP*GP*U)-3') タイプ: rna / ID: 4 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 6.812045 KDa |
| 配列 | 文字列: UGAGGUAGUA GGUUGUAUAG U |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.8 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 4.2) / 使用した粒子像数: 686832 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-8it0: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)