+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3558 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Centriolar Proximal MT triplet in centrosomes extracted from o.aries thymocytes | |||||||||
 マップデータ マップデータ | Centriolar proximal MT triplet from a mammal centrosome. Centrosomes were extracted from o.aries thymocytes. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 47.0 Å | |||||||||
 データ登録者 データ登録者 | Busselez J / Chichon FJ / Melero R / Carrascosa JL / Carazo JM | |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2019 ジャーナル: Sci Rep / 年: 2019タイトル: Cryo-Electron Tomography and Proteomics studies of centrosomes from differentiated quiescent thymocytes. 著者: Johan Busselez / Francisco Javier Chichón / Maria Josefa Rodríguez / Adan Alpízar / Séverine Isabelle Gharbi / Mònica Franch / Roberto Melero / Alberto Paradela / José L Carrascosa / José-Maria Carazo /   要旨: We have used cryo Electron Tomography, proteomics and immunolabeling to study centrosomes isolated from the young lamb thymus, an efficient source of quiescent differentiated cells. We compared the ...We have used cryo Electron Tomography, proteomics and immunolabeling to study centrosomes isolated from the young lamb thymus, an efficient source of quiescent differentiated cells. We compared the proteome of thymocyte centrosomes to data published for KE37 cells, focusing on proteins associated with centriole disengagement and centrosome separation. The data obtained enhances our understanding of the protein system joining the centrioles, a system comprised of a branched network of fibers linked to an apparently amorphous density that was partially characterized here. A number of proteins were localized to the amorphous density by immunolabeling (C-NAP1, cohesin SMC1, condensin SMC4 and NCAPD2), yet not DNA. In conjuction, these data not only extend our understanding of centrosomes but they will help refine the model that focus on the protein system associated with the centriolar junction. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3558.map.gz emd_3558.map.gz | 9.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3558-v30.xml emd-3558-v30.xml emd-3558.xml emd-3558.xml | 14.3 KB 14.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
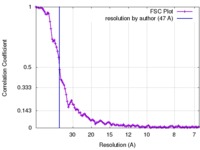
| FSC (解像度算出) |  emd_3558_fsc.xml emd_3558_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3558.png emd_3558.png | 40.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3558 http://ftp.pdbj.org/pub/emdb/structures/EMD-3558 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3558 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3558 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3558_validation.pdf.gz emd_3558_validation.pdf.gz | 226.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3558_full_validation.pdf.gz emd_3558_full_validation.pdf.gz | 225.2 KB | 表示 | |
| XML形式データ |  emd_3558_validation.xml.gz emd_3558_validation.xml.gz | 12.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3558 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3558 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3558 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3558 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3558.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3558.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Centriolar proximal MT triplet from a mammal centrosome. Centrosomes were extracted from o.aries thymocytes. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.42 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Centrosome enriched from young lamb thymocytes
| 全体 | 名称: Centrosome enriched from young lamb thymocytes |
|---|---|
| 要素 |
|
-超分子 #1: Centrosome enriched from young lamb thymocytes
| 超分子 | 名称: Centrosome enriched from young lamb thymocytes / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 詳細: The centrosome were extracted accordingly to Komesli et al. 1989 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 230000 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.076 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 / 構成要素 - 濃度: 10.0 mM / 構成要素 - 式: C8H18N2O6S2 / 構成要素 - 名称: Pipes / 詳細: pH rectification with KOH |
| グリッド | モデル: quantifoil 3.5/1 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: FORMVAR / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC |
| 詳細 | The sample was enriched by two step of sedimentation in sucrose gradient. The sucrose was washed out in several drops of buffer, the last of which included fiducial markers (10 nm bovine serum albumin (BSA)-coated gold beads) for tilt series alignment |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: Gatan 968 Quantum エネルギーフィルター - エネルギー下限: -10 eV エネルギーフィルター - エネルギー上限: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 平均露光時間: 1.0 sec. / 平均電子線量: 1.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 5.906 µm / 最小 デフォーカス(補正後): 5.54 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)