+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi (Focused map on PYY(3-36)-Y2R) | |||||||||
 マップデータ マップデータ | Focused map (PYY(3-36)-Y2R) | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
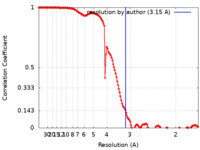
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.15 Å | |||||||||
 データ登録者 データ登録者 | Kang H / Park C / Kim J / Choi H-J | |||||||||
| 資金援助 |  韓国, 2件 韓国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Structural basis for Y2 receptor-mediated neuropeptide Y and peptide YY signaling. 著者: Hyunook Kang / Chaehee Park / Yeol Kyo Choi / Jungnam Bae / Sohee Kwon / Jinuk Kim / Chulwon Choi / Chaok Seok / Wonpil Im / Hee-Jung Choi /   要旨: Neuropeptide Y (NPY) and its receptors are expressed in various human tissues including the brain where they regulate appetite and emotion. Upon NPY stimulation, the neuropeptide Y1 and Y2 receptors ...Neuropeptide Y (NPY) and its receptors are expressed in various human tissues including the brain where they regulate appetite and emotion. Upon NPY stimulation, the neuropeptide Y1 and Y2 receptors (YR and YR, respectively) activate G signaling, but their physiological responses to food intake are different. In addition, deletion of the two N-terminal amino acids of peptide YY (PYY(3-36)), the endogenous form found in circulation, can stimulate YR but not YR, suggesting that YR and YR may have distinct ligand-binding modes. Here, we report the cryo-electron microscopy structures of the PYY(3-36)‒YR‒G and NPY‒YR‒G complexes. Using cell-based assays, molecular dynamics simulations, and structural analysis, we revealed the molecular basis of the exclusive binding of PYY(3-36) to YR. Furthermore, we demonstrated that YR favors G protein signaling over β-arrestin signaling upon activation, whereas YR does not show a preference between these two pathways. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35498.map.gz emd_35498.map.gz | 230.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35498-v30.xml emd-35498-v30.xml emd-35498.xml emd-35498.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35498_fsc.xml emd_35498_fsc.xml | 13.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35498.png emd_35498.png | 38.2 KB | ||
| その他 |  emd_35498_half_map_1.map.gz emd_35498_half_map_1.map.gz emd_35498_half_map_2.map.gz emd_35498_half_map_2.map.gz | 226.9 MB 226.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35498 http://ftp.pdbj.org/pub/emdb/structures/EMD-35498 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35498 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35498 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35498_validation.pdf.gz emd_35498_validation.pdf.gz | 817.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35498_full_validation.pdf.gz emd_35498_full_validation.pdf.gz | 817.5 KB | 表示 | |
| XML形式データ |  emd_35498_validation.xml.gz emd_35498_validation.xml.gz | 21.6 KB | 表示 | |
| CIF形式データ |  emd_35498_validation.cif.gz emd_35498_validation.cif.gz | 27.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35498 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35498 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35498 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35498 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35498.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35498.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused map (PYY(3-36)-Y2R) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.849 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map B
| ファイル | emd_35498_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_35498_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex structure of PYY(3-36)-Y2R-Gi-scFv16
| 全体 | 名称: Complex structure of PYY(3-36)-Y2R-Gi-scFv16 |
|---|---|
| 要素 |
|
-超分子 #1: Complex structure of PYY(3-36)-Y2R-Gi-scFv16
| 超分子 | 名称: Complex structure of PYY(3-36)-Y2R-Gi-scFv16 / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: Guanine nucleotide-binding protein
| 超分子 | 名称: Guanine nucleotide-binding protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3, #5 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: scFv16
| 超分子 | 名称: scFv16 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #6 |
|---|
-超分子 #4: PYY(3-36)
| 超分子 | 名称: PYY(3-36) / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #4 |
|---|
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.75 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)