+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | C2 reconstruction of the concave tetramer in the cube-like assembly of 201Phi2-1 gp105 | |||||||||
 マップデータ マップデータ | The cryo-EM map of the phage-encoded nucleus-like shell protein gp105 concave tetramer reconstructed with C2 symmetry imposed. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | jumbo phage 201phi2-1 / gp105 / nucleus-like structure. / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 | host cell cytoplasm / Chimallin 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas phage 201phi2-1 (ファージ) Pseudomonas phage 201phi2-1 (ファージ) | |||||||||
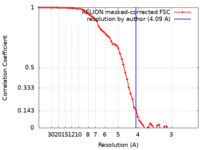
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.09 Å | |||||||||
 データ登録者 データ登録者 | Liu Z / Xiang Y | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Front Microbiol / 年: 2023 ジャーナル: Front Microbiol / 年: 2023タイトル: Structural studies of the nucleus-like assembly of jumbo bacteriophage 201φ2-1. 著者: Zhe Liu / Ye Xiang /  要旨: The jumbo phages encode proteins that assemble to form a nucleus-like compartment in infected cells. Here we report the cryo-EM structure and biochemistry characterization of gp105, a protein that is ...The jumbo phages encode proteins that assemble to form a nucleus-like compartment in infected cells. Here we report the cryo-EM structure and biochemistry characterization of gp105, a protein that is encoded by the jumbo phage 201φ2-1 and is involved in the formation of the nucleus-like compartment in phage 201φ2-1 infected . We found that, although most gp105 molecules are in the monomeric state in solution, a small portion of gp105 assemble to form large sheet-like assemblies and small cube-like particles. Reconstruction of the cube-like particles showed that the particle consists of six flat head-to-tail tetramers arranged into an octahedral cube. The four molecules at the contact interface of two head-to-tail tetramers are 2-fold symmetry-related and constitute a concave tetramer. Further reconstructions without applying symmetry showed that molecules in the particles around the distal ends of a 3-fold axis are highly dynamic and have the tendency to open up the assembly. Local classifications and refinements of the concave tetramers in the cube-like particle resulted in a map of the concave tetramer at a resolution of 4.09 Å. Structural analysis of the concave tetramer indicates that the N and C terminal fragments of gp105 are important for mediating the intermolecular interactions, which was further confirmed by mutagenesis studies. Biochemistry assays showed that, in solution, the cube-like particles of gp105 are liable to either disassemble to form the monomers or recruit more molecules to form the high molecular weight lattice-like assembly. We also found that monomeric gp105s can self-assemble to form large sheet-like assemblies , and the assembly of gp105 is a reversible dynamic process and temperature-dependent. Taken together, our results revealed the dynamic assembly of gp105, which helps to understand the development and function of the nucleus-like compartment assembled by phage-encoded proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35432.map.gz emd_35432.map.gz | 3.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35432-v30.xml emd-35432-v30.xml emd-35432.xml emd-35432.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35432_fsc.xml emd_35432_fsc.xml | 6.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35432.png emd_35432.png | 156.6 KB | ||
| Filedesc metadata |  emd-35432.cif.gz emd-35432.cif.gz | 5.5 KB | ||
| その他 |  emd_35432_half_map_1.map.gz emd_35432_half_map_1.map.gz emd_35432_half_map_2.map.gz emd_35432_half_map_2.map.gz | 16.1 MB 16.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35432 http://ftp.pdbj.org/pub/emdb/structures/EMD-35432 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35432 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35432 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35432_validation.pdf.gz emd_35432_validation.pdf.gz | 729.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35432_full_validation.pdf.gz emd_35432_full_validation.pdf.gz | 729.3 KB | 表示 | |
| XML形式データ |  emd_35432_validation.xml.gz emd_35432_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  emd_35432_validation.cif.gz emd_35432_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35432 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35432 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35432 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35432 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8iggMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35432.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35432.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The cryo-EM map of the phage-encoded nucleus-like shell protein gp105 concave tetramer reconstructed with C2 symmetry imposed. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.25 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: The cryo-EM half map of the phage-encoded nucleus-like...
| ファイル | emd_35432_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The cryo-EM half map of the phage-encoded nucleus-like shell protein gp105 concave tetramer reconstructed with C2 symmetry imposed. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: The cryo-EM half map of the phage-encoded nucleus-like...
| ファイル | emd_35432_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The cryo-EM half map of the phage-encoded nucleus-like shell protein gp105 concave tetramer reconstructed with C2 symmetry imposed. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 201Phi2-1 gp105
| 全体 | 名称: 201Phi2-1 gp105 |
|---|---|
| 要素 |
|
-超分子 #1: 201Phi2-1 gp105
| 超分子 | 名称: 201Phi2-1 gp105 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage 201phi2-1 (ファージ) Pseudomonas phage 201phi2-1 (ファージ) |
-分子 #1: Chimallin
| 分子 | 名称: Chimallin / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage 201phi2-1 (ファージ) Pseudomonas phage 201phi2-1 (ファージ) |
| 分子量 | 理論値: 70.654516 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIRDTATNTT QTQAAPQQAP AQQFTQAPQE KPMQSTQSQP TPSYAGTGGI NSQFTRSGNV QGGDARASEA LTVFTRLKEQ AVAQQDLAD DFSILRFDRD QHQVGWSSLV IAKQISLNGQ PVIAVRPLIL PNNSIELPKR KTNIVNGMQT DVIESDIDVG T VFSAQYFN ...文字列: MIRDTATNTT QTQAAPQQAP AQQFTQAPQE KPMQSTQSQP TPSYAGTGGI NSQFTRSGNV QGGDARASEA LTVFTRLKEQ AVAQQDLAD DFSILRFDRD QHQVGWSSLV IAKQISLNGQ PVIAVRPLIL PNNSIELPKR KTNIVNGMQT DVIESDIDVG T VFSAQYFN RLSTYVQNTL GKPGAKVVLA GPFPIPADLV LKDSELQLRN LLIKSVNACD DILALHSGER PFTIAGLKGQ QG ETLAAKV DIRTQPLHDT VGNPIRADIV VTTQRVRRNG QQENEFYETD VKLNQVAMFT NLERTPQAQA QTLFPNQQQV ATP APWVAS VVITDVRNAD GIQANTPEMY WFALSNAFRS THGHAWARPF LPMTGVAKDM KDIGALGWMS ALRNRIDTKA ANFD DAQFG QLMLSQVQPN PVFQIDLNRM GETAQMDSLQ LDAAGGPNAQ KAAATIIRQI NNLGGGGFER FFDHTTQPIL ERTGQ VIDL GNWFDGDEKR DRRDLDNLAA LNAAEGNENE FWGFYGAQLN PNLHPDLRNR QSRNYDRQYL GSTVTYTGKA ERCTYN AKF IEALDRYLAE AGLQITMDNT SVLNSGQRFM GNSVIGNNMV SGQAQVHSAY AGTQGFNTQY QTGPSSFYAL EHHHHHH UniProtKB: Chimallin |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)