+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the ISFba1 TnpB-reRNA-dsDNA complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA / Complex / RNA BINDING PROTEIN/RNA / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 機能・相同性 | :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Firmicutes bacterium AM43-11BH (バクテリア) Firmicutes bacterium AM43-11BH (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Yin M / Zhou F / Zhu Y / Huang Z | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2024 ジャーナル: Cell Res / 年: 2024タイトル: Discovery and structural mechanism of DNA endonucleases guided by RAGATH-18-derived RNAs. 著者: Kuan Ren / Fengxia Zhou / Fan Zhang / Mingyu Yin / Yuwei Zhu / Shouyu Wang / Yan Chen / Tengjin Huang / Zixuan Wu / Jiale He / Anqi Zhang / Changyou Guo / Zhiwei Huang /  要旨: CRISPR-Cas systems and IS200/IS605 transposon-associated TnpBs have been utilized for the development of genome editing technologies. Using bioinformatics analysis and biochemical experiments, here ...CRISPR-Cas systems and IS200/IS605 transposon-associated TnpBs have been utilized for the development of genome editing technologies. Using bioinformatics analysis and biochemical experiments, here we present a new family of RNA-guided DNA endonucleases. Our bioinformatics analysis initially identifies the stable co-occurrence of conserved RAGATH-18-derived RNAs (reRNAs) and their upstream IS607 TnpBs with an average length of 390 amino acids. IS607 TnpBs form programmable DNases through interaction with reRNAs. We discover the robust dsDNA interference activity of IS607 TnpB systems in bacteria and human cells. Further characterization of the Firmicutes bacteria IS607 TnpB system (ISFba1 TnpB) reveals that its dsDNA cleavage activity is remarkably sensitive to single mismatches between the guide and target sequences in human cells. Our findings demonstrate that a length of 20 nt in the guide sequence of reRNA achieves the highest DNA cleavage activity for ISFba1 TnpB. A cryo-EM structure of the ISFba1 TnpB effector protein bound by its cognate RAGATH-18 motif-containing reRNA and a dsDNA target reveals the mechanisms underlying reRNA recognition by ISFba1 TnpB, reRNA-guided dsDNA targeting, and the sensitivity of the ISFba1 TnpB system to base mismatches between the guide and target DNA. Collectively, this study identifies the IS607 TnpB family of compact and specific RNA-guided DNases with great potential for application in gene editing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35323.map.gz emd_35323.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35323-v30.xml emd-35323-v30.xml emd-35323.xml emd-35323.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35323.png emd_35323.png | 77.9 KB | ||
| Filedesc metadata |  emd-35323.cif.gz emd-35323.cif.gz | 5.9 KB | ||
| その他 |  emd_35323_half_map_1.map.gz emd_35323_half_map_1.map.gz emd_35323_half_map_2.map.gz emd_35323_half_map_2.map.gz | 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35323 http://ftp.pdbj.org/pub/emdb/structures/EMD-35323 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35323 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35323 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35323_validation.pdf.gz emd_35323_validation.pdf.gz | 822.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35323_full_validation.pdf.gz emd_35323_full_validation.pdf.gz | 822.3 KB | 表示 | |
| XML形式データ |  emd_35323_validation.xml.gz emd_35323_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_35323_validation.cif.gz emd_35323_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35323 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35323 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35323 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35323 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8iazMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35323.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35323.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_35323_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
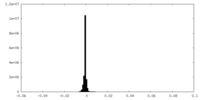
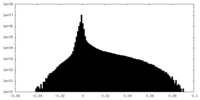
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35323_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
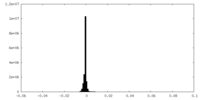
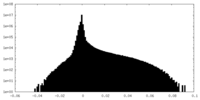
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The complex of protein with gRNA and target DNA
| 全体 | 名称: The complex of protein with gRNA and target DNA |
|---|---|
| 要素 |
|
-超分子 #1: The complex of protein with gRNA and target DNA
| 超分子 | 名称: The complex of protein with gRNA and target DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Firmicutes bacterium AM43-11BH (バクテリア) Firmicutes bacterium AM43-11BH (バクテリア) |
-分子 #1: Transposase
| 分子 | 名称: Transposase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Firmicutes bacterium AM43-11BH (バクテリア) Firmicutes bacterium AM43-11BH (バクテリア) |
| 分子量 | 理論値: 44.549645 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: LLKSFKTEIN PSEEQKVKIH KTIGTCRFIY NFYLAHNKEL YDKGEKFMSG KSFSVWLNNE YLPQNPDKLW IKEVSSKSVK HSIENGCIA FTRFFKHQSA FPNLKKKGKS DVKMYFVKNN PKDCRCERHR INIPSLGWVR IKEKGYIPTT KDGYVIKSGT V SMKADRYY ...文字列: LLKSFKTEIN PSEEQKVKIH KTIGTCRFIY NFYLAHNKEL YDKGEKFMSG KSFSVWLNNE YLPQNPDKLW IKEVSSKSVK HSIENGCIA FTRFFKHQSA FPNLKKKGKS DVKMYFVKNN PKDCRCERHR INIPSLGWVR IKEKGYIPTT KDGYVIKSGT V SMKADRYY VSVLVEISNN KIANNSNAGI GIDLGLKDFA IVSNGKTYKN INKSARLKKH EKQLIREQRS LSRKYENLKK GE STQKANI QKQRLKVQKL HHRMDNIRTD YINKTIAEIV KTKPSYITIE DLNVKGMMKN RHLSKAVASQ KFYEFRTKLQ AKC NENGIE LRVVDRWYPS SKTCHCCGAV KKDLKLSDRI FKCSCGYVED RAFNAALNLR DAITYEVA UniProtKB: UNIPROTKB: A0A417B524 |
-分子 #2: RNA (207-MER)
| 分子 | 名称: RNA (207-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Firmicutes bacterium AM43-11BH (バクテリア) Firmicutes bacterium AM43-11BH (バクテリア) |
| 分子量 | 理論値: 66.616352 KDa |
| 配列 | 文字列: CUAAAACGCA AACGUAAGUA UGUACCGAAG GCUAUUUUCG GGAAUUUACG ACUAUGGAGU GUACAAGAAC UUGUGAGUAG AUAUGAUUU CGGUCAAUCC AAAGCAUACA CGAUGAAAUA GUAAGUAAUG UUCGUGAGAA CUUACAUUAU CUCGAUGUGA G CAUAUUUA ...文字列: CUAAAACGCA AACGUAAGUA UGUACCGAAG GCUAUUUUCG GGAAUUUACG ACUAUGGAGU GUACAAGAAC UUGUGAGUAG AUAUGAUUU CGGUCAAUCC AAAGCAUACA CGAUGAAAUA GUAAGUAAUG UUCGUGAGAA CUUACAUUAU CUCGAUGUGA G CAUAUUUA AUCACAUUUU GAGUGGCAGC UGAUGGUCCA UGUCUGUUA |
-分子 #3: DNA (5'-D(P*AP*CP*AP*TP*GP*GP*AP*CP*CP*AP*TP*CP*AP*GP*CP*TP*CP*CP...
| 分子 | 名称: DNA (5'-D(P*AP*CP*AP*TP*GP*GP*AP*CP*CP*AP*TP*CP*AP*GP*CP*TP*CP*CP*TP*AP*AP*TP*GP*G)-3') タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Firmicutes bacterium AM43-11BH (バクテリア) Firmicutes bacterium AM43-11BH (バクテリア) |
| 分子量 | 理論値: 7.338756 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DT)(DG)(DG)(DA)(DC)(DC)(DA) (DT)(DC)(DA)(DG)(DC)(DT)(DC)(DC)(DT)(DA) (DA)(DT)(DG)(DG) |
-分子 #4: DNA (5'-D(P*CP*CP*AP*TP*TP*AP*GP*GP*AP*GP*CP*TP*GP*AP*TP*G)-3')
| 分子 | 名称: DNA (5'-D(P*CP*CP*AP*TP*TP*AP*GP*GP*AP*GP*CP*TP*GP*AP*TP*G)-3') タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Firmicutes bacterium AM43-11BH (バクテリア) Firmicutes bacterium AM43-11BH (バクテリア) |
| 分子量 | 理論値: 4.938216 KDa |
| 配列 | 文字列: (DC)(DC)(DA)(DT)(DT)(DA)(DG)(DG)(DA)(DG) (DC)(DT)(DG)(DA)(DT)(DG) |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 369379 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)