+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3-fold block local reconstruction of SFTSV virion | |||||||||
 マップデータ マップデータ | C3 block local reconstruction | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SFTSV / virion structure / Gn/Gc glycoproteins / VIRUS / VIRAL PROTEIN | |||||||||
| 生物種 |  Dabie bandavirus (ウイルス) Dabie bandavirus (ウイルス) | |||||||||
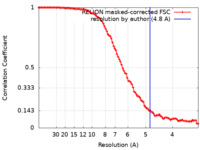
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Du S / Peng R / Qi J / Li C | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structure of severe fever with thrombocytopenia syndrome virus. 著者: Shouwen Du / Ruchao Peng / Wang Xu / Xiaoyun Qu / Yuhang Wang / Jiamin Wang / Letian Li / Mingyao Tian / Yudong Guan / Jigang Wang / Guoqing Wang / Hao Li / Lingcong Deng / Xiaoshuang Shi / ...著者: Shouwen Du / Ruchao Peng / Wang Xu / Xiaoyun Qu / Yuhang Wang / Jiamin Wang / Letian Li / Mingyao Tian / Yudong Guan / Jigang Wang / Guoqing Wang / Hao Li / Lingcong Deng / Xiaoshuang Shi / Yidan Ma / Fengting Liu / Minhua Sun / Zhengkai Wei / Ningyi Jin / Wei Liu / Jianxun Qi / Quan Liu / Ming Liao / Chang Li /   要旨: The severe fever with thrombocytopenia syndrome virus (SFTSV) is a tick-borne human-infecting bunyavirus, which utilizes two envelope glycoproteins, Gn and Gc, to enter host cells. However, the ...The severe fever with thrombocytopenia syndrome virus (SFTSV) is a tick-borne human-infecting bunyavirus, which utilizes two envelope glycoproteins, Gn and Gc, to enter host cells. However, the structure and organization of these glycoproteins on virion surface are not yet known. Here we describe the structure of SFTSV determined by single particle reconstruction, which allows mechanistic insights into bunyavirus assembly at near-atomic resolution. The SFTSV Gn and Gc proteins exist as heterodimers and further assemble into pentameric and hexameric peplomers, shielding the Gc fusion loops by both intra- and inter-heterodimer interactions. Individual peplomers are associated mainly through the ectodomains, in which the highly conserved glycans on N914 of Gc play a crucial role. This elaborate assembly stabilizes Gc in the metastable prefusion conformation and creates some cryptic epitopes that are only accessible in the intermediate states during virus entry. These findings provide an important basis for developing vaccines and therapeutic drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35177.map.gz emd_35177.map.gz | 30 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35177-v30.xml emd-35177-v30.xml emd-35177.xml emd-35177.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35177_fsc.xml emd_35177_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35177.png emd_35177.png | 97.5 KB | ||
| Filedesc metadata |  emd-35177.cif.gz emd-35177.cif.gz | 5.5 KB | ||
| その他 |  emd_35177_half_map_1.map.gz emd_35177_half_map_1.map.gz emd_35177_half_map_2.map.gz emd_35177_half_map_2.map.gz | 67.6 MB 67.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35177 http://ftp.pdbj.org/pub/emdb/structures/EMD-35177 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35177 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35177 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35177_validation.pdf.gz emd_35177_validation.pdf.gz | 974 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35177_full_validation.pdf.gz emd_35177_full_validation.pdf.gz | 973.5 KB | 表示 | |
| XML形式データ |  emd_35177_validation.xml.gz emd_35177_validation.xml.gz | 17 KB | 表示 | |
| CIF形式データ |  emd_35177_validation.cif.gz emd_35177_validation.cif.gz | 22.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35177 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35177 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35177 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35177 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35177.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35177.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C3 block local reconstruction | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.68 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: C3 block local reconstruction half map 1
| ファイル | emd_35177_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C3 block local reconstruction half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
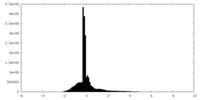
| 密度ヒストグラム |
-ハーフマップ: C3 block local reconstruction half map 1
| ファイル | emd_35177_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C3 block local reconstruction half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dabie bandavirus
| 全体 | 名称:  Dabie bandavirus (ウイルス) Dabie bandavirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Dabie bandavirus
| 超分子 | 名称: Dabie bandavirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2748958 / 生物種: Dabie bandavirus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|
-分子 #1: Gn glycoprotein
| 分子 | 名称: Gn glycoprotein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Dabie bandavirus (ウイルス) Dabie bandavirus (ウイルス) |
| 配列 | 文字列: MMKVIWFSSL ICLVIQCGGD SGPIICAGPI HSNKSANIPH LLGYSEKICQ IDRLIHVSSW LRNHSQFQGY VGQRGGRSQV SYYPAENSY SRWSGLLSPC DADWLGMLVV KKAKGSDMIV PGPSYKGKVF FERPTFDGYV GWGCSSGKSR TESGELCSSD S GTSSGLLP ...文字列: MMKVIWFSSL ICLVIQCGGD SGPIICAGPI HSNKSANIPH LLGYSEKICQ IDRLIHVSSW LRNHSQFQGY VGQRGGRSQV SYYPAENSY SRWSGLLSPC DADWLGMLVV KKAKGSDMIV PGPSYKGKVF FERPTFDGYV GWGCSSGKSR TESGELCSSD S GTSSGLLP SDRVLWIGDV ACQPMTPIPE ETFLELKSFS QSEFPDICKI DGVVFNQCEG ESLPQPFDVA WMDVGHSHKI IM REHKTKW VQESSSKDFV CYKAGTGPCS ESEEKTCKTS GSCRGDMQFC KVAGCEHGEE ASEAKCRCSL VHKPGEVVVS YGG MRVRPK CYGFSRMMAT LEVNPPEQRT GQCTGCHLEC INGGVRLITL TSELKSATVC ASHFCSSATS GKKSTEIQFH SGSL VGRTA IHVKGALVDG TEFTFEGSCM FPDGCDAVDC TFCREFLKNP QCYPAKKWLF IIIVILLGYA GLMLLTNVLK AIGIW GSWV IAPVKLIFAI IKKLMRTVSC LMGKLMDRGR QVIHEEIGEN REGNQDDVRI EMARPRRVRH WMYSPVILTI LAIGLA |
-分子 #2: Gc glycoprotein
| 分子 | 名称: Gc glycoprotein / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Dabie bandavirus (ウイルス) Dabie bandavirus (ウイルス) |
| 配列 | 文字列: ESCDEMVHAD SKLVSCRQGS GNMKECVTTG RALLPAVNPG QEACLHFTAP GSPDSKCLKI KVKRINLKCK KSSSYFVPDA RSRCTSVRR CRWAGDCQSG CPPHSTSNSF SDDWAGKMDR AGLGFSGCSD GCGGAACGCF NAAPSCIFWR KWVENPHGII W KVSPCAAW ...文字列: ESCDEMVHAD SKLVSCRQGS GNMKECVTTG RALLPAVNPG QEACLHFTAP GSPDSKCLKI KVKRINLKCK KSSSYFVPDA RSRCTSVRR CRWAGDCQSG CPPHSTSNSF SDDWAGKMDR AGLGFSGCSD GCGGAACGCF NAAPSCIFWR KWVENPHGII W KVSPCAAW VPSAVIELTM PSGEVRTFHP MSGIPTQVFK GVSVTYLGSD MEVSGLTDLC EIEELKSKKL ALAPCNQAGM GV VGKVGEI QCSSEESART IKKDGCIWNA DLVGIELRVD DAVCYSKITS VEAVANYSAI PTTIGGLRFE RSHDSLGKIS GSP LDITAI RGSFSVNYRG LRLSLSEITA TCTGEVTNVS GCYSCMTGAK VSIKLHSSKN STAHVRCKGD ETAFSVLEGV HSYT VSLSF DHAVVDEQCQ LNCGGHESQV TLKGNLIFLD VPKFVDGSYM QTYHSSVPTG ANIPSPTDWL NALFGNGLSR WILGV IGVL LGGLALFFLI MSLFKLGTKQ VFRSRTKLA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)