+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of D5 ATP-ADP form | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MPVX / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Li YN / Zhu J / Guo YY / Yan RH | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural insight into the assembly and working mechanism of helicase-primase D5 from Mpox virus. 著者: Yaning Li / Jing Zhu / Yingying Guo / Renhong Yan /  要旨: The Mpox pandemic, caused by the Mpox virus (or monkeypox virus, MPXV), has gained global attention. The D5 protein, a putative helicase-primase found in MPXV, plays a vital role in viral replication ...The Mpox pandemic, caused by the Mpox virus (or monkeypox virus, MPXV), has gained global attention. The D5 protein, a putative helicase-primase found in MPXV, plays a vital role in viral replication and genome uncoating. Here we determined multiple cryo-EM structures of full-length hexameric D5 in diverse states. These states were captured during ATP hydrolysis while moving along the single-stranded DNA (ssDNA) track. Through comprehensive structural analysis combined with the helicase activity system, we revealed that when the primase domain is truncated or the interaction between the primase and helicase domains is disrupted, the double-stranded DNA (dsDNA) unwinds into ssDNA, suggesting a critical regulatory role of the primase domain. Two transition states bound with ssDNA substrate during unwinding reveals that two ATP molecules were consumed to drive DNA moving forward two nucleotides. Collectively, our findings shed light on the molecular mechanism that links ATP hydrolysis to the DNA unwinding in poxviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35055.map.gz emd_35055.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35055-v30.xml emd-35055-v30.xml emd-35055.xml emd-35055.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35055.png emd_35055.png | 26.6 KB | ||
| Filedesc metadata |  emd-35055.cif.gz emd-35055.cif.gz | 6 KB | ||
| その他 |  emd_35055_half_map_1.map.gz emd_35055_half_map_1.map.gz emd_35055_half_map_2.map.gz emd_35055_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35055 http://ftp.pdbj.org/pub/emdb/structures/EMD-35055 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35055 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35055 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35055_validation.pdf.gz emd_35055_validation.pdf.gz | 816 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35055_full_validation.pdf.gz emd_35055_full_validation.pdf.gz | 815.6 KB | 表示 | |
| XML形式データ |  emd_35055_validation.xml.gz emd_35055_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_35055_validation.cif.gz emd_35055_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35055 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35055 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35055 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35055 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hweMC  8hwaC  8hwbC  8hwcC  8hwdC  8hwfC  8hwgC  8hwhC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35055.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35055.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0773 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35055_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
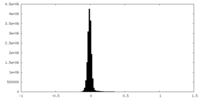
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35055_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
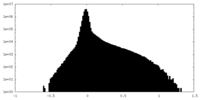
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : D5 ATP-ADP form
| 全体 | 名称: D5 ATP-ADP form |
|---|---|
| 要素 |
|
-超分子 #1: D5 ATP-ADP form
| 超分子 | 名称: D5 ATP-ADP form / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
-分子 #1: Primase D5
| 分子 | 名称: Primase D5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 90.476344 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAAIRGNDV IFVLKTIGVP SACRQNEDPR FVEAFKCDEL ERYIDNNPEC TLFESLRDEE AYSIVRIFMD VDLDACLDEI DYLTAIQDF IIEVSNCVAR FAFTECGAIH ENVIKSMRSN FSLTKSTNRD KTSFHIIFLD TYTTMDTLIA MKRTLLELSR S SENPLTRS ...文字列: MDAAIRGNDV IFVLKTIGVP SACRQNEDPR FVEAFKCDEL ERYIDNNPEC TLFESLRDEE AYSIVRIFMD VDLDACLDEI DYLTAIQDF IIEVSNCVAR FAFTECGAIH ENVIKSMRSN FSLTKSTNRD KTSFHIIFLD TYTTMDTLIA MKRTLLELSR S SENPLTRS IDTAVYRRKT TLRVVGTRKN PNCDTIHVMQ PPHDNIEDYL FTYVDMNNNS YYFSLQRRLE DLVPDKLWEP GF ISFEDAI KRVSKIFINS IINFNDLDEN NFTTVPLVID YVTPCALCKK RSHKHPHQLS LENGAIRIYK TGNPHSCKVK IVP LDGNKL FNIAQRILDT NSVLLTERGD HIVWINNSWK FNSEEPLITK LILSIRHQLP KEYSSELLCP RKRKTVEANI RDML VDSVE TDTYPDKLPF KNGVLDLVDG MFYSGDDAKK YTCTVSTGFK FDDTKFVEDS PEMEELMNII NDIQPLTDEN KKNRE LYEK TLSSCLCGAT KGCLTFFFGE TATGKSTTKR LLKSAIGDLF VETGQTILTD VLDKGPNPFI ANMHLKRSVF CSELPD FAC SGSKKIRSDN IKKLTEPCVI GRPCFSNKIN NRNHATIIID TNYKPVFDRI DNALMRRIAV VRFRTHFSQP SGREAAE NN DAYDKVKLLD EGLDGKIQNN RYRFAFLYLL VKWYKKYHIP IMKLYPTPEE IPDFAFYLKI GTLLVSSSVK HIPLMTDL S KKGYILYDNV VTLPLTTFQQ KISKYFNSRL FGHDIESFIN RHKKFANVSD EYLQYIFIED ISSP UniProtKB: Uncoating factor OPG117 |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 5 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 5 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0.6) / 使用した粒子像数: 321782 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)