登録情報 データベース : EMDB / ID : EMD-35041タイトル Cryo-EM structure of the J-K-St region of EMCV IRES in complex with eIF4G-HEAT1 and eIF4A Post-process map 複合体 : Ternary complex of the J-K-St region of EMCV IRES with eIF4A and eIF4G-HEAT1タンパク質・ペプチド : Eukaryotic initiation factor 4A-Iタンパク質・ペプチド : Eukaryotic translation initiation factor 4 gamma 1RNA : IRES RNA (J-K-St)リガンド : MAGNESIUM IONリガンド : water / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / 手法 / / 解像度 : 3.76 Å Suzuki H / Fujiyoshi Y / Imai S / Shimada I 資金援助 Organization Grant number 国 Japan Agency for Medical Research and Development (AMED) 21ae0121028h0001 Japan Society for the Promotion of Science (JSPS) 20H00451
ジャーナル : Nat Commun / 年 : 2023タイトル : Dynamically regulated two-site interaction of viral RNA to capture host translation initiation factor.著者 : Shunsuke Imai / Hiroshi Suzuki / Yoshinori Fujiyoshi / Ichio Shimada / 要旨 : Many RNA viruses employ internal ribosome entry sites (IRESs) in their genomic RNA to commandeer the host's translational machinery for replication. The IRES from encephalomyocarditis virus (EMCV) ... Many RNA viruses employ internal ribosome entry sites (IRESs) in their genomic RNA to commandeer the host's translational machinery for replication. The IRES from encephalomyocarditis virus (EMCV) interacts with eukaryotic translation initiation factor 4 G (eIF4G), recruiting the ribosomal subunit for translation. Here, we analyze the three-dimensional structure of the complex composed of EMCV IRES, the HEAT1 domain fragment of eIF4G, and eIF4A, by cryo-electron microscopy. Two distinct eIF4G-interacting domains on the IRES are identified, and complex formation changes the angle therebetween. Further, we explore the dynamics of these domains by using solution NMR spectroscopy, revealing conformational equilibria in the microsecond to millisecond timescale. In the lowly-populated conformations, the base-pairing register of one domain is shifted with the structural transition of the three-way junction, as in the complex structure. Our study provides insights into the viral RNA's sophisticated strategy for optimal docking to hijack the host protein. 履歴 登録 2022年12月24日 - ヘッダ(付随情報) 公開 2023年8月2日 - マップ公開 2023年8月2日 - 更新 2023年9月13日 - 現状 2023年9月13日 処理サイト : PDBj / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) /
Homo sapiens (ヒト) /  Encephalomyocarditis virus (脳心筋炎ウイルス)
Encephalomyocarditis virus (脳心筋炎ウイルス) データ登録者
データ登録者 日本, 2件
日本, 2件  引用
引用 ジャーナル: Nat Commun / 年: 2023
ジャーナル: Nat Commun / 年: 2023
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_35041.map.gz
emd_35041.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-35041-v30.xml
emd-35041-v30.xml emd-35041.xml
emd-35041.xml EMDBヘッダ
EMDBヘッダ emd_35041_fsc.xml
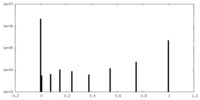
emd_35041_fsc.xml FSCデータファイル
FSCデータファイル emd_35041.png
emd_35041.png emd_35041_msk_1.map
emd_35041_msk_1.map マスクマップ
マスクマップ emd_35041_half_map_1.map.gz
emd_35041_half_map_1.map.gz emd_35041_half_map_2.map.gz
emd_35041_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-35041
http://ftp.pdbj.org/pub/emdb/structures/EMD-35041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35041
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35041 emd_35041_validation.pdf.gz
emd_35041_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_35041_full_validation.pdf.gz
emd_35041_full_validation.pdf.gz emd_35041_validation.xml.gz
emd_35041_validation.xml.gz emd_35041_validation.cif.gz
emd_35041_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35041
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35041 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35041
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35041

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
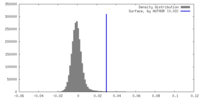
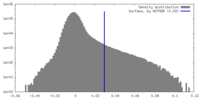
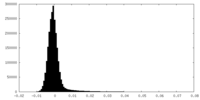
マップ ダウンロード / ファイル: emd_35041.map.gz / 形式: CCP4 / 大きさ: 9.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_35041.map.gz / 形式: CCP4 / 大きさ: 9.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_35041_msk_1.map
emd_35041_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Encephalomyocarditis virus (脳心筋炎ウイルス)
Encephalomyocarditis virus (脳心筋炎ウイルス)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)