+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CXCR3-DNGi complex activated by VUF11222 | |||||||||
 マップデータ マップデータ | sharpened map (Phenix.AutoSharpen) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | G protein coupled receptor / chemokine receptor / CXCR3 / VUF11222 / complex / SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Oplophorus-luciferin 2-monooxygenase / Oplophorus-luciferin 2-monooxygenase activity / regulation of leukocyte migration / chemokine binding / C-X-C chemokine binding / chemokine receptor activity / C-X-C chemokine receptor activity / C-C chemokine receptor activity / positive regulation of chemotaxis / C-C chemokine binding ...Oplophorus-luciferin 2-monooxygenase / Oplophorus-luciferin 2-monooxygenase activity / regulation of leukocyte migration / chemokine binding / C-X-C chemokine binding / chemokine receptor activity / C-X-C chemokine receptor activity / C-C chemokine receptor activity / positive regulation of chemotaxis / C-C chemokine binding / negative regulation of execution phase of apoptosis / Chemokine receptors bind chemokines / negative regulation of endothelial cell proliferation / positive regulation of execution phase of apoptosis / regulation of cell adhesion / positive regulation of protein localization to cell cortex / Adenylate cyclase inhibitory pathway / T cell migration / D2 dopamine receptor binding / response to prostaglandin E / G protein-coupled serotonin receptor binding / adenylate cyclase regulator activity / adenylate cyclase-inhibiting serotonin receptor signaling pathway / cellular response to forskolin / regulation of mitotic spindle organization / negative regulation of angiogenesis / bioluminescence / positive regulation of release of sequestered calcium ion into cytosol / cell chemotaxis / Regulation of insulin secretion / positive regulation of cholesterol biosynthetic process / G protein-coupled receptor binding / calcium-mediated signaling / electron transport chain / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / G-protein beta/gamma-subunit complex binding / response to peptide hormone / centriolar satellite / Olfactory Signaling Pathway / Activation of the phototransduction cascade / G beta:gamma signalling through PLC beta / Presynaptic function of Kainate receptors / Thromboxane signalling through TP receptor / G protein-coupled acetylcholine receptor signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / G-protein activation / Activation of G protein gated Potassium channels / Inhibition of voltage gated Ca2+ channels via Gbeta/gamma subunits / Prostacyclin signalling through prostacyclin receptor / G beta:gamma signalling through CDC42 / Glucagon signaling in metabolic regulation / G beta:gamma signalling through BTK / positive regulation of angiogenesis / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) / ADP signalling through P2Y purinoceptor 12 / chemotaxis / Sensory perception of sweet, bitter, and umami (glutamate) taste / photoreceptor disc membrane / Glucagon-type ligand receptors / Adrenaline,noradrenaline inhibits insulin secretion / Vasopressin regulates renal water homeostasis via Aquaporins / GDP binding / G alpha (z) signalling events / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / cellular response to catecholamine stimulus / ADORA2B mediated anti-inflammatory cytokines production / ADP signalling through P2Y purinoceptor 1 / G beta:gamma signalling through PI3Kgamma / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / adenylate cyclase-activating dopamine receptor signaling pathway / GPER1 signaling / Inactivation, recovery and regulation of the phototransduction cascade / cellular response to prostaglandin E stimulus / G-protein beta-subunit binding / heterotrimeric G-protein complex / G alpha (12/13) signalling events / sensory perception of taste / extracellular vesicle / signaling receptor complex adaptor activity / signaling receptor activity / Thrombin signalling through proteinase activated receptors (PARs) / G protein activity / positive regulation of cytosolic calcium ion concentration / GTPase binding / retina development in camera-type eye / Ca2+ pathway / midbody / cell cortex / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / fibroblast proliferation / G alpha (i) signalling events / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / G alpha (s) signalling events / phospholipase C-activating G protein-coupled receptor signaling pathway / angiogenesis / G alpha (q) signalling events / Ras protein signal transduction / periplasmic space / Extra-nuclear estrogen signaling 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Oplophorus gracilirostris (甲殻類) Oplophorus gracilirostris (甲殻類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.94 Å | |||||||||
 データ登録者 データ登録者 | Jiao HZ / Hu HL | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural insights into the activation and inhibition of CXC chemokine receptor 3. 著者: Haizhan Jiao / Bin Pang / Aijun Liu / Qiang Chen / Qi Pan / Xiankun Wang / Yunong Xu / Ying-Chih Chiang / Ruobing Ren / Hongli Hu /  要旨: The chemotaxis of CD4 type 1 helper cells and CD8 cytotoxic lymphocytes, guided by interferon-inducible CXC chemokine 9-11 (CXCL9-11) and CXC chemokine receptor 3 (CXCR3), plays a critical role in ...The chemotaxis of CD4 type 1 helper cells and CD8 cytotoxic lymphocytes, guided by interferon-inducible CXC chemokine 9-11 (CXCL9-11) and CXC chemokine receptor 3 (CXCR3), plays a critical role in type 1 immunity. Here we determined the structures of human CXCR3-DNG complexes activated by chemokine CXCL11, peptidomimetic agonist PS372424 and biaryl-type agonist VUF11222, and the structure of inactive CXCR3 bound to noncompetitive antagonist SCH546738. Structural analysis revealed that PS372424 shares a similar orthosteric binding pocket to the N terminus of CXCL11, while VUF11222 buries deeper and activates the receptor in a distinct manner. We showed an allosteric binding site between TM5 and TM6, accommodating SCH546738 in the inactive CXCR3. SCH546738 may restrain the receptor at an inactive state by preventing the repacking of TM5 and TM6. By revealing the binding patterns and the pharmacological properties of the four modulators, we present the activation mechanisms of CXCR3 and provide insights for future drug development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34916.map.gz emd_34916.map.gz | 64.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34916-v30.xml emd-34916-v30.xml emd-34916.xml emd-34916.xml | 24.9 KB 24.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34916.png emd_34916.png | 112.6 KB | ||
| Filedesc metadata |  emd-34916.cif.gz emd-34916.cif.gz | 7.2 KB | ||
| その他 |  emd_34916_additional_1.map.gz emd_34916_additional_1.map.gz emd_34916_additional_2.map.gz emd_34916_additional_2.map.gz emd_34916_half_map_1.map.gz emd_34916_half_map_1.map.gz emd_34916_half_map_2.map.gz emd_34916_half_map_2.map.gz | 41.2 MB 65 MB 65.5 MB 65.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34916 http://ftp.pdbj.org/pub/emdb/structures/EMD-34916 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34916 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34916 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34916.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34916.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map (Phenix.AutoSharpen) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: scaled map (CCPEM.LocalScale)
| ファイル | emd_34916_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | scaled map (CCPEM.LocalScale) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
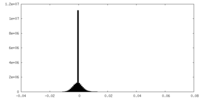
| 密度ヒストグラム |
-追加マップ: unsharpened map (Relion.AutoSharpen)
| ファイル | emd_34916_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map (Relion.AutoSharpen) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
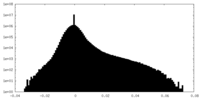
| 密度ヒストグラム |
-ハーフマップ: half map A (Relion.AutoRefine)
| ファイル | emd_34916_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A (Relion.AutoRefine) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
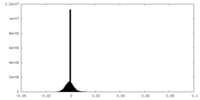
| 密度ヒストグラム |
-ハーフマップ: half map B (Relion.AutoRefine)
| ファイル | emd_34916_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B (Relion.AutoRefine) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
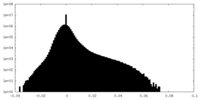
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CXCR3-VUF11222-DNGi-scFv16
| 全体 | 名称: CXCR3-VUF11222-DNGi-scFv16 |
|---|---|
| 要素 |
|
-超分子 #1: CXCR3-VUF11222-DNGi-scFv16
| 超分子 | 名称: CXCR3-VUF11222-DNGi-scFv16 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Guanine nucleotide-binding protein G(i) subunit alpha-1
| 分子 | 名称: Guanine nucleotide-binding protein G(i) subunit alpha-1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 40.445059 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGCTLSAEDK AAVERSKMID RNLREDGEKA AREVKLLLLG AGESGKSTIV KQMKIIHEAG YSEEECKQYK AVVYSNTIQS IIAIIRAMG RLKIDFGDSA RADDARQLFV LAGAAEEGFM TAELAGVIKR LWKDSGVQAC FNRSREYQLN DSAAYYLNDL D RIAQPNYI ...文字列: MGCTLSAEDK AAVERSKMID RNLREDGEKA AREVKLLLLG AGESGKSTIV KQMKIIHEAG YSEEECKQYK AVVYSNTIQS IIAIIRAMG RLKIDFGDSA RADDARQLFV LAGAAEEGFM TAELAGVIKR LWKDSGVQAC FNRSREYQLN DSAAYYLNDL D RIAQPNYI PTQQDVLRTR VKTTGIVETH FTFKDLHFKM FDVGAQRSER KKWIHCFEGV TAIIFCVALS DYDLVLAEDE EM NRMHESM KLFDSICNNK WFTDTSIILF LNKKDLFEEK IKKSPLTICY PEYAGSNTYE EAAAYIQCQF EDLNKRKDTK EIY THFTCS TDTKNVQFVF DAVTDVIIKN NLKDCGLF UniProtKB: Guanine nucleotide-binding protein G(i) subunit alpha-1 |
-分子 #2: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1
| 分子 | 名称: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 42.005895 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHHHHH LEVLFQGPGS SGSELDQLRQ EAEQLKNQIR DARKACADAT LSQITNNIDP VGRIQMRTRR TLRGHLAKIY AMHWGTDSR LLVSASQDGK LIIWDSYTTN KVHAIPLRSS WVMTCAYAPS GNYVACGGLD NICSIYNLKT REGNVRVSRE L AGHTGYLS ...文字列: HHHHHHHHHH LEVLFQGPGS SGSELDQLRQ EAEQLKNQIR DARKACADAT LSQITNNIDP VGRIQMRTRR TLRGHLAKIY AMHWGTDSR LLVSASQDGK LIIWDSYTTN KVHAIPLRSS WVMTCAYAPS GNYVACGGLD NICSIYNLKT REGNVRVSRE L AGHTGYLS CCRFLDDNQI VTSSGDTTCA LWDIETGQQT TTFTGHTGDV MSLSLAPDTR LFVSGACDAS AKLWDVREGM CR QTFTGHE SDINAICFFP NGNAFATGSD DATCRLFDLR ADQELMTYSH DNIICGITSV SFSKSGRLLL AGYDDFNCNV WDA LKADRA GVLAGHDNRV SCLGVTDDGM AVATGSWDSF LKIWNGASGA SGASGASVSG WRLFKKIS UniProtKB: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1 |
-分子 #3: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2
| 分子 | 名称: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 7.861143 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASNNTASIA QARKLVEQLK MEANIDRIKV SKAAADLMAY CEAHAKEDPL LTPVPASENP FREKKFFCAI L UniProtKB: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2 |
-分子 #4: Soluble cytochrome b562,C-X-C chemokine receptor type 3,Oplophoru...
| 分子 | 名称: Soluble cytochrome b562,C-X-C chemokine receptor type 3,Oplophorus-luciferin 2-monooxygenase catalytic subunit タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO / EC番号: Oplophorus-luciferin 2-monooxygenase |
|---|---|
| 由来(天然) | 生物種:  Oplophorus gracilirostris (甲殻類) Oplophorus gracilirostris (甲殻類) |
| 分子量 | 理論値: 72.918008 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKTIIALSYI FCLVFADYKD DDDKGSADLE DNWETLNDNL KVIEKADNAA QVKDALTKMR AAALDAQKAT PPKLEDKSPD SPEMKDFRH GFDILVGQID DALKLANEGK VKEAQAAAEQ LKTTRNAYIQ KYLLVPRGSM VLEVSDHQVL NDAEVAALLE N FSSSYDYG ...文字列: MKTIIALSYI FCLVFADYKD DDDKGSADLE DNWETLNDNL KVIEKADNAA QVKDALTKMR AAALDAQKAT PPKLEDKSPD SPEMKDFRH GFDILVGQID DALKLANEGK VKEAQAAAEQ LKTTRNAYIQ KYLLVPRGSM VLEVSDHQVL NDAEVAALLE N FSSSYDYG ENESDSCCTS PPCPQDFSLN FDRAFLPALY SLLFLLGLLG NGAVAAVLLS RRTALSSTDT FLLHLAVADT LL VLTLPLW AVDAAVQWVF GSGLCKVAGA LFNINFYAGA LLLACISFDR YLNIVHATQL YRRGPPARVT LTCLAVWGLC LLF ALPDFI FLSAHHDERL NATHCQYNFP QVGRTALRVL QLVAGFLLPL LVMAYCYAHI LAVLLVSRGQ RRLRAMRLVV VVVV AFALC WTPYHLVVLV DILMDLGALA RNCGRESRVD VAKSVTSGLG YMHCCLNPLL YAFVGVKFRE RMWMLLLRLG CPNQR GLQR QPSSSRRDSS WSETSVFTLE DFVGDWEQTA AYNLDQVLEQ GGVSSLLQNL AVSVTPIQRI VRSGENALKI DIHVII PYE GLSADQMAQI EEVFKVVYPV DDHHFKVILP YGTLVIDGVT PNMLNYFGRP YEGIAVFDGK KITVTGTLWN GNKIIDE RL ITPDGSMLFR VTINS UniProtKB: Soluble cytochrome b562, C-X-C chemokine receptor type 3, Oplophorus-luciferin 2-monooxygenase catalytic subunit |
-分子 #5: single-chain variable fragment scFv16
| 分子 | 名称: single-chain variable fragment scFv16 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.784896 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DVQLVESGGG LVQPGGSRKL SCSASGFAFS SFGMHWVRQA PEKGLEWVAY ISSGSGTIYY ADTVKGRFTI SRDDPKNTLF LQMTSLRSE DTAMYYCVRS IYYYGSSPFD FWGQGTTLTV SSGGGGSGGG GSGGGGSDIV MTQATSSVPV TPGESVSISC R SSKSLLHS ...文字列: DVQLVESGGG LVQPGGSRKL SCSASGFAFS SFGMHWVRQA PEKGLEWVAY ISSGSGTIYY ADTVKGRFTI SRDDPKNTLF LQMTSLRSE DTAMYYCVRS IYYYGSSPFD FWGQGTTLTV SSGGGGSGGG GSGGGGSDIV MTQATSSVPV TPGESVSISC R SSKSLLHS NGNTYLYWFL QRPGQSPQLL IYRMSNLASG VPDRFSGSGS GTAFTLTISR LEAEDVGVYY CMQHLEYPLT FG AGTKLEL KAAAHHHHHH HH |
-分子 #6: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 6 / コピー数: 2 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #7: [4-(2-bromophenyl)phenyl]methyl-[[(1R,5S)-6,6-dimethyl-2-bicyclo[...
| 分子 | 名称: [4-(2-bromophenyl)phenyl]methyl-[[(1R,5S)-6,6-dimethyl-2-bicyclo[3.1.1]hept-2-enyl]methyl]-dimethyl-azanium タイプ: ligand / ID: 7 / コピー数: 1 / 式: 4IE |
|---|---|
| 分子量 | 理論値: 425.424 Da |
| Chemical component information | 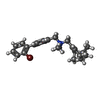 ChemComp-4IE: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 54.31 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.94 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 162856 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー












































 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)