+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Streptococcus thermophilus Cas1-Cas2-DnaQ-prespacer complex, conformation2 | |||||||||
 マップデータ マップデータ | Streptococcus thermophilus Cas1-Cas2-DnaQ-prespacer complex, conformation2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas / adaptation / IMMUNE SYSTEM | |||||||||
| 生物種 |  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.12 Å | |||||||||
 データ登録者 データ登録者 | Chen Q / Yu Y / Luo Y | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Innovation (Camb) / 年: 2023 ジャーナル: Innovation (Camb) / 年: 2023タイトル: DnaQ mediates directional spacer acquisition in the CRISPR-Cas system by a time-dependent mechanism. 著者: Dongmei Tang / Tingting Jia / Yongbo Luo / Biqin Mou / Jie Cheng / Shiqian Qi / Shaohua Yao / Zhaoming Su / Yamei Yu / Qiang Chen /  要旨: In the spacer acquisition stage of CRISPR-Cas immunity, spacer orientation and protospacer adjacent motif (PAM) removal are two prerequisites for functional spacer integration. Cas4 has been ...In the spacer acquisition stage of CRISPR-Cas immunity, spacer orientation and protospacer adjacent motif (PAM) removal are two prerequisites for functional spacer integration. Cas4 has been implicated in both processing the prespacer and determining the spacer orientation. In Cas4-lacking systems, host 3'-5' DnaQ family exonucleases were recently reported to play a Cas4-like role. However, the molecular details of DnaQ functions remain elusive. Here, we characterized the spacer acquisition of the adaptation module of the type I-E system, in which a DnaQ domain naturally fuses with Cas2. We presented X-ray crystal structures and cryo-electron microscopy structures of this adaptation module. Our biochemical data showed that DnaQ trimmed PAM-containing and PAM-deficient overhangs with different efficiencies. Based on these results, we proposed a time-dependent model for DnaQ-mediated spacer acquisition to elucidate PAM removal and spacer orientation determination in Cas4-lacking CRISPR-Cas systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34811.map.gz emd_34811.map.gz | 40.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34811-v30.xml emd-34811-v30.xml emd-34811.xml emd-34811.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34811.png emd_34811.png | 24.8 KB | ||
| その他 |  emd_34811_half_map_1.map.gz emd_34811_half_map_1.map.gz emd_34811_half_map_2.map.gz emd_34811_half_map_2.map.gz | 39.7 MB 39.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34811 http://ftp.pdbj.org/pub/emdb/structures/EMD-34811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34811 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34811_validation.pdf.gz emd_34811_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34811_full_validation.pdf.gz emd_34811_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_34811_validation.xml.gz emd_34811_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_34811_validation.cif.gz emd_34811_validation.cif.gz | 13.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34811 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34811 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34811 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34811 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34811.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34811.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Streptococcus thermophilus Cas1-Cas2-DnaQ-prespacer complex, conformation2 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: the first half map of conformation2
| ファイル | emd_34811_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | the first half map of conformation2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
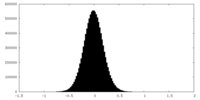
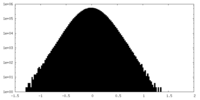
| 密度ヒストグラム |
-ハーフマップ: the second half map of conformation2
| ファイル | emd_34811_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | the second half map of conformation2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
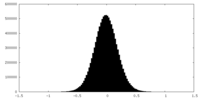
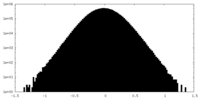
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cas1-Cas2-DnaQ-prespacer complex
| 全体 | 名称: Cas1-Cas2-DnaQ-prespacer complex |
|---|---|
| 要素 |
|
-超分子 #1: Cas1-Cas2-DnaQ-prespacer complex
| 超分子 | 名称: Cas1-Cas2-DnaQ-prespacer complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
-分子 #1: Cas1
| 分子 | 名称: Cas1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GASGSMVEKN GAKKTSLREL PKISDRVSFI YVEHAKINRV DSAITVLDSR GTVRIPAAMI GVLLLGPGTD ISHRAVELIG DTGTSMVWVG ERGVRQYAHG RSLAHSTKFL EKQAKLVSNS RLRLAVARKM YQMRFPDEDV SAMTMQQLRG REGARVRRVY RLQSEKYQVS ...文字列: GASGSMVEKN GAKKTSLREL PKISDRVSFI YVEHAKINRV DSAITVLDSR GTVRIPAAMI GVLLLGPGTD ISHRAVELIG DTGTSMVWVG ERGVRQYAHG RSLAHSTKFL EKQAKLVSNS RLRLAVARKM YQMRFPDEDV SAMTMQQLRG REGARVRRVY RLQSEKYQVS WTKREYNPDD FEGGDIVNQA LSAANVALYG LVHSIVIALG ASPGLGFVHT GHDLSFIYDI ADLYKAELTI PLAFEIAANF TEIDDIGKIA RQKVRDSFVD GKLIVRIVQD IQYLFDLDDD EELLVDTLSL WDDKDMLVKH GVSYKEEL |
-分子 #2: Cas2-DnaQ
| 分子 | 名称: Cas2-DnaQ / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPFTVVTLKS VPPSLRGDLT KWMQEIAIGV YVGNFNSRIR EKLWNRIQAN VGEGEATISY YYRNEIGYQF DMINSQKSVV DFDGIPLVLI PNSKTSSENY PKLGYSNAAK SRKIKRYSSY RGPQANSLKP YVVIDIETDG LDEKKNTIIE IGAVKFNGQQ VEEFNALIKY ...文字列: MPFTVVTLKS VPPSLRGDLT KWMQEIAIGV YVGNFNSRIR EKLWNRIQAN VGEGEATISY YYRNEIGYQF DMINSQKSVV DFDGIPLVLI PNSKTSSENY PKLGYSNAAK SRKIKRYSSY RGPQANSLKP YVVIDIETDG LDEKKNTIIE IGAVKFNGQQ VEEFNALIKY EEKLPPTIFK LTGISKSLLD QEGRDLKEVL SEFLLFIGDL TLVGYNIHFD IQFINNKLNK FGLPLLINKT HDIMRYVKDE KLFLDNYQLQ TALKSYGIED SVPHRALKDA RLIYHLSTKV NKFLARMKEK S |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 65.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.465 µm / 最小 デフォーカス(公称値): 1.213 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.12 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 16289 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)