+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human SGLT2-MAP17 complex with Canagliflozin | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ion transport / Sodium transport / Sugar transport / Symport / Transport / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報low-affinity D-glucose:sodium symporter activity / Defective SLC5A2 causes renal glucosuria (GLYS1) / alpha-glucoside transport / alpha-glucoside transmembrane transporter activity / D-glucose:sodium symporter activity / renal D-glucose absorption / hexose transmembrane transport / D-glucose import across plasma membrane / Cellular hexose transport / D-glucose transmembrane transporter activity ...low-affinity D-glucose:sodium symporter activity / Defective SLC5A2 causes renal glucosuria (GLYS1) / alpha-glucoside transport / alpha-glucoside transmembrane transporter activity / D-glucose:sodium symporter activity / renal D-glucose absorption / hexose transmembrane transport / D-glucose import across plasma membrane / Cellular hexose transport / D-glucose transmembrane transporter activity / sodium ion import across plasma membrane / sodium ion transport / carbohydrate metabolic process / apical plasma membrane / extracellular exosome / metal ion binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
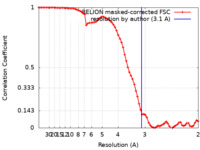
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Hiraizumi M / Kishida H / Miyaguchi I / Nureki O | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Transport and inhibition mechanism of the human SGLT2-MAP17 glucose transporter. 著者: Masahiro Hiraizumi / Tomoya Akashi / Kouta Murasaki / Hiroyuki Kishida / Taichi Kumanomidou / Nao Torimoto / Osamu Nureki / Ikuko Miyaguchi /  要旨: Sodium-glucose cotransporter 2 (SGLT2) is imporant in glucose reabsorption. SGLT2 inhibitors suppress renal glucose reabsorption, therefore reducing blood glucose levels in patients with type 2 ...Sodium-glucose cotransporter 2 (SGLT2) is imporant in glucose reabsorption. SGLT2 inhibitors suppress renal glucose reabsorption, therefore reducing blood glucose levels in patients with type 2 diabetes. We and others have developed several SGLT2 inhibitors starting from phlorizin, a natural product. Using cryo-electron microscopy, we present the structures of human (h)SGLT2-MAP17 complexed with five natural or synthetic inhibitors. The four synthetic inhibitors (including canagliflozin) bind the transporter in the outward conformations, while phlorizin binds it in the inward conformation. The phlorizin-hSGLT2 interaction exhibits biphasic kinetics, suggesting that phlorizin alternately binds to the extracellular and intracellular sides. The Na-bound outward-facing and unbound inward-open structures of hSGLT2-MAP17 suggest that the MAP17-associated bundle domain functions as a scaffold, with the hash domain rotating around the Na-binding site. Thus, Na binding stabilizes the outward-facing conformation, and its release promotes state transition to inward-open conformation, exhibiting a role of Na in symport mechanism. These results provide structural evidence for the Na-coupled alternating-access mechanism proposed for the transporter family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34673.map.gz emd_34673.map.gz | 1.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34673-v30.xml emd-34673-v30.xml emd-34673.xml emd-34673.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34673_fsc.xml emd_34673_fsc.xml | 6.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34673.png emd_34673.png | 169.9 KB | ||
| マスクデータ |  emd_34673_msk_1.map emd_34673_msk_1.map | 6.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34673.cif.gz emd-34673.cif.gz | 6.2 KB | ||
| その他 |  emd_34673_half_map_1.map.gz emd_34673_half_map_1.map.gz emd_34673_half_map_2.map.gz emd_34673_half_map_2.map.gz | 6.1 MB 6.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34673 http://ftp.pdbj.org/pub/emdb/structures/EMD-34673 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34673 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34673 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34673_validation.pdf.gz emd_34673_validation.pdf.gz | 772.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34673_full_validation.pdf.gz emd_34673_full_validation.pdf.gz | 772.1 KB | 表示 | |
| XML形式データ |  emd_34673_validation.xml.gz emd_34673_validation.xml.gz | 10.4 KB | 表示 | |
| CIF形式データ |  emd_34673_validation.cif.gz emd_34673_validation.cif.gz | 14 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34673 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34673 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34673 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34673 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34673.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34673.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34673_msk_1.map emd_34673_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34673_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34673_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Binary complex of Sodium/glucose cotransporter 2 with PDZK1-inter...
| 全体 | 名称: Binary complex of Sodium/glucose cotransporter 2 with PDZK1-interacting protein 1. |
|---|---|
| 要素 |
|
-超分子 #1: Binary complex of Sodium/glucose cotransporter 2 with PDZK1-inter...
| 超分子 | 名称: Binary complex of Sodium/glucose cotransporter 2 with PDZK1-interacting protein 1. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Electrogenic Na+-coupled sugar simporter that actively transports D-glucose at the plasma membrane, with a Na+ to sugar coupling ratio of 1:1. Transporter activity is driven by a ...詳細: Electrogenic Na+-coupled sugar simporter that actively transports D-glucose at the plasma membrane, with a Na+ to sugar coupling ratio of 1:1. Transporter activity is driven by a transmembrane Na+ electrochemical gradient set by the Na+/K+ pump |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Sodium/glucose cotransporter 2
| 分子 | 名称: Sodium/glucose cotransporter 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 73.247703 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GPGSMEEHTE AGSAPEMGAQ KALIDNPADI LVIAAYFLLV IGVGLWSMCR TNRGTVGGYF LAGRSMVWWP VGASLFASNI GSGHFVGLA GTGAASGLAV AGFEWNALFV VLLLGWLFAP VYLTAGVITM PQYLRKRFGG RRIRLYLSVL SLFLYIFTKI S VDMFSGAV ...文字列: GPGSMEEHTE AGSAPEMGAQ KALIDNPADI LVIAAYFLLV IGVGLWSMCR TNRGTVGGYF LAGRSMVWWP VGASLFASNI GSGHFVGLA GTGAASGLAV AGFEWNALFV VLLLGWLFAP VYLTAGVITM PQYLRKRFGG RRIRLYLSVL SLFLYIFTKI S VDMFSGAV FIQQALGWNI YASVIALLGI TMIYTVTGGL AALMYTDTVQ TFVILGGACI LMGYAFHEVG GYSGLFDKYL GA ATSLTVS EDPAVGNISS FCYRPRPDSY HLLRHPVTGD LPWPALLLGL TIVSGWYWCS DQVIVQRCLA GKSLTHIKAG CIL CGYLKL TPMFLMVMPG MISRILYPDE VACVVPEVCR RVCGTEVGCS NIAYPRLVVK LMPNGLRGLM LAVMLAALMS SLAS IFNSS STLFTMDIYT RLRPRAGDRE LLLVGRLWVV FIVVVSVAWL PVVQAAQGGQ LFDYIQAVSS YLAPPVSAVF VLALF VPRV NEQGAFWGLI GGLLMGLARL IPEFSFGSGS CVQPSACPAF LCGVHYLYFA IVLFFCSGLL TLTVSLCTAP IPRKHL HRL VFSLRHSKEE REDLDADEQQ GSSLPVQNGC PESAMEMNEP QAPAPSLFRQ CLLWFCGMSR GGVGSPPPLT QEEAAAA AR RLEDISEDPS WARVVNLNAL LMMAVAVFLW GFYA UniProtKB: Sodium/glucose cotransporter 2 |
-分子 #2: PDZK1-interacting protein 1
| 分子 | 名称: PDZK1-interacting protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.235 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSALSLLILG LLTAVPPASC QQGLGNLQPW MQGLIAVAVF LVLVAIAFAV NHFWCQEEPE PAHMILTVGN KADGVLVGTD GRYSSMAAS FRSSEHENAY ENVPEEEGKV RSTPM UniProtKB: PDZK1-interacting protein 1 |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 1 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: (2~{S},3~{R},4~{R},5~{S},6~{R})-2-[3-[[5-(4-fluorophenyl)thiophen...
| 分子 | 名称: (2~{S},3~{R},4~{R},5~{S},6~{R})-2-[3-[[5-(4-fluorophenyl)thiophen-2-yl]methyl]-4-methyl-phenyl]-6-(hydroxymethyl)oxane-3,4,5-triol タイプ: ligand / ID: 4 / コピー数: 1 / 式: L3R |
|---|---|
| 分子量 | 理論値: 444.516 Da |
| Chemical component information | 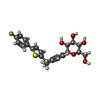 ChemComp-L3R: |
-分子 #5: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 5 / コピー数: 1 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 4 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 64.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)