+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of SARS-CoV-2 RNA-dependent RNA polymerase with gossypol binding | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | SARS-CoV-2 / RdRp / Inhibitor / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral genome replication / methyltransferase activity / protein guanylyltransferase activity / RNA endonuclease activity producing 3'-phosphomonoesters, hydrolytic mechanism / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / Maturation of replicase proteins ...viral genome replication / methyltransferase activity / protein guanylyltransferase activity / RNA endonuclease activity producing 3'-phosphomonoesters, hydrolytic mechanism / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / Maturation of replicase proteins / ISG15-specific peptidase activity / TRAF3-dependent IRF activation pathway / Transcription of SARS-CoV-2 sgRNAs / Translation of Replicase and Assembly of the Replication Transcription Complex / snRNP Assembly / Replication of the SARS-CoV-2 genome / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ / double membrane vesicle viral factory outer membrane / host cell endoplasmic reticulum-Golgi intermediate compartment / SARS coronavirus main proteinase / 5'-3' DNA helicase activity / 3'-5'-RNA exonuclease activity / host cell endosome / symbiont-mediated degradation of host mRNA / mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / omega peptidase activity / endonuclease activity / mRNA (guanine-N7)-methyltransferase / SARS-CoV-2 modulates host translation machinery / methyltransferase cap1 / host cell Golgi apparatus / methylation / symbiont-mediated suppression of host NF-kappaB cascade / symbiont-mediated perturbation of host ubiquitin-like protein modification / DNA helicase / methyltransferase cap1 activity / ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ / single-stranded RNA binding / host cell perinuclear region of cytoplasm / regulation of autophagy / viral protein processing / lyase activity / host cell endoplasmic reticulum membrane / RNA helicase / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / symbiont-mediated suppression of host gene expression / copper ion binding / viral translational frameshifting / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / lipid binding / DNA-templated transcription / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell nucleus / ATP hydrolysis activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | ||||||||||||
 データ登録者 データ登録者 | Wang W / Ren M / Li F | ||||||||||||
| 資金援助 | 3件
| ||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of SARS-CoV-2 RNA-dependent RNA polymerase with gossypol binding 著者: Wang W / Li W / Ren M / Li F | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34370.map.gz emd_34370.map.gz | 2.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34370-v30.xml emd-34370-v30.xml emd-34370.xml emd-34370.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34370.png emd_34370.png | 93.5 KB | ||
| Filedesc metadata |  emd-34370.cif.gz emd-34370.cif.gz | 5.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34370 http://ftp.pdbj.org/pub/emdb/structures/EMD-34370 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34370 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34370 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34370_validation.pdf.gz emd_34370_validation.pdf.gz | 311.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34370_full_validation.pdf.gz emd_34370_full_validation.pdf.gz | 311.1 KB | 表示 | |
| XML形式データ |  emd_34370_validation.xml.gz emd_34370_validation.xml.gz | 5.3 KB | 表示 | |
| CIF形式データ |  emd_34370_validation.cif.gz emd_34370_validation.cif.gz | 5.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34370 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34370 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34370 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34370 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gy6MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34370.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34370.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
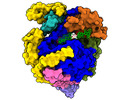
-全体 : SARS-CoV-2 Nsp12-Nsp7-Nsp8 complex bound to gossypol
| 全体 | 名称: SARS-CoV-2 Nsp12-Nsp7-Nsp8 complex bound to gossypol |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 Nsp12-Nsp7-Nsp8 complex bound to gossypol
| 超分子 | 名称: SARS-CoV-2 Nsp12-Nsp7-Nsp8 complex bound to gossypol タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: RNA-directed RNA polymerase
| 分子 | 名称: RNA-directed RNA polymerase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 106.780977 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SADAQSFLNR VCGVSAARLT PCGTGTSTDV VYRAFDIYND KVAGFAKFLK TNCCRFQEKD EDDNLIDSYF VVKRHTFSNY QHEETIYNL LKDCPAVAKH DFFKFRIDGD MVPHISRQRL TKYTMADLVY ALRHFDEGNC DTLKEILVTY NCCDDDYFNK K DWYDFVEN ...文字列: SADAQSFLNR VCGVSAARLT PCGTGTSTDV VYRAFDIYND KVAGFAKFLK TNCCRFQEKD EDDNLIDSYF VVKRHTFSNY QHEETIYNL LKDCPAVAKH DFFKFRIDGD MVPHISRQRL TKYTMADLVY ALRHFDEGNC DTLKEILVTY NCCDDDYFNK K DWYDFVEN PDILRVYANL GERVRQALLK TVQFCDAMRN AGIVGVLTLD NQDLNGNWYD FGDFIQTTPG SGVPVVDSYY SL LMPILTL TRALTAESHV DTDLTKPYIK WDLLKYDFTE ERLKLFDRYF KYWDQTYHPN CVNCLDDRCI LHCANFNVLF STV FPPTSF GPLVRKIFVD GVPFVVSTGY HFRELGVVHN QDVNLHSSRL SFKELLVYAA DPAMHAASGN LLLDKRTTCF SVAA LTNNV AFQTVKPGNF NKDFYDFAVS KGFFKEGSSV ELKHFFFAQD GNAAISDYDY YRYNLPTMCD IRQLLFVVEV VDKYF DCYD GGCINANQVI VNNLDKSAGF PFNKWGKARL YYDSMSYEDQ DALFAYTKRN VIPTITQMNL KYAISAKNRA RTVAGV SIC STMTNRQFHQ KLLKSIAATR GATVVIGTSK FYGGWHNMLK TVYSDVENPH LMGWDYPKCD RAMPNMLRIM ASLVLAR KH TTCCSLSHRF YRLANECAQV LSEMVMCGGS LYVKPGGTSS GDATTAYANS VFNICQAVTA NVNALLSTDG NKIADKYV R NLQHRLYECL YRNRDVDTDF VNEFYAYLRK HFSMMILSDD AVVCFNSTYA SQGLVASIKN FKSVLYYQNN VFMSEAKCW TETDLTKGPH EFCSQHTMLV KQGDDYVYLP YPDPSRILGA GCFVDDIVKT DGTLMIERFV SLAIDAYPLT KHPNQEYADV FHLYLQYIR KLHDELTGHM LDMYSVMLTN DNTSRYWEPE FYEAMYTPHT VLQ UniProtKB: Replicase polyprotein 1ab |
-分子 #2: Non-structural protein 8
| 分子 | 名称: Non-structural protein 8 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.903047 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AIASEFSSLP SYAAFATAQE AYEQAVANGD SEVVLKKLKK SLNVAKSEFD RDAAMQRKLE KMADQAMTQM YKQARSEDKR AKVTSAMQT MLFTMLRKLD NDALNNIINN ARDGCVPLNI IPLTTAAKLM VVIPDYNTYK NTCDGTTFTY ASALWEIQQV V DADSKIVQ ...文字列: AIASEFSSLP SYAAFATAQE AYEQAVANGD SEVVLKKLKK SLNVAKSEFD RDAAMQRKLE KMADQAMTQM YKQARSEDKR AKVTSAMQT MLFTMLRKLD NDALNNIINN ARDGCVPLNI IPLTTAAKLM VVIPDYNTYK NTCDGTTFTY ASALWEIQQV V DADSKIVQ LSEISMDNSP NLAWPLIVTA LRANSAVKLQ UniProtKB: Replicase polyprotein 1ab |
-分子 #3: Non-structural protein 7
| 分子 | 名称: Non-structural protein 7 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.248804 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SKMSDVKCTS VVLLSVLQQL RVESSSKLWA QCVQLHNDIL LAKDTTEAFE KMVSLLSVLL SMQGAVDINK LCEEMLDNRA TLQ UniProtKB: Replicase polyprotein 1a |
-分子 #4: Gossypol
| 分子 | 名称: Gossypol / タイプ: ligand / ID: 4 / コピー数: 2 / 式: GO3 |
|---|---|
| 分子量 | 理論値: 518.554 Da |
| Chemical component information |  ChemComp-GO3: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 使用した粒子像数: 1224147 |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)
















