+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | TLR3-mAb12-poly(I:C) | |||||||||
 マップデータ マップデータ | TLR3-mAb12-poly(I:C) | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 13.2 Å | |||||||||
 データ登録者 データ登録者 | Lim CS / Jang YH / Lee GY / Han GM / Lee JO | |||||||||
| 資金援助 |  韓国, 2件 韓国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: TLR3 forms a highly organized cluster when bound to a poly(I:C) RNA ligand. 著者: Chan Seok Lim / Yoon Ha Jang / Ga Young Lee / Gu Min Han / Hye Jin Jeong / Ji Won Kim / Jie-Oh Lee /  要旨: Toll-like Receptor 3 (TLR3) initiates a potent anti-viral immune response by binding to double-stranded RNA ligands. Previous crystallographic studies showed that TLR3 forms a homodimer when bound to ...Toll-like Receptor 3 (TLR3) initiates a potent anti-viral immune response by binding to double-stranded RNA ligands. Previous crystallographic studies showed that TLR3 forms a homodimer when bound to a 46-base pair RNA ligand. However, this short RNA fails to initiate a robust immune response. To obtain structural insights into the length dependency of TLR3 ligands, we determine the cryo-electron microscopy structure of full-length TLR3 in a complex with a synthetic RNA ligand with an average length of ~400 base pairs. In the structure, the dimeric TLR3 units are clustered along the double-stranded RNA helix in a highly organized and cooperative fashion with a uniform inter-dimer spacing of 103 angstroms. The intracellular and transmembrane domains are dispensable for the clustering because their deletion does not interfere with the cluster formation. Our structural observation suggests that ligand-induced clustering of TLR3 dimers triggers the ordered assembly of intracellular signaling adaptors and initiates a robust innate immune response. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34367.map.gz emd_34367.map.gz | 229.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34367-v30.xml emd-34367-v30.xml emd-34367.xml emd-34367.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34367.png emd_34367.png | 29.1 KB | ||
| その他 |  emd_34367_half_map_1.map.gz emd_34367_half_map_1.map.gz emd_34367_half_map_2.map.gz emd_34367_half_map_2.map.gz | 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34367 http://ftp.pdbj.org/pub/emdb/structures/EMD-34367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34367 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34367_validation.pdf.gz emd_34367_validation.pdf.gz | 686.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34367_full_validation.pdf.gz emd_34367_full_validation.pdf.gz | 686 KB | 表示 | |
| XML形式データ |  emd_34367_validation.xml.gz emd_34367_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_34367_validation.cif.gz emd_34367_validation.cif.gz | 18.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34367 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34367 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34367.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34367.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TLR3-mAb12-poly(I:C) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.831 Å | ||||||||||||||||||||||||||||||||||||
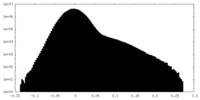
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: low-pass filtered (> 7 angstrom)
| ファイル | emd_34367_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | low-pass filtered (> 7 angstrom) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
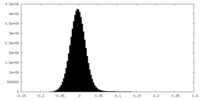
| 密度ヒストグラム |
-ハーフマップ: low-pass filtered (> 7 angstrom)
| ファイル | emd_34367_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | low-pass filtered (> 7 angstrom) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
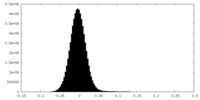
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TLR3-mAb12-poly(I:C)
| 全体 | 名称: TLR3-mAb12-poly(I:C) |
|---|---|
| 要素 |
|
-超分子 #1: TLR3-mAb12-poly(I:C)
| 超分子 | 名称: TLR3-mAb12-poly(I:C) / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / 詳細: Fab form of mAb12 bound to TLR3-poly(I:C) complex |
|---|
-超分子 #2: TLR3
| 超分子 | 名称: TLR3 / タイプ: complex / キメラ: Yes / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: mAb12
| 超分子 | 名称: mAb12 / タイプ: complex / キメラ: Yes / ID: 3 / 親要素: 1 / 含まれる分子: #2 / 詳細: Fab form |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #4: poly(I:C)
| 超分子 | 名称: poly(I:C) / タイプ: complex / キメラ: Yes / ID: 4 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.26 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 5.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 13.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.2) / 使用した粒子像数: 6864 |
|---|---|
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: OTHER |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)