+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Lloviu cuevavirus nucleoprotein-RNA complex | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | nucleoprotein / VIRAL PROTEIN | |||||||||||||||||||||
| 機能・相同性 | Ebola nucleoprotein / Ebola nucleoprotein / viral RNA genome packaging / helical viral capsid / viral nucleocapsid / host cell cytoplasm / ribonucleoprotein complex / Nucleoprotein 機能・相同性情報 機能・相同性情報 | |||||||||||||||||||||
| 生物種 |  Lloviu cuevavirus (ウイルス) / synthetic construct (人工物) Lloviu cuevavirus (ウイルス) / synthetic construct (人工物) | |||||||||||||||||||||
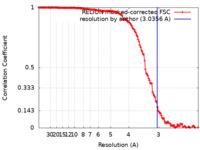
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0356 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Hu SF / Fujita-Fujiharu Y / Sugita Y / Wendt L / Muramoto Y / Nakano M / Hoenen T / Noda T | |||||||||||||||||||||
| 資金援助 |  日本, 6件 日本, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: PNAS Nexus / 年: 2023 ジャーナル: PNAS Nexus / 年: 2023タイトル: Cryoelectron microscopic structure of the nucleoprotein-RNA complex of the European filovirus, Lloviu virus. 著者: Shangfan Hu / Yoko Fujita-Fujiharu / Yukihiko Sugita / Lisa Wendt / Yukiko Muramoto / Masahiro Nakano / Thomas Hoenen / Takeshi Noda /   要旨: Lloviu virus (LLOV) is a novel filovirus detected in Schreiber's bats in Europe. The isolation of the infectious LLOV from bats has raised public health concerns. However, the virological and ...Lloviu virus (LLOV) is a novel filovirus detected in Schreiber's bats in Europe. The isolation of the infectious LLOV from bats has raised public health concerns. However, the virological and molecular characteristics of LLOV remain largely unknown. The nucleoprotein (NP) of LLOV encapsidates the viral genomic RNA to form a helical NP-RNA complex, which acts as a scaffold for nucleocapsid formation and de novo viral RNA synthesis. In this study, using single-particle cryoelectron microscopy, we determined two structures of the LLOV NP-RNA helical complex, comprising a full-length and a C-terminally truncated NP. The two helical structures were identical, demonstrating that the N-terminal region determines the helical arrangement of the NP. The LLOV NP-RNA protomers displayed a structure similar to that in the Ebola and Marburg virus, but the spatial arrangements in the helix differed. Structure-based mutational analysis identified amino acids involved in the helical assembly and viral RNA synthesis. These structures advance our understanding of the filovirus nucleocapsid formation and provide a structural basis for the development of antifiloviral therapeutics. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34016.map.gz emd_34016.map.gz | 228.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34016-v30.xml emd-34016-v30.xml emd-34016.xml emd-34016.xml | 21.5 KB 21.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34016_fsc.xml emd_34016_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34016.png emd_34016.png | 234.4 KB | ||
| Filedesc metadata |  emd-34016.cif.gz emd-34016.cif.gz | 7 KB | ||
| その他 |  emd_34016_half_map_1.map.gz emd_34016_half_map_1.map.gz emd_34016_half_map_2.map.gz emd_34016_half_map_2.map.gz | 194 MB 193.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34016 http://ftp.pdbj.org/pub/emdb/structures/EMD-34016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34016 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34016_validation.pdf.gz emd_34016_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34016_full_validation.pdf.gz emd_34016_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_34016_validation.xml.gz emd_34016_validation.xml.gz | 21.7 KB | 表示 | |
| CIF形式データ |  emd_34016_validation.cif.gz emd_34016_validation.cif.gz | 28.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34016 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34016 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34016 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34016 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ypwMC  7yr8C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34016.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34016.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.13 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34016_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
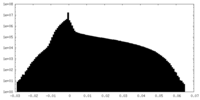
| 投影像・断面図 |
| ||||||||||||
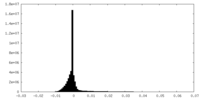
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34016_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Lloviu cuevavirus nucleoprotein RNA complex
| 全体 | 名称: Lloviu cuevavirus nucleoprotein RNA complex |
|---|---|
| 要素 |
|
-超分子 #1: Lloviu cuevavirus nucleoprotein RNA complex
| 超分子 | 名称: Lloviu cuevavirus nucleoprotein RNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Lloviu cuevavirus (ウイルス) Lloviu cuevavirus (ウイルス) |
-分子 #1: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lloviu cuevavirus (ウイルス) / 株: isolate Bat/Spain/Asturias-Bat86/2003 Lloviu cuevavirus (ウイルス) / 株: isolate Bat/Spain/Asturias-Bat86/2003 |
| 分子量 | 理論値: 84.553094 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MNRYLGHGTR TSRENTNLSE LHGILSLGLN VDHTIVRKKS IPLFEIGNSD QVCNWIIQII EAGVDLQEVA DSFLTMLCVN HAYQGDPNL FLESPAAHYL KGHGIHFEIQ HRDNVDHITD LLGVGSRDKS LRKTLSALEF EPGGTTTAGM FLSFASLFLP K LVVGERAC ...文字列: MNRYLGHGTR TSRENTNLSE LHGILSLGLN VDHTIVRKKS IPLFEIGNSD QVCNWIIQII EAGVDLQEVA DSFLTMLCVN HAYQGDPNL FLESPAAHYL KGHGIHFEIQ HRDNVDHITD LLGVGSRDKS LRKTLSALEF EPGGTTTAGM FLSFASLFLP K LVVGERAC LEKVQRQIQI HAEQGLIQYP TQWQSVGHMM VVFRLIRVNF VLKFLLVHQG MHMMAGHDAN DAIIANSISQ TR FSGLLIV KTVLEHILQK TEAGVQLHPL ARTSKVKGEL LAFKSALEAL ASHREYAPFA RLLNLSGVNN LEHGLYPQLS AIA LGVATA HGSTLAGVNV SEQYQQLREA ATEAEKQLQQ HSEMRELETL GLDEQERKIL ATFHSRKNEI NIQQTSSILA IRKE RLRKL TEALTEEKNK NALDDEDESE EDDWSPENRG IRSNKGSTKE PSSYTASRTE EDRNNYNSKD DHLSGKEQMS TQQES GADD LDLFDLDDDG DTNSQDPNNR QKQSDTQQTQ ESSDRSDYSR RPAYDWPPGD RPHTTQATDE HTDLLNKDHR RNQVKP GRR GNDPRTLPLI SFDDNEGEIL DDKSDLPAPD THSDPDTEES EEEHPDEELL PPAPKYSTKT SEQEPGDWKQ PTSPLST IP EEEGGHEANN DNSESDLIDQ MYQHIFKTEG AYAAINYYYK TTGRPVTFTS NNNHDYTFPQ DTEGLFPPWE GKENQKVA E ILTNSLHETG QEWADMSAKE RYLFLINN UniProtKB: Nucleoprotein |
-分子 #2: RNA (5'-R(P*UP*UP*UP*UP*UP*U)-3')
| 分子 | 名称: RNA (5'-R(P*UP*UP*UP*UP*UP*U)-3') / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 1.792037 KDa |
| 配列 | 文字列: UUUUUU |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Apply 1.25 micro litters of sample from each side of the grid and blot for 7 seconds before plugging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: Microscope was modified with a Cs corrector with two hexapole elements |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 3122 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 59000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)