+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of CasPi with guide RNA and target DNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CasPi complex / RNA BINDING PROTEIN / RNA BINDING PROTEIN-DNA-RNA complex | |||||||||
| 機能・相同性 | Transposase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Armatimonadetes bacterium (バクテリア) / Armatimonadetes bacterium (バクテリア) /  Armatimonadota bacterium (バクテリア) Armatimonadota bacterium (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.36 Å | |||||||||
 データ登録者 データ登録者 | Li CP / Wang J / Liu JJ / Zhang S | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2023 ジャーナル: Cell Res / 年: 2023タイトル: The compact Casπ (Cas12l) 'bracelet' provides a unique structural platform for DNA manipulation. 著者: Ao Sun / Cheng-Ping Li / Zhihang Chen / Shouyue Zhang / Dan-Yuan Li / Yun Yang / Long-Qi Li / Yuqian Zhao / Kaichen Wang / Zhaofu Li / Jinxia Liu / Sitong Liu / Jia Wang / Jun-Jie Gogo Liu /  要旨: CRISPR-Cas modules serve as the adaptive nucleic acid immune systems for prokaryotes, and provide versatile tools for nucleic acid manipulation in various organisms. Here, we discovered a new ...CRISPR-Cas modules serve as the adaptive nucleic acid immune systems for prokaryotes, and provide versatile tools for nucleic acid manipulation in various organisms. Here, we discovered a new miniature type V system, CRISPR-Casπ (Cas12l) (~860 aa), from the environmental metagenome. Complexed with a large guide RNA (~170 nt) comprising the tracrRNA and crRNA, Casπ (Cas12l) recognizes a unique 5' C-rich PAM for DNA cleavage under a broad range of biochemical conditions, and generates gene editing in mammalian cells. Cryo-EM study reveals a 'bracelet' architecture of Casπ effector encircling the DNA target at 3.4 Å resolution, substantially different from the canonical 'two-lobe' architectures of Cas12 and Cas9 nucleases. The large guide RNA serves as a 'two-arm' scaffold for effector assembly. Our study expands the knowledge of DNA targeting mechanisms by CRISPR effectors, and offers an efficient but compact platform for DNA manipulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33983.map.gz emd_33983.map.gz | 49.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33983-v30.xml emd-33983-v30.xml emd-33983.xml emd-33983.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33983.png emd_33983.png | 106.5 KB | ||
| Filedesc metadata |  emd-33983.cif.gz emd-33983.cif.gz | 6.9 KB | ||
| その他 |  emd_33983_half_map_1.map.gz emd_33983_half_map_1.map.gz emd_33983_half_map_2.map.gz emd_33983_half_map_2.map.gz | 49 MB 49 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33983 http://ftp.pdbj.org/pub/emdb/structures/EMD-33983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33983 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33983_validation.pdf.gz emd_33983_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33983_full_validation.pdf.gz emd_33983_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_33983_validation.xml.gz emd_33983_validation.xml.gz | 11.9 KB | 表示 | |
| CIF形式データ |  emd_33983_validation.cif.gz emd_33983_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33983 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33983 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33983 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33983 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7yojMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33983.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33983.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.856 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33983_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
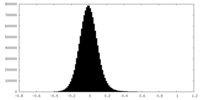
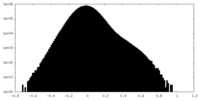
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33983_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
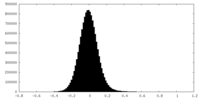
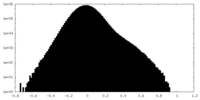
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CasPi
| 全体 | 名称: CasPi |
|---|---|
| 要素 |
|
-超分子 #1: CasPi
| 超分子 | 名称: CasPi / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Armatimonadetes bacterium (バクテリア) Armatimonadetes bacterium (バクテリア) |
| 分子量 | 理論値: 177.28 KDa |
-分子 #1: CasPi
| 分子 | 名称: CasPi / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Armatimonadota bacterium (バクテリア) Armatimonadota bacterium (バクテリア) |
| 分子量 | 理論値: 98.731234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKATKEVKS KRVEALRQVA YQRLERLERK AQKIGAHLRK PGKAADLQSL HYLLHKVEVE YHDIARNLEK DPTWTPKPKM RREKRAIVP ESGPAAPLPT TAKGEPGRPA NRHIPPPVPL DSARIPEDQQ SMGQGSGGRS WCSAPFVEVK LPPTQWSNVR E KLLKFRIE ...文字列: MAKATKEVKS KRVEALRQVA YQRLERLERK AQKIGAHLRK PGKAADLQSL HYLLHKVEVE YHDIARNLEK DPTWTPKPKM RREKRAIVP ESGPAAPLPT TAKGEPGRPA NRHIPPPVPL DSARIPEDQQ SMGQGSGGRS WCSAPFVEVK LPPTQWSNVR E KLLKFRIE DDADIVRRWA EAKFGSIETA RDGLRASAEI GTSPDVWRSF ISRAISNGKK DFEPLLSLDD DELTADATAE RV VRRWHQI DWVGRMLDSI LETVPSGVSK DTFRSRVESR LKTFHSSVNS FELKKRKDGT VERKRKHTNP QFPYLSPSAV SID PDVVTM EAVELLQMQP EERFAKDPND ANGRMRLRVL QAELGKARRE ALGRRGEKAP PWSGRKVFRG TTTRKREACL VWDK EAQAD GLYFALVMSG GPKIDDKRFV YMDGQPLQSD WQLHNGVAGK AKSCRAMPLI LKHDFLRWYH RHIKNHDVNA PLEKR CVHT TTQFVFVEPD EKKGLQPRLF IRPVFKFYDP VYEVPDSHSI DKKPDCRYLI GIARGVNYPY RAAVYDCETN SIIADK FVD GRKADWERIR NELAYHQRRR DLLRNSRASS AAIQREIRAI ARIRKRERGL NKVETVESIA RLVDWAEENL GKCNYCF VL ADLSSNLNLG RNNRVKHIAA IKEALINQMR KRGYRFKKSG KVDGVREESA WYTSAVAPSG WWAKKEEVDG AWKADKTR P LARKIGSYYC CEEIDGLHLR GVLKGLGRAK RLVLQSDDPS APTRRRGFGS ELFWDPYCTE LCGHAFPQGV VLDADFIGA FNIALRPLVR EELGKKAKAV DLADRHQTLN PTVALRCGVT AYEFVEVGGD PRGGLRKILL NPAEAVI UniProtKB: Transposase |
-分子 #2: DNA (5'-D(P*CP*GP*GP*GP*AP*TP*GP*CP*CP*CP*AP*G)-3')
| 分子 | 名称: DNA (5'-D(P*CP*GP*GP*GP*AP*TP*GP*CP*CP*CP*AP*G)-3') / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Armatimonadetes bacterium (バクテリア) Armatimonadetes bacterium (バクテリア) |
| 分子量 | 理論値: 3.688405 KDa |
| 配列 | 文字列: (DC)(DG)(DG)(DG)(DA)(DT)(DG)(DC)(DC)(DC) (DA)(DG) |
-分子 #3: DNA (30-MER)
| 分子 | 名称: DNA (30-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Armatimonadetes bacterium (バクテリア) Armatimonadetes bacterium (バクテリア) |
| 分子量 | 理論値: 9.192878 KDa |
| 配列 | 文字列: (DG)(DT)(DT)(DC)(DA)(DC)(DC)(DA)(DG)(DG) (DG)(DT)(DG)(DT)(DC)(DG)(DC)(DC)(DC)(DT) (DG)(DG)(DG)(DC)(DA)(DT)(DC)(DC)(DC) (DG) |
-分子 #4: RNA (174-MER)
| 分子 | 名称: RNA (174-MER) / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Armatimonadetes bacterium (バクテリア) Armatimonadetes bacterium (バクテリア) |
| 分子量 | 理論値: 56.411703 KDa |
| 配列 | 文字列: GUCUGCCGAA GACGCCGCAC GGAGCCUGGG CCGGAAUCGU AGAUCGAACG CGGCAUCGAA GCCCUGCAGC CCUUCGGGGC CAAGGCGGC GCAGCAAGCC UCUUUCAGGC GGCAGAGUCC UUUAGAGUGU GAGAGACACU CUAAAGGAAU GAAAGAGGGC G ACACCCUG GUGAAC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析 #1
画像解析 #1
+ 画像解析 #2
画像解析 #2
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE |
|---|---|
| 得られたモデル |  PDB-7yoj: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)