[English] 日本語
 Yorodumi
Yorodumi- EMDB-33680: CryoEM structure of type III-E CRISPR gRAMP-crRNA in complex with... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | CryoEM structure of type III-E CRISPR gRAMP-crRNA in complex with TPR-CHAT | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Nuclease / STRUCTURAL PROTEIN-RNA COMPLEX | |||||||||
| Function / homology | CHAT domain / CHAT domain / : / CRISPR type III-associated protein / RAMP superfamily / defense response to virus / RAMP superfamily protein / CHAT domain protein Function and homology information Function and homology information | |||||||||
| Biological species |  Candidatus Scalindua brodae (bacteria) Candidatus Scalindua brodae (bacteria) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.61 Å | |||||||||
 Authors Authors | Zhang JT / Cui N / Huang HD / Jia N | |||||||||
| Funding support | 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2022 Journal: Nat Commun / Year: 2022Title: Structural basis for the non-self RNA-activated protease activity of the type III-E CRISPR nuclease-protease Craspase. Authors: Ning Cui / Jun-Tao Zhang / Zhuolin Li / Xiao-Yu Liu / Chongyuan Wang / Hongda Huang / Ning Jia /  Abstract: The RNA-targeting type III-E CRISPR-gRAMP effector interacts with a caspase-like protease TPR-CHAT to form the CRISPR-guided caspase complex (Craspase), but their functional mechanism is unknown. ...The RNA-targeting type III-E CRISPR-gRAMP effector interacts with a caspase-like protease TPR-CHAT to form the CRISPR-guided caspase complex (Craspase), but their functional mechanism is unknown. Here, we report cryo-EM structures of the type III-E gRAMP and gRAMP-TPR-CHAT complexes, before and after either self or non-self RNA target binding, and elucidate the mechanisms underlying RNA-targeting and non-self RNA-induced protease activation. The associated TPR-CHAT adopted a distinct conformation upon self versus non-self RNA target binding, with nucleotides at positions -1 and -2 of the CRISPR-derived RNA (crRNA) serving as a sensor. Only binding of the non-self RNA target activated the TPR-CHAT protease, leading to cleavage of Csx30 protein. Furthermore, TPR-CHAT structurally resembled eukaryotic separase, but with a distinct mechanism for protease regulation. Our findings should facilitate the development of gRAMP-based RNA manipulation tools, and advance our understanding of the virus-host discrimination process governed by a nuclease-protease Craspase during type III-E CRISPR-Cas immunity. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_33680.map.gz emd_33680.map.gz | 59.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-33680-v30.xml emd-33680-v30.xml emd-33680.xml emd-33680.xml | 18.8 KB 18.8 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_33680.png emd_33680.png | 79.7 KB | ||
| Masks |  emd_33680_msk_1.map emd_33680_msk_1.map | 64 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-33680.cif.gz emd-33680.cif.gz | 7 KB | ||
| Others |  emd_33680_half_map_1.map.gz emd_33680_half_map_1.map.gz emd_33680_half_map_2.map.gz emd_33680_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33680 http://ftp.pdbj.org/pub/emdb/structures/EMD-33680 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33680 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33680 | HTTPS FTP |
-Related structure data
| Related structure data |  7y84MC  7y80C  7y81C  7y82C  7y83C  7y85C M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_33680.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_33680.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Mask #1
| File |  emd_33680_msk_1.map emd_33680_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
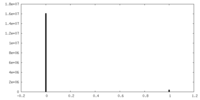
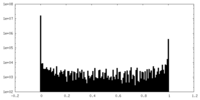
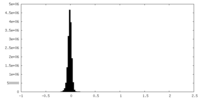
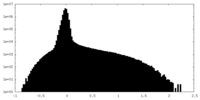
| Density Histograms |
-Half map: #2
| File | emd_33680_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_33680_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Type III-E CRISPR gRAMP-crRNA in complex with TPR-CHAT
| Entire | Name: Type III-E CRISPR gRAMP-crRNA in complex with TPR-CHAT |
|---|---|
| Components |
|
-Supramolecule #1: Type III-E CRISPR gRAMP-crRNA in complex with TPR-CHAT
| Supramolecule | Name: Type III-E CRISPR gRAMP-crRNA in complex with TPR-CHAT type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#3 |
|---|---|
| Source (natural) | Organism:  Candidatus Scalindua brodae (bacteria) Candidatus Scalindua brodae (bacteria) |
-Supramolecule #2: RAMP superfamily protein/CHAT
| Supramolecule | Name: RAMP superfamily protein/CHAT / type: complex / ID: 2 / Parent: 1 / Macromolecule list: #1, #3 |
|---|---|
| Source (natural) | Organism:  Candidatus Scalindua brodae (bacteria) Candidatus Scalindua brodae (bacteria) |
-Supramolecule #3: crRNA
| Supramolecule | Name: crRNA / type: complex / ID: 3 / Parent: 1 / Macromolecule list: #2 |
|---|
-Macromolecule #1: RAMP superfamily protein
| Macromolecule | Name: RAMP superfamily protein / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Candidatus Scalindua brodae (bacteria) Candidatus Scalindua brodae (bacteria) |
| Molecular weight | Theoretical: 198.652688 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MHHHHHHKSN DMNITVELTF FEPYRLVEWF DWDARKKSHS AMRGQAFAQW TWKGKGRTAG KSFITGTLVR SAVIKAVEEL LSLNNGKWE GVPCCNGSFQ TDESKGKKPS FLRKRHTLQW QANNKNICDK EEACPFCILL GRFDNAGKVH ERNKDYDIHF S NFDLDHKQ ...String: MHHHHHHKSN DMNITVELTF FEPYRLVEWF DWDARKKSHS AMRGQAFAQW TWKGKGRTAG KSFITGTLVR SAVIKAVEEL LSLNNGKWE GVPCCNGSFQ TDESKGKKPS FLRKRHTLQW QANNKNICDK EEACPFCILL GRFDNAGKVH ERNKDYDIHF S NFDLDHKQ EKNDLRLVDI ASGRILNRVD FDTGKAKDYF RTWEADYETY GTYTGRITLR NEHAKKLLLA SLGFVDKLCG AL CRIEVIK KSESPLPSDT KEQSYTKDDT VEVLSEDHND ELRKQAEVIV EAFKQNDKLE KIRILADAIR TLRLHGEGVI EKD ELPDGK EERDKGHHLW DIKVQGTALR TKLKELWQSN KDIGWRKFTE MLGSNLYLIY KKETGGVSTR FRILGDTEYY SKAH DSEGS DLFIPVTPPE GIETKEWIIV GRLKAATPFY FGVQQPSDSI PGKEKKSEDS LVINEHTSFN ILLDKENRYR IPRSA LRGA LRRDLRTAFG SGCNVSLGGQ ILCNCKVCIE MRRITLKDSV SDFSEPPEIR YRIAKNPGTA TVEDGSLFDI EVGPEG LTF PFVLRYRGHK FPEQLSSVIR YWEENDGKNG MAWLGGLDST GKGRFALKDI KIFEWDLNQK INEYIKERGM RGKEKEL LE MGESSLPDGL IPYKFFEERE CLFPYKENLK PQWSEVQYTI EVGSPLLTAD TISALTEPGN RDAIAYKKRV YNDGNNAI E PEPRFAVKSE THRGIFRTAV GRRTGDLGKE DHEDCTCDMC IIFGNEHESS KIRFEDLELI NGNEFEKLEK HIDHVAIDR FTGGALDKAK FDTYPLAGSP KKPLKLKGRF WIKKGFSGDH KLLITTALSD IRDGLYPLGS KGGVGYGWVA GISIDDNVPD DFKEMINKT EMPLPEEVEE SNNGPINNDY VHPGHQSPKQ DHKNKNIYYP HYFLDSGSKV YREKDIITHE EFTEELLSGK I NCKLETLT PLIIPDTSDE NGLKLQGNKP GHKNYKFFNI NGELMIPGSE LRGMLRTHFE ALTKSCFAIF GEDSTLSWRM NA DEKDYKI DSNSIRKMES QRNPKYRIPD ELQKELRNSG NGLFNRLYTS ERRFWSDVSN KFENSIDYKR EILRCAGRPK NYK GGIIRQ RKDSLMAEEL KVHRLPLYDN FDIPDSAYKA NDHCRKSATC STSRGCRERF TCGIKVRDKN RVFLNAANNN RQYL NNIKK SNHDLYLQYL KGEKKIRFNS KVITGSERSP IDVIAELNER GRQTGFIKLS GLNNSNKSQG NTGTTFNSGW DRFEL NILL DDLETRPSKS DYPRPRLLFT KDQYEYNITK RCERVFEIDK GNKTGYPVDD QIKKNYEDIL DSYDGIKDQE VAERFD TFT RGSKLKVGDL VYFHIDGDNK IDSLIPVRIS RKCASKTLGG KLDKALHPCT GLSDGLCPGC HLFGTTDYKG RVKFGFA KY ENGPEWLITR GNNPERSLTL GVLESPRPAF SIPDDESEIP GRKFYLHHNG WRIIRQKQLE IRETVQPERN VTTEVMDK G NVFSFDVRFE NLREWELGLL LQSLDPGKNI AHKLGKGKPY GFGSVKIKID SLHTFKINSN NDKIKRVPQS DIREYINKG YQKLIEWSGN NSIQKGNVLP QWHVIPHIDK LYKLLWVPFL NDSKLEPDVR YPVLNEESKG YIEGSDYTYK KLGDKDNLPY KTRVKGLTT PWSPWNPFQV IAEHEEQEVN VTGSRPSVTD KIERDGKMV UniProtKB: RAMP superfamily protein |
-Macromolecule #3: CHAT domain protein
| Macromolecule | Name: CHAT domain protein / type: protein_or_peptide / ID: 3 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Candidatus Scalindua brodae (bacteria) Candidatus Scalindua brodae (bacteria) |
| Molecular weight | Theoretical: 85.523141 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MNNTEENIDR IQEPTREDID RKEAERLLDE AFNPRTKPVD RKKIINSALK ILIGLYKEKK DDLTSASFIS IARAYYLVSI TILPKGTTI PEKKKEALRK GIEFIDRAIN KFNGSILDSQ RAFRIKSVLS IEFNRIDREK CDNIKLKNLL NEAVDKGCTD F DTYEWDIQ ...String: MNNTEENIDR IQEPTREDID RKEAERLLDE AFNPRTKPVD RKKIINSALK ILIGLYKEKK DDLTSASFIS IARAYYLVSI TILPKGTTI PEKKKEALRK GIEFIDRAIN KFNGSILDSQ RAFRIKSVLS IEFNRIDREK CDNIKLKNLL NEAVDKGCTD F DTYEWDIQ IAIRLCELGV DMEGHFDNLI KSNKANDLQK AKAYYFIKKD DHKAKEHMDK CTASLKYTPC SHRLWDETVG FI ERLKGDS STLWRDFAIK TYRSCRVQEK ETGTLRLRWY WSRHRVLYDM AFLAVKEQAD DEEPDVNVKQ AKIKKLAEIS DSL KSRFSL RLSDMEKMPK SDDESNHEFK KFLDKCVTAY QDGYVINRSE DKEGQGENKS TTSKQPEPRP QAKLLELTQV PEGW VVVHF YLNKLEGMGN AIVFDKCANS WQYKEFQYKE LFEVFLTWQA NYNLYKENAA EHLVTLCKKI GETMPFLFCD NFIPN GKDV LFVPHDFLHR LPLHGSIENK TNGKLFLENH SCCYLPAWSF ASEKEASTSD EYVLLKNFDQ GHFETLQNNQ IWGTQS VKD GASSDDLENI RNNPRLLTIL CHGEANMSNP FRSMLKLANG GITYLEILNS VKGLKGSQVI LGACETDLVP PLSDVMD EH YSVATALLLI GAAGVVGTMW KVRSNKTKSL IEWKLENIEY KLNEWQKETG GAAYKDHPPT FYRSIAFRSI GFPLGGSG W SHPQFEKGGG SGGGSGGWSH PQFEK UniProtKB: CHAT domain protein |
-Macromolecule #2: crRNA
| Macromolecule | Name: crRNA / type: rna / ID: 2 / Number of copies: 1 |
|---|---|
| Source (natural) | Organism:  Candidatus Scalindua brodae (bacteria) Candidatus Scalindua brodae (bacteria) |
| Molecular weight | Theoretical: 35.43209 KDa |
| Sequence | String: GUUAUGAAAC AAGAGAAGGA CUUAAUGUCA CGGUACCCAA UUUUCUGCCC CGGACUCCAC GGCUGUUACU AGAGGUUAUG AAACAAGAG AAGGACUUAA UGUCACGGUA C |
-Macromolecule #4: ZINC ION
| Macromolecule | Name: ZINC ION / type: ligand / ID: 4 / Number of copies: 4 / Formula: ZN |
|---|---|
| Molecular weight | Theoretical: 65.409 Da |
-Macromolecule #5: MAGNESIUM ION
| Macromolecule | Name: MAGNESIUM ION / type: ligand / ID: 5 / Number of copies: 1 / Formula: MG |
|---|---|
| Molecular weight | Theoretical: 24.305 Da |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 8 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: SPOT SCAN / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.5 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
| Startup model | Type of model: INSILICO MODEL |
|---|---|
| Final reconstruction | Resolution.type: BY AUTHOR / Resolution: 2.61 Å / Resolution method: FSC 0.143 CUT-OFF / Number images used: 611350 |
| Initial angle assignment | Type: RANDOM ASSIGNMENT |
| Final angle assignment | Type: RANDOM ASSIGNMENT |
 Movie
Movie Controller
Controller








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)