+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
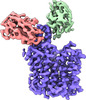
| タイトル | CryoEM structure of QacA (D411N), an antibacterial efflux transporter from Staphylococcus aureus | ||||||||||||
 マップデータ マップデータ | Refined map obtained from Non-uniform refinement and used for density modification and model building of QacA structure | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Drug proton antiporter / major facilitator superfamily(MFS) / Staphylococcus aureus / antibacterial efflux / QacA / MEMBRANE PROTEIN / MEMBRANE PROTEIN-IMMUNE SYSTEM complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   | ||||||||||||
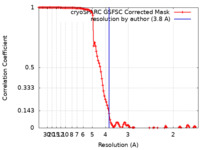
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | ||||||||||||
 データ登録者 データ登録者 | Penmatsa A / Majumder P | ||||||||||||
| 資金援助 |  インド, 3件 インド, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2023 ジャーナル: EMBO J / 年: 2023タイトル: Cryo-EM structure of antibacterial efflux transporter QacA from Staphylococcus aureus reveals a novel extracellular loop with allosteric role. 著者: Puja Majumder / Shahbaz Ahmed / Pragya Ahuja / Arunabh Athreya / Rakesh Ranjan / Aravind Penmatsa /  要旨: Efflux of antibacterial compounds is a major mechanism for developing antimicrobial resistance. In the Gram-positive pathogen Staphylococcus aureus, QacA, a 14 transmembrane helix containing major ...Efflux of antibacterial compounds is a major mechanism for developing antimicrobial resistance. In the Gram-positive pathogen Staphylococcus aureus, QacA, a 14 transmembrane helix containing major facilitator superfamily antiporter, mediates proton-coupled efflux of mono and divalent cationic antibacterial compounds. In this study, we report the cryo-EM structure of QacA, with a single mutation D411N that improves homogeneity and retains efflux activity against divalent cationic compounds like dequalinium and chlorhexidine. The structure of substrate-free QacA, complexed to two single-domain camelid antibodies, was elucidated to a resolution of 3.6 Å. The structure displays an outward-open conformation with an extracellular helical hairpin loop (EL7) between transmembrane helices 13 and 14, which is conserved in a subset of DHA2 transporters. Removal of the EL7 hairpin loop or disrupting the interface formed between EL7 and EL1 compromises efflux activity. Chimeric constructs of QacA with a helical hairpin and EL1 grafted from other DHA2 members, LfrA and SmvA, restore activity in the EL7 deleted QacA revealing the allosteric and vital role of EL7 hairpin in antibacterial efflux in QacA and related members. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33612.map.gz emd_33612.map.gz | 167.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33612-v30.xml emd-33612-v30.xml emd-33612.xml emd-33612.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33612_fsc.xml emd_33612_fsc.xml | 13.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33612.png emd_33612.png | 157.3 KB | ||
| Filedesc metadata |  emd-33612.cif.gz emd-33612.cif.gz | 6.8 KB | ||
| その他 |  emd_33612_additional_1.map.gz emd_33612_additional_1.map.gz emd_33612_half_map_1.map.gz emd_33612_half_map_1.map.gz emd_33612_half_map_2.map.gz emd_33612_half_map_2.map.gz | 3 MB 165 MB 165 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33612 http://ftp.pdbj.org/pub/emdb/structures/EMD-33612 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33612 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33612 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33612_validation.pdf.gz emd_33612_validation.pdf.gz | 899.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33612_full_validation.pdf.gz emd_33612_full_validation.pdf.gz | 899 KB | 表示 | |
| XML形式データ |  emd_33612_validation.xml.gz emd_33612_validation.xml.gz | 19.9 KB | 表示 | |
| CIF形式データ |  emd_33612_validation.cif.gz emd_33612_validation.cif.gz | 25.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33612 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33612 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33612 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33612 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7y58MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33612.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33612.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined map obtained from Non-uniform refinement and used for density modification and model building of QacA structure | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.831 Å | ||||||||||||||||||||||||||||||||||||
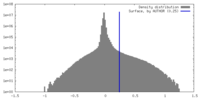
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: density modified map for building QacA structure
| ファイル | emd_33612_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | density modified map for building QacA structure | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
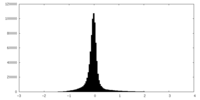
| 密度ヒストグラム |
-ハーフマップ: Halfmap B flipped for handedness
| ファイル | emd_33612_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap B flipped for handedness | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
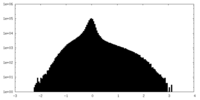
| 密度ヒストグラム |
-ハーフマップ: Half map A flipped for handed ness
| ファイル | emd_33612_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A flipped for handed ness | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
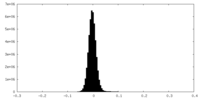
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Heterotrimer of QacA (54kDa) in complexed with two single domain ...
| 全体 | 名称: Heterotrimer of QacA (54kDa) in complexed with two single domain camelid antibodies (A4/B7) |
|---|---|
| 要素 |
|
-超分子 #1: Heterotrimer of QacA (54kDa) in complexed with two single domain ...
| 超分子 | 名称: Heterotrimer of QacA (54kDa) in complexed with two single domain camelid antibodies (A4/B7) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 54 kDa/nm |
-超分子 #2: QacA (54kDa)
| 超分子 | 名称: QacA (54kDa) / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: antibodies (A4/B7)
| 超分子 | 名称: antibodies (A4/B7) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|
-分子 #1: Antiseptic resistance protein
| 分子 | 名称: Antiseptic resistance protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 55.053457 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MISFFTKTTD MMTSKKRWTA LVVLAVSLFV VTMDMTILIM ALPELVRELE PSGTQQLWIV DIYSLVLAGF IIPLSAFADK WGRKKALLT GFALFGLVSL AIFFAESAEF VIAIRFLLGI AGALIMPTTL SMIRVIFENP KERATALAVW SIASSIGAVF G PIIGGALL ...文字列: MISFFTKTTD MMTSKKRWTA LVVLAVSLFV VTMDMTILIM ALPELVRELE PSGTQQLWIV DIYSLVLAGF IIPLSAFADK WGRKKALLT GFALFGLVSL AIFFAESAEF VIAIRFLLGI AGALIMPTTL SMIRVIFENP KERATALAVW SIASSIGAVF G PIIGGALL EQFSWHSAFL INVPFAIIAV VAGLFLLPES KLSKEKSHSW DIPSTILSIA GMIGLVWSIK EFSKEGLADI IP WVVIVLA ITMIVIFVKR NLSSSDPMLD VRLFKKRSFS AGTIAAFMTM FAMASVLLLA SQWLQVVEEL SPFKAGLYLL PMA IGDMVF APIAPGLAAR FGPKIVLPSG IGIAAIGMFI MYFFGHPLSY STMALALILV GAGMASLAVA SALIMLETPT SKAG NAAAV EESMYNLGNV FGVAVLGSLS SMLYRVFLDI SSFSSKGIVG DLAHVAEESV VGAVEVAKAT GIKQLANEAV TSFND AFVA TALVGGIIMI IISIVVYLLI PKSLDITKQK UniProtKB: Antiseptic resistance protein |
-分子 #2: Single-domain Indian camelid antibody (A4)
| 分子 | 名称: Single-domain Indian camelid antibody (A4) / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.697031 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQVQLQESGG GSVQTGGSLR LSCAASGYRY SDNCVGWFRQ APGREREAVA TYNSGSSTWY ADSVKGRFTI SQDSAKSTVY LQMNNLKPE DTAMYYCAGR NRLGSYCYMT GDFAYWGQGT QVTVSS |
-分子 #3: single-domain indian camelid antibody(B7)
| 分子 | 名称: single-domain indian camelid antibody(B7) / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.458037 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQVQLQESGG GSVQAGGSLR LSCAASGYTN SRKCMGWFRQ IPGKEREGVA AIYGFGRGLI LYADSVKGRF TISQDNAKNT VYLQMNSLK PEDTAMYYCA ADSPGSCLSR SGYNYWGQGT QVTVSS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7 詳細: 30mM Hepes, pH7.0 120mM NaCl 2 % glycerol 1mM Undecyl maltoside |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV / 詳細: blot time of 5 to 6 seconds before plunging. |
| 詳細 | QacA complex concentrated to 3 to 4 mg/ml before grid preparation |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: OTHER / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 7770 / 実像数: 7770 / 平均露光時間: 2.0 sec. / 平均電子線量: 51.0 e/Å2 詳細: Images were collected in movie mode at 50 images per movie |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Real space refinement |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 201 |
| 得られたモデル |  PDB-7y58: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)