+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | map focused on the interface between BA.2 RBD and ACE2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SARS-Cov-2 / VIRAL PROTEIN / VIRAL PROTEIN-HYDROLASE complex | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Li YN / Shen YP / Zhang YY / Yan RH | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2023 ジャーナル: Viruses / 年: 2023タイトル: Structural Basis for the Enhanced Infectivity and Immune Evasion of Omicron Subvariants. 著者: Yaning Li / Yaping Shen / Yuanyuan Zhang / Renhong Yan /  要旨: The Omicron variants of SARS-CoV-2 have emerged as the dominant strains worldwide, causing the COVID-19 pandemic. Each Omicron subvariant contains at least 30 mutations on the spike protein (S ...The Omicron variants of SARS-CoV-2 have emerged as the dominant strains worldwide, causing the COVID-19 pandemic. Each Omicron subvariant contains at least 30 mutations on the spike protein (S protein) compared to the original wild-type (WT) strain. Here we report the cryo-EM structures of the trimeric S proteins from the BA.1, BA.2, BA.3, and BA.4/BA.5 subvariants, with BA.4 and BA.5 sharing the same S protein mutations, each in complex with the surface receptor ACE2. All three receptor-binding domains of the S protein from BA.2 and BA.4/BA.5 are "up", while the BA.1 S protein has two "up" and one "down". The BA.3 S protein displays increased heterogeneity, with the majority in the all "up" RBD state. The different conformations preferences of the S protein are consistent with their varied transmissibility. By analyzing the position of the glycan modification on Asn343, which is located at the S309 epitopes, we have uncovered the underlying immune evasion mechanism of the Omicron subvariants. Our findings provide a molecular basis of high infectivity and immune evasion of Omicron subvariants, thereby offering insights into potential therapeutic interventions against SARS-CoV-2 variants. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33579.map.gz emd_33579.map.gz | 86 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33579-v30.xml emd-33579-v30.xml emd-33579.xml emd-33579.xml | 13.2 KB 13.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33579.png emd_33579.png | 19.9 KB | ||
| その他 |  emd_33579_half_map_1.map.gz emd_33579_half_map_1.map.gz emd_33579_half_map_2.map.gz emd_33579_half_map_2.map.gz | 84.6 MB 84.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33579 http://ftp.pdbj.org/pub/emdb/structures/EMD-33579 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33579 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33579 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33579_validation.pdf.gz emd_33579_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33579_full_validation.pdf.gz emd_33579_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_33579_validation.xml.gz emd_33579_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_33579_validation.cif.gz emd_33579_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33579 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33579 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33579 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33579 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33579.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33579.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.087 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_33579_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
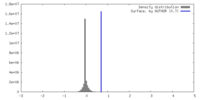
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33579_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
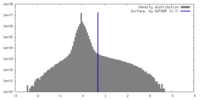
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : S-ECD (Omicron BA.3) in complex with 2 PD of ACE2
| 全体 | 名称: S-ECD (Omicron BA.3) in complex with 2 PD of ACE2 |
|---|---|
| 要素 |
|
-超分子 #1: S-ECD (Omicron BA.3) in complex with 2 PD of ACE2
| 超分子 | 名称: S-ECD (Omicron BA.3) in complex with 2 PD of ACE2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: S-ECD (Omicron BA.3)
| 超分子 | 名称: S-ECD (Omicron BA.3) / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: PD of ACE2
| 超分子 | 名称: PD of ACE2 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4 |
|---|
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0.6) / 使用した粒子像数: 35756 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)