+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Fft3-nucleosome complex with Fft3 bound to SHL+2 position of the nucleosome | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA binding / remodeler / nucleosome / Fft3-nucleosome complex / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報attachment of telomeric heterochromatin to nuclear envelope / ATP-dependent H3-H4 histone complex chaperone activity / HDMs demethylate histones / PKMTs methylate histone lysines / Interleukin-7 signaling / Chromatin modifying enzymes / Condensation of Prophase Chromosomes / SUMOylation of chromatin organization proteins / Metalloprotease DUBs / E3 ubiquitin ligases ubiquitinate target proteins ...attachment of telomeric heterochromatin to nuclear envelope / ATP-dependent H3-H4 histone complex chaperone activity / HDMs demethylate histones / PKMTs methylate histone lysines / Interleukin-7 signaling / Chromatin modifying enzymes / Condensation of Prophase Chromosomes / SUMOylation of chromatin organization proteins / Metalloprotease DUBs / E3 ubiquitin ligases ubiquitinate target proteins / Factors involved in megakaryocyte development and platelet production / RCAF complex / RMTs methylate histone arginines / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / polytene chromosome band / SIRT1 negatively regulates rRNA expression / NoRC negatively regulates rRNA expression / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / Formation of the beta-catenin:TCF transactivating complex / PRC2 methylates histones and DNA / HDACs deacetylate histones / Ub-specific processing proteases / RNA Polymerase I Promoter Escape / MLL4 and MLL3 complexes regulate expression of PPARG target genes in adipogenesis and hepatic steatosis / Regulation of endogenous retroelements by KRAB-ZFP proteins / larval somatic muscle development / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Senescence-Associated Secretory Phenotype (SASP) / Transcriptional regulation by small RNAs / Estrogen-dependent gene expression / HATs acetylate histones / UCH proteinases / Assembly of the ORC complex at the origin of replication / Oxidative Stress Induced Senescence / polytene chromosome / histone chaperone activity / DNA double-strand break processing / nucleosome array spacer activity / nucleosomal DNA binding / transcription elongation-coupled chromatin remodeling / nuclear chromosome / DNA repair-dependent chromatin remodeling / replication fork processing / heterochromatin / structural constituent of chromatin / nucleosome / heterochromatin formation / nucleosome assembly / chromatin organization / chromosome / DNA helicase / damaged DNA binding / forked DNA-dependent helicase activity / single-stranded 3'-5' DNA helicase activity / four-way junction helicase activity / double-stranded DNA helicase activity / chromatin remodeling / protein heterodimerization activity / chromatin binding / protein-containing complex binding / chromatin / positive regulation of transcription by RNA polymerase II / ATP hydrolysis activity / DNA binding / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Nan Z / Tao J / Yangao H | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of Fft3-nucleosome complex with Fft3 bound to SHL+2 position of the nucleosome (Class I Fft3-nucleosome complex) 著者: Nan Z / Tao J / Yangao H | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33520.map.gz emd_33520.map.gz | 39.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33520-v30.xml emd-33520-v30.xml emd-33520.xml emd-33520.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33520.png emd_33520.png | 212.9 KB | ||
| Filedesc metadata |  emd-33520.cif.gz emd-33520.cif.gz | 7 KB | ||
| その他 |  emd_33520_half_map_1.map.gz emd_33520_half_map_1.map.gz emd_33520_half_map_2.map.gz emd_33520_half_map_2.map.gz | 32.9 MB 32.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33520 http://ftp.pdbj.org/pub/emdb/structures/EMD-33520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33520 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7xyfMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33520.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33520.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
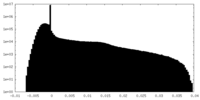
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33520_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
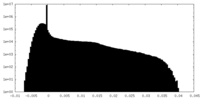
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33520_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
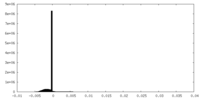
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Fft3-nucleosome complex with Fft3 bound to SHL+2 of th...
| 全体 | 名称: Complex of Fft3-nucleosome complex with Fft3 bound to SHL+2 of the nucleosome (Class I Fft3-nucleosome complex) |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Fft3-nucleosome complex with Fft3 bound to SHL+2 of th...
| 超分子 | 名称: Complex of Fft3-nucleosome complex with Fft3 bound to SHL+2 of the nucleosome (Class I Fft3-nucleosome complex) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #6-#7, #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Histone H2A
| 分子 | 名称: Histone H2A / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.552494 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AKSRSNRAGL QFPVGRIHRL LRKGNYAERV GAGAPVYLAA VMEYLAAEVL ELAGNAARDN KKTRIIPRHL QLAIRNDEEL NKLLSGVTI AQGGVLPNIQ AVLLPKK UniProtKB: Histone H2A |
-分子 #2: Histone H2B
| 分子 | 名称: Histone H2B / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 10.721382 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RKRKESYAIY IYKVLKQVHP DTGISSKAMS IMNSFVNDIF ERIAAEASRL AHYNKRSTIT SREIQTAVRL LLPGELAKHA VSEGTKAVT KYTSSK UniProtKB: Histone H2B |
-分子 #5: ATP-dependent helicase fft3
| 分子 | 名称: ATP-dependent helicase fft3 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 78.351766 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: IDTAALKEEV LKY(MSE)NRCSTQ DLAD(MSE)TGCTL AEAEF(MSE)VAKR PFPDLESALV VKQPRPVIPK GRRGRREK T PLGPRLVGIC (MSE)EI(MSE)RGYFVV DALIRQCEQL GGKIQRGIEA WGLSNTATSD EGETSLVNFD Q(MSE)KSFGT PA NSSFITTPPA ...文字列: IDTAALKEEV LKY(MSE)NRCSTQ DLAD(MSE)TGCTL AEAEF(MSE)VAKR PFPDLESALV VKQPRPVIPK GRRGRREK T PLGPRLVGIC (MSE)EI(MSE)RGYFVV DALIRQCEQL GGKIQRGIEA WGLSNTATSD EGETSLVNFD Q(MSE)KSFGT PA NSSFITTPPA SFSPDIKLQD YQIIGINWLY LLYELKLAGI LADE(MSE)GLGKT CQTIAFFSLL (MSE)DKNINGPHL VIAPAST(MSE)E NWLREFAKFC PKLKIELYYG SQVEREEIRE RINSNKDSYN V(MSE)LTTYRLAA TSKADRLFLR NQK FNVCVY DEGHYLKNRA SERYRHL(MSE)SI PADFRVLLTG TPLQNNLKEL ISLLAFILPH VFDYGLKSLD VIFT(MSE)K KSP ESDFERALLS EQRVSRAK(MSE)(MSE) (MSE)APFVLRRKK SQVLDALPKK TRIIEFCEFS EEERRRYDDF ASKQS VNSL LDENV(MSE)KTNL DTNANLAKKK STAGFVLVQL RKLADHP(MSE)LF RIHYKDDILR Q(MSE)AKAI(MSE)NEP QYKKANELY IFED(MSE)QY(MSE)SD IELHNLCCKF PSINSFQLKD EPW(MSE)DATKVR KLKKLLTNAV ENGDRVVLF SQFTQVLDIL QLV(MSE)KSLNLK FLRFDGSTQV DFRQDLIDQF YADESINVFL LSTKAGGFGI NLACAN(MSE)VIL YD VSFNPFD DLQAEDRAHR VGQKKEVTVY KFVVKDTIEE HIQRLANAKI A UniProtKB: ATP-dependent helicase fft3 |
-分子 #6: Histone H3
| 分子 | 名称: Histone H3 / タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.48841 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PHRYRPGTVA LREIRRYQKS TELLIRKLPF QRLVREIAQD FKTDLRFQSS AVMALQEASE AYLVGLFEDT NLCAIHAKRV TIMPKDIQL ARRIRGERA UniProtKB: Histone H3 |
-分子 #7: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.86159 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RHRKVLRDNI QGITKPAIRR LARRGGVKRI SGLIYEETRG VLKVFLENVI RDAVTYTEHA KRKTVTAMDV VYALKRQGRT LYGFGG UniProtKB: Histone H4 |
-分子 #3: DNA (167-MER)
| 分子 | 名称: DNA (167-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.27484 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT) ...文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC)(DC) (DC)(DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DT) (DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG)(DG) (DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC) (DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC)(DG) (DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT) (DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC) (DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC)(DG) (DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC) (DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC)(DT) (DC)(DC)(DA) |
-分子 #4: DNA (167-MER)
| 分子 | 名称: DNA (167-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 44.85657 KDa |
| 配列 | 文字列: (DT)(DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT) ...文字列: (DT)(DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT)(DC)(DT)(DA) (DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT)(DA) (DA)(DA) (DC)(DG)(DC)(DA)(DC)(DG)(DT) (DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT)(DC) (DC)(DC)(DC) (DC)(DG)(DC)(DG)(DT)(DT) (DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC)(DA) (DA)(DG)(DG)(DG) (DG)(DA)(DT)(DT)(DA) (DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT)(DC) (DT)(DC)(DC)(DA)(DG) (DG)(DC)(DA)(DC) (DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT)(DA) (DT)(DA)(DT)(DA)(DC)(DA) (DT)(DC)(DC) (DT)(DG)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.5 µm |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 178242 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)