+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 spike glycoprotein in complex with neutralizing antibody UT28K | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
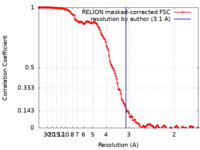
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Ozawa T / Tani H / Anraku Y / Kita S / Igarashi E / Saga Y / Inasaki N / Kawasuji H / Yamada H / Sasaki S ...Ozawa T / Tani H / Anraku Y / Kita S / Igarashi E / Saga Y / Inasaki N / Kawasuji H / Yamada H / Sasaki S / Someoka M / Sasaki J / Hayakawa Y / Yamamoto Y / Morinaga Y / Kurosawa N / Isobe M / Fukuhara H / Maenaka K / Hashiguchi T / Kishi H / Kitajima I / Saito S / Niimi H | |||||||||||||||
| 資金援助 |  日本, 4件 日本, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: MAbs / 年: 2022 ジャーナル: MAbs / 年: 2022タイトル: Novel super-neutralizing antibody UT28K is capable of protecting against infection from a wide variety of SARS-CoV-2 variants. 著者: Tatsuhiko Ozawa / Hideki Tani / Yuki Anraku / Shunsuke Kita / Emiko Igarashi / Yumiko Saga / Noriko Inasaki / Hitoshi Kawasuji / Hiroshi Yamada / So-Ichiro Sasaki / Mayu Somekawa / Jiei ...著者: Tatsuhiko Ozawa / Hideki Tani / Yuki Anraku / Shunsuke Kita / Emiko Igarashi / Yumiko Saga / Noriko Inasaki / Hitoshi Kawasuji / Hiroshi Yamada / So-Ichiro Sasaki / Mayu Somekawa / Jiei Sasaki / Yoshihiro Hayakawa / Yoshihiro Yamamoto / Yoshitomo Morinaga / Nobuyuki Kurosawa / Masaharu Isobe / Hideo Fukuhara / Katsumi Maenaka / Takao Hashiguchi / Hiroyuki Kishi / Isao Kitajima / Shigeru Saito / Hideki Niimi /  要旨: Many potent neutralizing SARS-CoV-2 antibodies have been developed and used for therapies. However, the effectiveness of many antibodies has been reduced against recently emerging SARS-CoV-2 ...Many potent neutralizing SARS-CoV-2 antibodies have been developed and used for therapies. However, the effectiveness of many antibodies has been reduced against recently emerging SARS-CoV-2 variants, especially the Omicron variant. We identified a highly potent SARS-CoV-2 neutralizing antibody, UT28K, in COVID-19 convalescent individuals who recovered from a severe condition. UT28K showed efficacy in neutralizing SARS-CoV-2 in an assay and prophylactic treatment, and the reactivity to the Omicron strain was reduced. The structural analyses revealed that antibody UT28K Fab and SARS-CoV-2 RBD protein interactions were mainly chain-dominated antigen-antibody interactions. In addition, a mutation analysis suggested that the emergence of a UT28K neutralization-resistant SARS-CoV-2 variant was unlikely, as this variant would likely lose its competitive advantage over circulating SARS-CoV-2. Our data suggest that UT28K offers potent protection against SARS-CoV-2, including newly emerging variants. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33045.map.gz emd_33045.map.gz | 170.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33045-v30.xml emd-33045-v30.xml emd-33045.xml emd-33045.xml | 24.2 KB 24.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33045_fsc.xml emd_33045_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33045.png emd_33045.png | 99.8 KB | ||
| マスクデータ |  emd_33045_msk_1.map emd_33045_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| その他 |  emd_33045_half_map_1.map.gz emd_33045_half_map_1.map.gz emd_33045_half_map_2.map.gz emd_33045_half_map_2.map.gz | 171.2 MB 171.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33045 http://ftp.pdbj.org/pub/emdb/structures/EMD-33045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33045 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33045_validation.pdf.gz emd_33045_validation.pdf.gz | 819.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33045_full_validation.pdf.gz emd_33045_full_validation.pdf.gz | 818.8 KB | 表示 | |
| XML形式データ |  emd_33045_validation.xml.gz emd_33045_validation.xml.gz | 21 KB | 表示 | |
| CIF形式データ |  emd_33045_validation.cif.gz emd_33045_validation.cif.gz | 27.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33045 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33045 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33045 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33045 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33045.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33045.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33045_msk_1.map emd_33045_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33045_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33045_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-COV-2 spike glycoprotein in complex with UT28K
| 全体 | 名称: SARS-COV-2 spike glycoprotein in complex with UT28K |
|---|---|
| 要素 |
|
-超分子 #1: SARS-COV-2 spike glycoprotein in complex with UT28K
| 超分子 | 名称: SARS-COV-2 spike glycoprotein in complex with UT28K / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 50 KDa |
-超分子 #2: SARS-CoV-2 spike glycoprotein
| 超分子 | 名称: SARS-CoV-2 spike glycoprotein / タイプ: complex / キメラ: Yes / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-超分子 #3: UT28K Fab
| 超分子 | 名称: UT28K Fab / タイプ: complex / キメラ: Yes / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: SARS-CoV-2 spike glycoprotein
| 分子 | 名称: SARS-CoV-2 spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SSQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHVS GTNGTKRFDN PVLPFNDGVY FASTEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL GVYYHKNNKS WMESEFRVYS SANNCTFEYV SQPFLMDLEG ...文字列: SSQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHVS GTNGTKRFDN PVLPFNDGVY FASTEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL GVYYHKNNKS WMESEFRVYS SANNCTFEYV SQPFLMDLEG KQGNFKNLRE FVFKNIDGYF KIYSKHTPIN LVRDLPQGFS ALEPLVDLPI GINITRFQTL LALHRSYLTP GDSSSGWTAG AAAYYVGYLQ PRTFLLKYNE NGTITDAVDC ALDPLSETKC TLKSFTVEKG IYQTSNFRVQ PTESIVRFPN ITNLCPFGEV FNATRFASVY AWNRKRISNC VADYSVLYNS ASFSTFKCYG VSPTKLNDLC FTNVYADSFV IRGDEVRQIA PGQTGKIADY NYKLPDDFTG CVIAWNSNNL DSKVGGNYNY LYRLFRKSNL KPFERDISTE IYQAGSTPCN GVEGFNCYFP LQSYGFQPTN GVGYQPYRVV VLSFELLHAP ATVCGPKKST NLVKNKCVNF NFNGLTGTGV LTESNKKFLP FQQFGRDIAD TTDAVRDPQT LEILDITPCS FGGVSVITPG TNTSNQVAVL YQDVNCTEVP VAIHADQLTP TWRVYSTGSN VFQTRAGCLI GAEHVNNSYE CDIPIGAGIC ASYQTQTQTN SPGSAGSVAS QSIIAYTMSL GAENSVAYSN NSIAIPTNFT ISVTTEILPV SMTKTSVDCT MYICGDSTEC SNLLLQYGSF CTQLNRALTG IAVEQDKNTQ EVFAQVKQIY KTPPIKDFGG FNFSQILPDP SKPSKRSPIE DLLFNKVTLA DAGFIKQYGD CLGDIAARDL ICAQKFNGLT VLPPLLTDEM IAQYTSALLA GTITSGWTFG AGPALQIPFP MQMAYRFNGI GVTQNVLYEN QKLIANQFNS AIGKIQDSLS STPSALGKLQ DVVNQNAQAL NTLVKQLSSN FGAISSVLND ILSRLDPPEA EVQIDRLITG RLQSLQTYVT QQLIRAAEIR ASANLAATKM SECVLGQSKR VDFCGKGYHL MSFPQSAPHG VVFLHVTYVP AQEKNFTTAP AICHDGKAHF PREGVFVSNG THWFVTQRNF YEPQIITTDN TFVSGNCDVV IGIVNNTVYD PLQPELDSFK EELDKYFKNH TSPDVDLGDI SGINASVVNI QKEIDRLNEV AKNLNESLID LQELGKYEQY I |
-分子 #2: UT28K Fab heavy chain
| 分子 | 名称: UT28K Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDWIWRILFL VGAATGAHSQ MQLVQSGPEV KKPGTSVKVS CKASGFTFSI SAVQWVRQAR GQRLEWIGWI VVGSGNTNYA QKFQERVTIT RDMSTSTAYM ELSSLRFEDT AVYYCAAPYC GGDCSDGFDI WGQGTMVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV ...文字列: MDWIWRILFL VGAATGAHSQ MQLVQSGPEV KKPGTSVKVS CKASGFTFSI SAVQWVRQAR GQRLEWIGWI VVGSGNTNYA QKFQERVTIT RDMSTSTAYM ELSSLRFEDT AVYYCAAPYC GGDCSDGFDI WGQGTMVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV SWNSGALTSG VHTFPAVLQS SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKKVE PKSGSSGHHH HHHHHHH |
-分子 #3: UT28K Fab light chain
| 分子 | 名称: UT28K Fab light chain / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: METPAQLLFL LLLWLPDTTG EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP DRFSGSGSGT DFTLTISRLE PEDFAVYYCQ QYGNSPWTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK ...文字列: METPAQLLFL LLLWLPDTTG EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP DRFSGSGSGT DFTLTISRLE PEDFAVYYCQ QYGNSPWTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS QESVTEQDSK DSTYSLSSTL TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 2217 / 平均露光時間: 1.5 sec. / 平均電子線量: 53.14 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X