+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
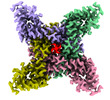
| タイトル | Ion channel | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channel / TRANSPORT PROTEIN | |||||||||
| 生物種 |  Emiliania huxleyi (真核生物) Emiliania huxleyi (真核生物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.83 Å | |||||||||
 データ登録者 データ登録者 | Jiang D / Zhang J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: N-type fast inactivation of a eukaryotic voltage-gated sodium channel. 著者: Jiangtao Zhang / Yiqiang Shi / Junping Fan / Huiwen Chen / Zhanyi Xia / Bo Huang / Juquan Jiang / Jianke Gong / Zhuo Huang / Daohua Jiang /  要旨: Voltage-gated sodium (Na) channels initiate action potentials. Fast inactivation of Na channels, mediated by an Ile-Phe-Met motif, is crucial for preventing hyperexcitability and regulating firing ...Voltage-gated sodium (Na) channels initiate action potentials. Fast inactivation of Na channels, mediated by an Ile-Phe-Met motif, is crucial for preventing hyperexcitability and regulating firing frequency. Here we present cryo-electron microscopy structure of NaEh from the coccolithophore Emiliania huxleyi, which reveals an unexpected molecular gating mechanism for Na channel fast inactivation independent of the Ile-Phe-Met motif. An N-terminal helix of NaEh plugs into the open activation gate and blocks it. The binding pose of the helix is stabilized by multiple electrostatic interactions. Deletion of the helix or mutations blocking the electrostatic interactions completely abolished the fast inactivation. These strong interactions enable rapid inactivation, but also delay recovery from fast inactivation, which is ~160-fold slower than human Na channels. Together, our results provide mechanistic insights into fast inactivation of NaEh that fundamentally differs from the conventional local allosteric inhibition, revealing both surprising structural diversity and functional conservation of ion channel inactivation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33016.map.gz emd_33016.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33016-v30.xml emd-33016-v30.xml emd-33016.xml emd-33016.xml | 12.5 KB 12.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33016.png emd_33016.png | 151.9 KB | ||
| Filedesc metadata |  emd-33016.cif.gz emd-33016.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33016 http://ftp.pdbj.org/pub/emdb/structures/EMD-33016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33016 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33016.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33016.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ion channel complex
| 全体 | 名称: ion channel complex |
|---|---|
| 要素 |
|
-超分子 #1: ion channel complex
| 超分子 | 名称: ion channel complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Emiliania huxleyi (真核生物) Emiliania huxleyi (真核生物) |
-分子 #1: ion channel,GFP-TwinStrep
| 分子 | 名称: ion channel,GFP-TwinStrep / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Emiliania huxleyi (真核生物) Emiliania huxleyi (真核生物) |
| 分子量 | 理論値: 91.684469 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MIAAIHNARR KKREAAAAHK AQHRTAENSM DSLEDSTHET DAGERAQAGS TKLAWTDVVA PPPRKVVFWL PHQRKVFDFY ASQGVQYFT AFLIVSNFIF NCAEKEWDPY TDQLYQGLWR WGEFAFNTMF LIELLINFYG IAFCFWRYNW AWNTFDLVVV A IGTLTMAE ...文字列: MIAAIHNARR KKREAAAAHK AQHRTAENSM DSLEDSTHET DAGERAQAGS TKLAWTDVVA PPPRKVVFWL PHQRKVFDFY ASQGVQYFT AFLIVSNFIF NCAEKEWDPY TDQLYQGLWR WGEFAFNTMF LIELLINFYG IAFCFWRYNW AWNTFDLVVV A IGTLTMAE AIGGNFMPPS MALIRNLRAF RIFRLFKRIK SLNKIIVSLG KAIPGVANAF VIMVIIMCIY AILGVEFYHM TG SDGTYVT YNDNVKRGLC TGDEVELGQC SLNQTVSSET ARGYTYGEEY YGTFFRALYT LFQVLTGESW SEAVARPAVF ESH YDSFGP VLFYVSFIII CQIVLINVVV AVLLDKMVEE DDSEDPEKQT VAEKLSEMLS QEHAQLREIF RTWDEDNSGT ISIK EWRKA VKSMGYRGPI DVLDQIFASM DKDHSGELDY AEIDRMLSPT AARERRSSTH ANPKRSVKEE VVAMRAEFTD HVARL ETQI AALVLELQLQ RKPCGAEAPA PAHSRLAHDS DGAPTEPPPP AAPDHHHLED DEDTTQRVAA ALEVLFQGPS KGEELF TGV VPILVELDGD VNGHKFSVRG EGEGDATNGK LTLKFICTTG KLPVPWPTLV TTLTYGVQCF SRYPDHMKRH DFFKSAM PE GYVQERTISF KDDGTYKTRA EVKFEGDTLV NRIELKGIDF KEDGNILGHK LEYNFNSHNV YITADKQKNG IKANFKIR H NVEDGSVQLA DHYQQNTPIG DGPVLLPDNH YLSTQSVLSK DPNEKRDHMV LLEFVTAAGI THGMDEWSHP QFEKGGGSG GGSGGSAWSH PQFEK |
-分子 #2: ion channel
| 分子 | 名称: ion channel / タイプ: protein_or_peptide / ID: 2 / 詳細: N-terminal helix of either of the four protomers / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Emiliania huxleyi (真核生物) Emiliania huxleyi (真核生物) |
| 分子量 | 理論値: 1.913298 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MIAAIHNARR KKREAAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















