+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
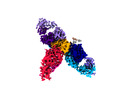
| タイトル | Cryo-EM structure of SARS-CoV-2 recombinant spike protein STFK in complex with three neutralizing antibodies | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SARS-CoV-2 / spike / vaccine / neutralizing antibody / Cryo-EM / VIRAL PROTEIN / IMMUNE SYSTEM-VIRAL PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / entry receptor-mediated virion attachment to host cell / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.81 Å | |||||||||
 データ登録者 データ登録者 | Zheng Q / Sun H | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2022 ジャーナル: Cell Host Microbe / 年: 2022タイトル: Lineage-mosaic and mutation-patched spike proteins for broad-spectrum COVID-19 vaccine. 著者: Yangtao Wu / Shaojuan Wang / Yali Zhang / Lunzhi Yuan / Qingbing Zheng / Min Wei / Yang Shi / Zikang Wang / Jian Ma / Kai Wang / Meifeng Nie / Jin Xiao / Zehong Huang / Peiwen Chen / Huilin ...著者: Yangtao Wu / Shaojuan Wang / Yali Zhang / Lunzhi Yuan / Qingbing Zheng / Min Wei / Yang Shi / Zikang Wang / Jian Ma / Kai Wang / Meifeng Nie / Jin Xiao / Zehong Huang / Peiwen Chen / Huilin Guo / Miaolin Lan / Jingjing Xu / Wangheng Hou / Yunda Hong / Dabing Chen / Hui Sun / Hualong Xiong / Ming Zhou / Che Liu / Wenjie Guo / Huiyu Guo / Jiahua Gao / Congling Gan / Zhixiong Li / Haitao Zhang / Xinrui Wang / Shaowei Li / Tong Cheng / Qinjian Zhao / Yixin Chen / Ting Wu / Tianying Zhang / Jun Zhang / Hua Cao / Huachen Zhu / Quan Yuan / Yi Guan / Ningshao Xia /  要旨: SARS-CoV-2 spread in humans results in continuous emergence of new variants, highlighting the need for vaccines with broad-spectrum antigenic coverage. Using inter-lineage chimera and mutation-patch ...SARS-CoV-2 spread in humans results in continuous emergence of new variants, highlighting the need for vaccines with broad-spectrum antigenic coverage. Using inter-lineage chimera and mutation-patch strategies, we engineered a recombinant monomeric spike variant (STFK1628x) that contains key regions and residues across multiple SAR-CoV-2 variants. STFK1628x demonstrated high immunogenicity and mutually complementary antigenicity to its prototypic form (STFK). In hamsters, a bivalent vaccine composed of STFK and STFK1628x elicited high titers of broad-spectrum neutralizing antibodies to 19 circulating SARS-CoV-2 variants, including Omicron sublineages BA.1, BA.1.1, BA.2, BA.2.12.1, BA.2.75, and BA.4/5. Furthermore, this vaccine conferred robust protection against intranasal challenges by either SARS-CoV-2 ancestral strain or immune-evasive Beta and Omicron BA.1. Strikingly, vaccination with the bivalent vaccine in hamsters effectively blocked within-cage virus transmission of ancestral SARS-CoV-2, Beta variant, and Omicron BA.1 to unvaccinated sentinels. Thus, our study provided insight and antigen candidates for the development of next-generation COVID-19 vaccines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32676.map.gz emd_32676.map.gz | 230 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32676-v30.xml emd-32676-v30.xml emd-32676.xml emd-32676.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32676.png emd_32676.png | 24.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32676 http://ftp.pdbj.org/pub/emdb/structures/EMD-32676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32676 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32676_validation.pdf.gz emd_32676_validation.pdf.gz | 474.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32676_full_validation.pdf.gz emd_32676_full_validation.pdf.gz | 473.9 KB | 表示 | |
| XML形式データ |  emd_32676_validation.xml.gz emd_32676_validation.xml.gz | 7.2 KB | 表示 | |
| CIF形式データ |  emd_32676_validation.cif.gz emd_32676_validation.cif.gz | 8.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32676 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32676 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7wp6MC  7wp8C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32676.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32676.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.778 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Cryo-EM structure of SARS-CoV-2 recombinant spike protein STFK in...
+超分子 #1: Cryo-EM structure of SARS-CoV-2 recombinant spike protein STFK in...
+超分子 #2: neutralizing antibodies
+超分子 #3: spike protein STFK
+分子 #1: 36H6 heavy chain
+分子 #2: 83H7 light chain
+分子 #3: 83H7 heavy chain
+分子 #4: Spike protein S1
+分子 #5: 85F7 heavy chain
+分子 #6: 36H6 light chain
+分子 #7: 85F7 light chain
+分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.81 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 162177 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー