+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure and dynamics of Odinarchaeota tubulin and the implications for eukaryotic microtubule evolution | ||||||||||||
 マップデータ マップデータ | Reconstruction of tubes of OdinTubulin sampled from Yellowstone Lower Culex Basin hot spring polymerized at 37 degrees Celsius | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Eukaryotic tubulin homolog / STRUCTURAL PROTEIN | ||||||||||||
| 生物種 |  Candidatus Odinarchaeota archaeon LCB_4 (古細菌) Candidatus Odinarchaeota archaeon LCB_4 (古細菌) | ||||||||||||
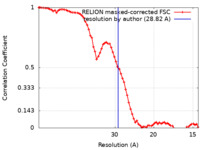
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 28.82 Å | ||||||||||||
 データ登録者 データ登録者 | Akil C / Ali S / Tran LT | ||||||||||||
| 資金援助 | 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Structure and dynamics of Odinarchaeota tubulin and the implications for eukaryotic microtubule evolution. 著者: Caner Akıl / Samson Ali / Linh T Tran / Jérémie Gaillard / Wenfei Li / Kenichi Hayashida / Mika Hirose / Takayuki Kato / Atsunori Oshima / Kosuke Fujishima / Laurent Blanchoin / Akihiro ...著者: Caner Akıl / Samson Ali / Linh T Tran / Jérémie Gaillard / Wenfei Li / Kenichi Hayashida / Mika Hirose / Takayuki Kato / Atsunori Oshima / Kosuke Fujishima / Laurent Blanchoin / Akihiro Narita / Robert C Robinson /     要旨: Tubulins are critical for the internal organization of eukaryotic cells, and understanding their emergence is an important question in eukaryogenesis. Asgard archaea are the closest known prokaryotic ...Tubulins are critical for the internal organization of eukaryotic cells, and understanding their emergence is an important question in eukaryogenesis. Asgard archaea are the closest known prokaryotic relatives to eukaryotes. Here, we elucidated the apo and nucleotide-bound x-ray structures of an Asgard tubulin from hydrothermal living Odinarchaeota (OdinTubulin). The guanosine 5'-triphosphate (GTP)-bound structure resembles a microtubule protofilament, with GTP bound between subunits, coordinating the "+" end subunit through a network of water molecules and unexpectedly by two cations. A water molecule is located suitable for GTP hydrolysis. Time course crystallography and electron microscopy revealed conformational changes on GTP hydrolysis. OdinTubulin forms tubules at high temperatures, with short curved protofilaments coiling around the tubule circumference, more similar to FtsZ, rather than running parallel to its length, as in microtubules. Thus, OdinTubulin represents an evolutionary stage intermediate between prokaryotic FtsZ and eukaryotic microtubule-forming tubulins. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32630.map.gz emd_32630.map.gz | 3.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32630-v30.xml emd-32630-v30.xml emd-32630.xml emd-32630.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32630_fsc.xml emd_32630_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32630.png emd_32630.png | 34 KB | ||
| Filedesc metadata |  emd-32630.cif.gz emd-32630.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32630 http://ftp.pdbj.org/pub/emdb/structures/EMD-32630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32630 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32630_validation.pdf.gz emd_32630_validation.pdf.gz | 379.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32630_full_validation.pdf.gz emd_32630_full_validation.pdf.gz | 379.3 KB | 表示 | |
| XML形式データ |  emd_32630_validation.xml.gz emd_32630_validation.xml.gz | 9.3 KB | 表示 | |
| CIF形式データ |  emd_32630_validation.cif.gz emd_32630_validation.cif.gz | 12.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32630 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32630 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32630 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32630 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32630.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32630.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of tubes of OdinTubulin sampled from Yellowstone Lower Culex Basin hot spring polymerized at 37 degrees Celsius | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.25 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tubulin homolog from Candidatus Odinarchaeota archaeon
| 全体 | 名称: Tubulin homolog from Candidatus Odinarchaeota archaeon |
|---|---|
| 要素 |
|
-超分子 #1: Tubulin homolog from Candidatus Odinarchaeota archaeon
| 超分子 | 名称: Tubulin homolog from Candidatus Odinarchaeota archaeon タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: OdinTubulin was sampled from Candidatus Odinarchaeota archaeon LCB_4 MAG whose habitat is Yellowstone Lower Culex Basin hot spring. |
|---|---|
| 由来(天然) | 生物種:  Candidatus Odinarchaeota archaeon LCB_4 (古細菌) Candidatus Odinarchaeota archaeon LCB_4 (古細菌) |
| 分子量 | 理論値: 47.6 kDa/nm |
-分子 #1: OdinTubulin
| 分子 | 名称: OdinTubulin / タイプ: other / ID: 1 詳細: Candidatus Odinarchaeota archaeon LCB_4 (Odin) metagenome-assembled genome 分類: other |
|---|---|
| 由来(天然) | 生物種:  Candidatus Odinarchaeota archaeon LCB_4 (古細菌) Candidatus Odinarchaeota archaeon LCB_4 (古細菌) |
| 配列 | 文字列: PMPGREILVL HVGQGGNQIG YNFWKTICEE HNIDIRSNQR KSVEEDKVDY KSVFLVEAPD GFHPRALFID LEPLAVEFLV KEMKLGSFFS EDLMVLSYSG AHNVWSIGYQ TGKKLIPVIL EKIRDTMPET LQGFLIIHTL GGGTGSGFGS LLTETLKKEF PGKGVLNFSV ...文字列: PMPGREILVL HVGQGGNQIG YNFWKTICEE HNIDIRSNQR KSVEEDKVDY KSVFLVEAPD GFHPRALFID LEPLAVEFLV KEMKLGSFFS EDLMVLSYSG AHNVWSIGYQ TGKKLIPVIL EKIRDTMPET LQGFLIIHTL GGGTGSGFGS LLTETLKKEF PGKGVLNFSV LPSEVNDVTL APYNTVLSLN HLSRFSDLVV LFDNTALIRI VKDQLNYPVI KQFSDLNFLI GRVMASITAS LRFPGPLNMD LMEMAHNLVA LPETKFIIPS VAPLTKEESE MSTELDLVER CFDPTHYMVN CSGQGKTISS VLMFRGNIAI ENAFSIMTDI KSNVAFAPGV HPDLGLKYGI CESAPVDFDK EVTLLSNNTI ISEVFNRVLE RFDSLFNRDW YTSHYVNAGT SKSNLKEARD NFDRIIKIYK EIEGSQ GENBANK: GENBANK: MDVT00000000.1 |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 6.8 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.9 構成要素:
詳細: 100 mM PIPES, pH 6.9, 0.5 mM EGTA, 0.5 mM MgSO4, 10% (v/v) glycerol and polymerized with 2 mM GTP | |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: MOLYBDENUM / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 前処理 - 雰囲気: AIR / 詳細: High vacuum | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 298 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 1217 / 平均露光時間: 2.42 sec. / 平均電子線量: 40.05 e/Å2 詳細: Images were recorded using a Falcon3 detector (FEI) at a pixel size of 1.45 angstrom per pixel and a frame rate of 60 frames/individual images |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)