+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of GH31 alpha-1,3-glucosidase from Lactococcus lactis subsp. cremoris | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報加水分解酵素; 糖加水分解酵素; 配糖体結合加水分解酵素または糖加水分解酵素 / hydrolase activity, hydrolyzing O-glycosyl compounds / carbohydrate metabolic process 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lactococcus lactis subsp. cremoris MG1363 (乳酸菌) Lactococcus lactis subsp. cremoris MG1363 (乳酸菌) | |||||||||
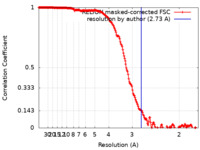
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.73 Å | |||||||||
 データ登録者 データ登録者 | Ikegaya M / Moriya T / Adachi N / Kawasaki M / Park EY / Miyazaki T | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2022 ジャーナル: J Biol Chem / 年: 2022タイトル: Structural basis of the strict specificity of a bacterial GH31 α-1,3-glucosidase for nigerooligosaccharides. 著者: Marina Ikegaya / Toshio Moriya / Naruhiko Adachi / Masato Kawasaki / Enoch Y Park / Takatsugu Miyazaki /  要旨: Carbohydrate-active enzymes are involved in the degradation, biosynthesis, and modification of carbohydrates and vary with the diversity of carbohydrates. The glycoside hydrolase (GH) family 31 is ...Carbohydrate-active enzymes are involved in the degradation, biosynthesis, and modification of carbohydrates and vary with the diversity of carbohydrates. The glycoside hydrolase (GH) family 31 is one of the most diverse families of carbohydrate-active enzymes, containing various enzymes that act on α-glycosides. However, the function of some GH31 groups remains unknown, as their enzymatic activity is difficult to estimate due to the low amino acid sequence similarity between characterized and uncharacterized members. Here, we performed a phylogenetic analysis and discovered a protein cluster (GH31_u1) sharing low sequence similarity with the reported GH31 enzymes. Within this cluster, we showed that a GH31_u1 protein from Lactococcus lactis (LlGH31_u1) and its fungal homolog demonstrated hydrolytic activities against nigerose [α-D-Glcp-(1→3)-D-Glc]. The k/K values of LlGH31_u1 against kojibiose and maltose were 13% and 2.1% of that against nigerose, indicating that LlGH31_u1 has a higher specificity to the α-1,3 linkage of nigerose than other characterized GH31 enzymes, including eukaryotic enzymes. Furthermore, the three-dimensional structures of LlGH31_u1 determined using X-ray crystallography and cryogenic electron microscopy revealed that LlGH31_u1 forms a hexamer and has a C-terminal domain comprising four α-helices, suggesting that it contributes to hexamerization. Finally, crystal structures in complex with nigerooligosaccharides and kojibiose along with mutational analysis revealed the active site residues involved in substrate recognition in this enzyme. This study reports the first structure of a bacterial GH31 α-1,3-glucosidase and provides new insight into the substrate specificity of GH31 enzymes and the physiological functions of bacterial and fungal GH31_u1 members. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32571.map.gz emd_32571.map.gz | 478 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32571-v30.xml emd-32571-v30.xml emd-32571.xml emd-32571.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32571_fsc.xml emd_32571_fsc.xml | 18.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
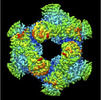
| 画像 |  emd_32571.png emd_32571.png | 206.9 KB | ||
| マスクデータ |  emd_32571_msk_1.map emd_32571_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| その他 |  emd_32571_half_map_1.map.gz emd_32571_half_map_1.map.gz emd_32571_half_map_2.map.gz emd_32571_half_map_2.map.gz | 412.4 MB 412.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32571 http://ftp.pdbj.org/pub/emdb/structures/EMD-32571 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32571 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32571 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32571_validation.pdf.gz emd_32571_validation.pdf.gz | 869.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32571_full_validation.pdf.gz emd_32571_full_validation.pdf.gz | 869 KB | 表示 | |
| XML形式データ |  emd_32571_validation.xml.gz emd_32571_validation.xml.gz | 25.5 KB | 表示 | |
| CIF形式データ |  emd_32571_validation.cif.gz emd_32571_validation.cif.gz | 33.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32571 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32571 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32571 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32571 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7wlgMC  7wj9C  7wjaC  7wjbC  7wjcC  7wjdC  7wjeC  7wjfC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-11171 (タイトル: Cryo-EM structure of GH31 alpha-1,3-glucosidase from Lactococcus lactis subsp. cremoris EMPIAR-11171 (タイトル: Cryo-EM structure of GH31 alpha-1,3-glucosidase from Lactococcus lactis subsp. cremorisData size: 911.2 Data #1: Cryo-EM structure of GH31 alpha-1,3-glucosidase from Lactococcus lactis subsp. cremoris [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32571.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32571.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_32571_msk_1.map emd_32571_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_32571_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_32571_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LlGH31_u1 hexamer
| 全体 | 名称: LlGH31_u1 hexamer |
|---|---|
| 要素 |
|
-超分子 #1: LlGH31_u1 hexamer
| 超分子 | 名称: LlGH31_u1 hexamer / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: homo hexamer |
|---|---|
| 由来(天然) | 生物種:  Lactococcus lactis subsp. cremoris MG1363 (乳酸菌) Lactococcus lactis subsp. cremoris MG1363 (乳酸菌) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 516 KDa |
-分子 #1: LlGH31_u1
| 分子 | 名称: LlGH31_u1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: ec: 3.2.1.84 |
|---|---|
| 由来(天然) | 生物種:  Lactococcus lactis subsp. cremoris MG1363 (乳酸菌) Lactococcus lactis subsp. cremoris MG1363 (乳酸菌)株: MG1363 |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MVSELESKYM NNNIIKFDKA RFTVLTEHLI RIEYSETGEF EERMTQMVQN REFSEVNFDI IEKEETIEII TSTVHLYYNG GEFTNASLFA DVKFNFSVYS NRWYFGEKSD GNLKGTTRTL DMIDGECPLE DGIMSKNGFA VLADKGKVLT ...文字列: MGSSHHHHHH SSGLVPRGSH MVSELESKYM NNNIIKFDKA RFTVLTEHLI RIEYSETGEF EERMTQMVQN REFSEVNFDI IEKEETIEII TSTVHLYYNG GEFTNASLFA DVKFNFSVYS NRWYFGEKSD GNLKGTTRTL DMIDGECPLE DGIMSKNGFA VLADKGKVLT EVGDIAGNSV STIDLYLFAY GRDYRQALKD FYQLTGNTPK LPRFALGNWW SRYYDYSDKS YLALMDKFTD KKVPLSVSVI DMDWHKVSEV PSRFGSGWTG YSWNKKLFPN PENFIDELHQ RKLKVTLNDH PADGIRAFED PYPQVAQTLD LNTELEEAAK FDFDNLKFRK AYFEEVHGPL EKEGVDFWWI DWQQGAISKS GVDPLWLLNH YQYQNAQKKH KNNIILSRYA GPGSHRYPLG FSGDSVISWA SLDFQPYFTS TASNIGYTWW SHDIGGHMQG YKDAELSLRW LQFGVFSPIN RLHSSKSEFT SKEPWHFDAV IEQSMIDFLQ LRHQLIPYLY SANLITASEG RALVEPLYYE YPMEEEAYQH RNQYLFGEQL MVAPITEKMN SLLQMGSVEV WFPEGTWYDF FSGQPYDGKV SLKVYREITE MPVFAKAGAI IPLDKNPLKK EEIPSEIIWK IFPGADGEYL LLEEDNETKA EFVNGIFTVT SKKESSRKHT IIYGEHEIVS AKRGEFSIDL NGKEENFDWN FSTALFRRLD IAEISYEQKD EILQQLSLIE EHEKQVAFIK TNENQELQNS LFELLYSGK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.58 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: The grid was washed by acetone prior to use. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting time was 5 seconds (blot force 15). | |||||||||
| 詳細 | This sample was mono-disperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 995 / 平均露光時間: 65.95 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 120000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)