+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SaCas9-sgRNA-DNA ternary complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
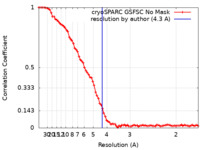
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Du WH / Huang Q / Zhu HX / Xue DM / Zheng S | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2023 ジャーナル: Int J Mol Sci / 年: 2023タイトル: Full-Length Model of SaCas9-sgRNA-DNA Complex in Cleavage State. 著者: Wenhao Du / Haixia Zhu / Jiaqiang Qian / Dongmei Xue / Sen Zheng / Qiang Huang /  要旨: Cas9 (SaCas9) is a widely used genome editing tool. Understanding its molecular mechanisms of DNA cleavage could effectively guide the engineering optimization of this system. Here, we determined ... Cas9 (SaCas9) is a widely used genome editing tool. Understanding its molecular mechanisms of DNA cleavage could effectively guide the engineering optimization of this system. Here, we determined the first cryo-electron microscopy structure of the SaCas9-sgRNA-DNA ternary complex. This structure reveals that the HNH nuclease domain is tightly bound to the cleavage site of the target DNA strand, and is in close contact with the WED and REC domains. Moreover, it captures the complete structure of the sgRNA, including the previously unresolved stem-loop 2. Based on this structure, we build a full-length model for the ternary complex in cleavage state. This model enables identification of the residues for the interactions between the HNH domain and the WED and REC domains. Moreover, we found that the stem-loop 2 of the sgRNA tightly binds to the PI and RuvC domains and may also regulate the position shift of the RuvC domain. Further mutagenesis and molecular dynamics simulations supported the idea that the interactions of the HNH domain with the WED and REC domains play an important role in the DNA cleavage. Thus, this study provides new mechanistic insights into the DNA cleavage of SaCas9 and is also useful for guiding the future engineering of SaCas9-mediated gene editing systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32104.map.gz emd_32104.map.gz | 45.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32104-v30.xml emd-32104-v30.xml emd-32104.xml emd-32104.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32104_fsc.xml emd_32104_fsc.xml | 8.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32104.png emd_32104.png | 88.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32104 http://ftp.pdbj.org/pub/emdb/structures/EMD-32104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32104 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32104_validation.pdf.gz emd_32104_validation.pdf.gz | 469.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32104_full_validation.pdf.gz emd_32104_full_validation.pdf.gz | 469.5 KB | 表示 | |
| XML形式データ |  emd_32104_validation.xml.gz emd_32104_validation.xml.gz | 10 KB | 表示 | |
| CIF形式データ |  emd_32104_validation.cif.gz emd_32104_validation.cif.gz | 12.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32104 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32104 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32104 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32104 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7vw3MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32104.map.gz / 形式: CCP4 / 大きさ: 48.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32104.map.gz / 形式: CCP4 / 大きさ: 48.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SaCas9-sgRNA-target DNA ternary complex
| 全体 | 名称: SaCas9-sgRNA-target DNA ternary complex |
|---|---|
| 要素 |
|
-超分子 #1: SaCas9-sgRNA-target DNA ternary complex
| 超分子 | 名称: SaCas9-sgRNA-target DNA ternary complex / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: SaCas9
| 超分子 | 名称: SaCas9 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|
-超分子 #3: sgRNA-target DNA
| 超分子 | 名称: sgRNA-target DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#4 |
|---|
-分子 #1: SaCas9
| 分子 | 名称: SaCas9 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MKRNYILGLA IGITSVGYGI IDYETRDVID AGVRLFKEAN VENNEGRRSK RGARRLKRRR RHRIQRVKKL LFDYNLLTDH SELSGINPYE ARVKGLSQKL SEEEFSAALL HLAKRRGVHN VNEVEEDTGN ELSTKEQISR NSKALEEKYV ...文字列: MGSSHHHHHH SSGLVPRGSH MKRNYILGLA IGITSVGYGI IDYETRDVID AGVRLFKEAN VENNEGRRSK RGARRLKRRR RHRIQRVKKL LFDYNLLTDH SELSGINPYE ARVKGLSQKL SEEEFSAALL HLAKRRGVHN VNEVEEDTGN ELSTKEQISR NSKALEEKYV AELQLERLKK DGEVRGSINR FKTSDYVKEA KQLLKVQKAY HQLDQSFIDT YIDLLETRRT YYEGPGEGSP FGWKDIKEWY EMLMGHCTYF PEELRSVKYA YNADLYNALN DLNNLVITRD ENEKLEYYEK FQIIENVFKQ KKKPTLKQIA KEILVNEEDI KGYRVTSTGK PEFTNLKVYH DIKDITARKE IIENAELLDQ IAKILTIYQS SEDIQEELTN LNSELTQEEI EQISNLKGYT GTHNLSLKAI NLILDELWHT NDNQIAIFNR LKLVPKKVDL SQQKEIPTTL VDDFILSPVV KRSFIQSIKV INAIIKKYGL PNDIIIELAR EKNSKDAQKM INEMQKRNRQ TNERIEEIIR TTGKENAKYL IEKIKLHDMQ EGKCLYSLEA IPLEDLLNNP FNYEVDHIIP RSVSFDNSFN NKVLVKQEEA SKKGNRTPFQ YLSSSDSKIS YETFKKHILN LAKGKGRISK TKKEYLLEER DINRFSVQKD FINRNLVDTR YATRGLMNLL RSYFRVNNLD VKVKSINGGF TSFLRRKWKF KKERNKGYKH HAEDALIIAN ADFIFKEWKK LDKAKKVMEN QMFEEKQAES MPEIETEQEY KEIFITPHQI KHIKDFKDYK YSHRVDKKPN RELINDTLYS TRKDDKGNTL IVNNLNGLYD KDNDKLKKLI NKSPEKLLMY HHDPQTYQKL KLIMEQYGDE KNPLYKYYEE TGNYLTKYSK KDNGPVIKKI KYYGNKLNAH LDITDDYPNS RNKVVKLSLK PYRFDVYLDN GVYKFVTVKN LDVIKKENYY EVNSKCYEEA KKLKKISNQA EFIASFYNND LIKINGELYR VIGVNNDLLN RIEVNMIDIT YREYLENMND KRPPRIIKTI ASKTQSIKKY STDILGNLYE VKSKKHPQII KKG |
-分子 #2: single-guide RNA (sgRNA)
| 分子 | 名称: single-guide RNA (sgRNA) / タイプ: rna / ID: 2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: GGUACCGCUC CAGUCGUUCA UGGUUUUAGU ACUCUGGAAA CAGAAUCUAC UAAAACAAGG CAAAAUGCCG UGUUUAUCUC GUCAACUUGU UGGCGAGAUU UUUUU |
-分子 #3: Target DNA strand(TS)
| 分子 | 名称: Target DNA strand(TS) / タイプ: dna / ID: 3 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: TCCAGAGTAC TAAAACATTC AACATGAACG ACTGGAGCGG TACTATAGTG AGTCGTATT |
-分子 #4: Non-target DNA strand (NTS)
| 分子 | 名称: Non-target DNA strand (NTS) / タイプ: dna / ID: 4 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: AATACGACTC ACTATAGTAC CGCTCCAGTC GTTCATGTTG AATGTTTTAG TACTCTGGA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.22 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 20mM Tris-Cl (pH 7.5), 100mM KCl, 5mM MgCl2, 1mM DTT | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | blot for 4 seconds before pluging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 10389 / 平均電子線量: 38.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)