+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rba sphaeroides PufX-KO RC-LH1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Photosystem / Complex / PHOTOSYNTHESIS / Mutant | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / photosynthesis, light reaction / : / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rhodobacter sphaeroides 2.4.1 (バクテリア) / Rhodobacter sphaeroides 2.4.1 (バクテリア) /  Cereibacter sphaeroides 2.4.1 (バクテリア) Cereibacter sphaeroides 2.4.1 (バクテリア) | |||||||||
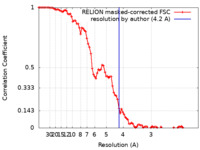
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Bracun L / Yamagata A | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis for the assembly and quinone transport mechanisms of the dimeric photosynthetic RC-LH1 supercomplex. 著者: Peng Cao / Laura Bracun / Atsushi Yamagata / Bern M Christianson / Tatsuki Negami / Baohua Zou / Tohru Terada / Daniel P Canniffe / Mikako Shirouzu / Mei Li / Lu-Ning Liu /    要旨: The reaction center (RC) and light-harvesting complex 1 (LH1) form a RC-LH1 core supercomplex that is vital for the primary reactions of photosynthesis in purple phototrophic bacteria. Some species ...The reaction center (RC) and light-harvesting complex 1 (LH1) form a RC-LH1 core supercomplex that is vital for the primary reactions of photosynthesis in purple phototrophic bacteria. Some species possess the dimeric RC-LH1 complex with a transmembrane polypeptide PufX, representing the largest photosynthetic complex in anoxygenic phototrophs. However, the details of the architecture and assembly mechanism of the RC-LH1 dimer are unclear. Here we report seven cryo-electron microscopy (cryo-EM) structures of RC-LH1 supercomplexes from Rhodobacter sphaeroides. Our structures reveal that two PufX polypeptides are positioned in the center of the S-shaped RC-LH1 dimer, interlocking association between the components and mediating RC-LH1 dimerization. Moreover, we identify another transmembrane peptide, designated PufY, which is located between the RC and LH1 subunits near the LH1 opening. PufY binds a quinone molecule and prevents LH1 subunits from completely encircling the RC, creating a channel for quinone/quinol exchange. Genetic mutagenesis, cryo-EM structures, and computational simulations provide a mechanistic understanding of the assembly and electron transport pathways of the RC-LH1 dimer and elucidate the roles of individual components in ensuring the structural and functional integrity of the photosynthetic supercomplex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32062.map.gz emd_32062.map.gz | 55.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32062-v30.xml emd-32062-v30.xml emd-32062.xml emd-32062.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32062_fsc.xml emd_32062_fsc.xml | 9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32062.png emd_32062.png | 76.1 KB | ||
| Filedesc metadata |  emd-32062.cif.gz emd-32062.cif.gz | 5.9 KB | ||
| その他 |  emd_32062_additional_1.map.gz emd_32062_additional_1.map.gz | 46.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32062 http://ftp.pdbj.org/pub/emdb/structures/EMD-32062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32062 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32062_validation.pdf.gz emd_32062_validation.pdf.gz | 697.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32062_full_validation.pdf.gz emd_32062_full_validation.pdf.gz | 697.3 KB | 表示 | |
| XML形式データ |  emd_32062_validation.xml.gz emd_32062_validation.xml.gz | 10.6 KB | 表示 | |
| CIF形式データ |  emd_32062_validation.cif.gz emd_32062_validation.cif.gz | 14 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32062 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32062 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32062 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32062 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7voyMC  7va9C  7vb9C  7vnmC  7vnyC  7vorC  7votC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32062.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32062.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_32062_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Rhodobacter sphaeroides PufX-KO RC-LH1
+超分子 #1: Rhodobacter sphaeroides PufX-KO RC-LH1
+分子 #1: Light-harvesting protein B-875 alpha chain
+分子 #2: Light-harvesting protein B-875 beta chain
+分子 #3: Reaction center protein L chain
+分子 #4: Reaction center protein M chain
+分子 #5: Reaction center protein H chain
+分子 #6: BACTERIOCHLOROPHYLL A
+分子 #7: BACTERIOPHEOPHYTIN A
+分子 #8: UBIQUINONE-10
+分子 #9: FE (II) ION
+分子 #10: SPEROIDENONE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 46.549 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)