+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Cas12c2-sgRNA-target DNA ternary complex | |||||||||
 マップデータ マップデータ | postprocess_masked.mrc from Relion | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cas12c / c2c3 / sgRNA / target DNA / CRISPR / RNA binding protein-RNA-DNA complex | |||||||||
| 生物種 |  uncultured archaeon (環境試料) / synthetic construct (人工物) uncultured archaeon (環境試料) / synthetic construct (人工物) | |||||||||
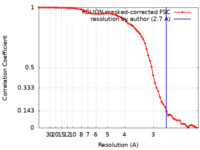
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Kurihara N / Hirano H / Tomita A / Kobayashi K | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: Structure of the type V-C CRISPR-Cas effector enzyme. 著者: Nina Kurihara / Ryoya Nakagawa / Hisato Hirano / Sae Okazaki / Atsuhiro Tomita / Kan Kobayashi / Tsukasa Kusakizako / Tomohiro Nishizawa / Keitaro Yamashita / David A Scott / Hiroshi Nishimasu / Osamu Nureki /    要旨: RNA-guided CRISPR-Cas nucleases are widely used as versatile genome-engineering tools. Recent studies identified functionally divergent type V Cas12 family enzymes. Among them, Cas12c2 binds a CRISPR ...RNA-guided CRISPR-Cas nucleases are widely used as versatile genome-engineering tools. Recent studies identified functionally divergent type V Cas12 family enzymes. Among them, Cas12c2 binds a CRISPR RNA (crRNA) and a trans-activating crRNA (tracrRNA) and recognizes double-stranded DNA targets with a short TN PAM. Here, we report the cryo-electron microscopy structures of the Cas12c2-guide RNA binary complex and the Cas12c2-guide RNA-target DNA ternary complex. The structures revealed that the crRNA and tracrRNA form an unexpected X-junction architecture, and that Cas12c2 recognizes a single T nucleotide in the PAM through specific hydrogen-bonding interactions with two arginine residues. Furthermore, our biochemical analyses indicated that Cas12c2 processes its precursor crRNA to a mature crRNA using the RuvC catalytic site through a unique mechanism. Collectively, our findings improve the mechanistic understanding of diverse type V CRISPR-Cas effectors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31808.map.gz emd_31808.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31808-v30.xml emd-31808-v30.xml emd-31808.xml emd-31808.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31808_fsc.xml emd_31808_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31808.png emd_31808.png | 116.2 KB | ||
| マスクデータ |  emd_31808_msk_1.map emd_31808_msk_1.map | 27 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-31808.cif.gz emd-31808.cif.gz | 7.1 KB | ||
| その他 |  emd_31808_additional_1.map.gz emd_31808_additional_1.map.gz emd_31808_half_map_1.map.gz emd_31808_half_map_1.map.gz emd_31808_half_map_2.map.gz emd_31808_half_map_2.map.gz | 23.5 MB 20.7 MB 20.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31808 http://ftp.pdbj.org/pub/emdb/structures/EMD-31808 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31808 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31808 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31808_validation.pdf.gz emd_31808_validation.pdf.gz | 744.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31808_full_validation.pdf.gz emd_31808_full_validation.pdf.gz | 744.2 KB | 表示 | |
| XML形式データ |  emd_31808_validation.xml.gz emd_31808_validation.xml.gz | 12.5 KB | 表示 | |
| CIF形式データ |  emd_31808_validation.cif.gz emd_31808_validation.cif.gz | 17.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31808 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31808 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31808 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31808 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31808.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31808.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocess_masked.mrc from Relion | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08073 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_31808_msk_1.map emd_31808_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
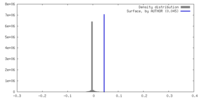
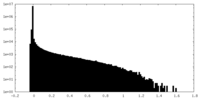
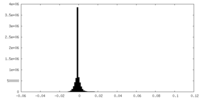
| 密度ヒストグラム |
-追加マップ: calculated by deepEMhancer using run class001.mrc from Refine3D
| ファイル | emd_31808_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | calculated by deepEMhancer using run_class001.mrc from Refine3D | ||||||||||||
| 投影像・断面図 |
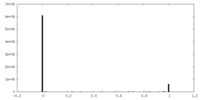
| ||||||||||||
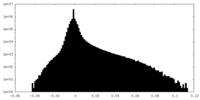
| 密度ヒストグラム |
-ハーフマップ: run half1 class001 unfil.mrc from Relion
| ファイル | emd_31808_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | run_half1_class001_unfil.mrc from Relion | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
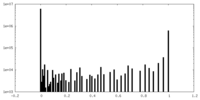
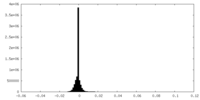
| 密度ヒストグラム |
-ハーフマップ: run half2 class001 unfil.mrc from Relion
| ファイル | emd_31808_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | run_half2_class001_unfil.mrc from Relion | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
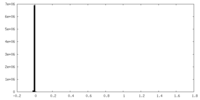
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cas12c2-sgRNA-DNA complex
| 全体 | 名称: Cas12c2-sgRNA-DNA complex |
|---|---|
| 要素 |
|
-超分子 #1: Cas12c2-sgRNA-DNA complex
| 超分子 | 名称: Cas12c2-sgRNA-DNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  uncultured archaeon (環境試料) uncultured archaeon (環境試料) |
-超分子 #2: Cas12c2
| 超分子 | 名称: Cas12c2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  uncultured archaeon (環境試料) uncultured archaeon (環境試料) |
-超分子 #3: sgRNA
| 超分子 | 名称: sgRNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-超分子 #4: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|
-分子 #1: Cas12c2
| 分子 | 名称: Cas12c2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured archaeon (環境試料) uncultured archaeon (環境試料) |
| 分子量 | 理論値: 138.348938 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKIEEGKGHH HHHHMTKHSI PLHAFRNSGA DARKWKGRIA LLAKRGKETM RTLQFPLEMS EPEAAAINTT PFAVAYNAIE GTGKGTLFD YWAKLHLAGF RFFPSGGAAT IFRQQAVFED ASWNAAFCQQ SGKDWPWLVP SKLYERFTKA PREVAKKDGS K KSIEFTQE ...文字列: MKIEEGKGHH HHHHMTKHSI PLHAFRNSGA DARKWKGRIA LLAKRGKETM RTLQFPLEMS EPEAAAINTT PFAVAYNAIE GTGKGTLFD YWAKLHLAGF RFFPSGGAAT IFRQQAVFED ASWNAAFCQQ SGKDWPWLVP SKLYERFTKA PREVAKKDGS K KSIEFTQE NVANESHVSL VGASITDKTP EDQKEFFLKM AGALAEKFDS WKSANEDRIV AMKVIDEFLK SEGLHLPSLE NI AVKCSVE TKPDNATVAW HDAPMSGVQN LAIGVFATCA SRIDNIYDLN GGKLSKLIQE SATTPNVTAL SWLFGKGLEY FRT TDIDTI MQDFNIPASA KESIKPLVES AQAIPTMTVL GKKNYAPFRP NFGGKIDSWI ANYASRLMLL NDILEQIEPG FELP QALLD NETLMSGIDM TGDELKELIE AVYAWVDAAK QGLATLLGRG GNVDDAVQTF EQFSAMMDTL NGTLNTISAR YVRAV EMAG KDEARLEKLI ECKFDIPKWC KSVPKLVGIS GGLPKVEEEI KVMNAAFKDV RARMFVRFEE IAAYVASKGA GMDVYD ALE KRELEQIKKL KSAVPERAHI QAYRAVLHRI GRAVQNCSEK TKQLFSSKVI EMGVFKNPSH LNNFIFNQKG AIYRSPF DR SRHAPYQLHA DKLLKNDWLE LLAEISATLM ASESTEQMED ALRLERTRLQ LQLSGLPDWE YPASLAKPDI EVEIQTAL K MQLAKDTVTS DVLQRAFNLY SSVLSGLTFK LLRRSFSLKM RFSVADTTQL IYVPKVCDWA IPKQYLQAEG EIGIAARVV TESSPAKMVT EVEMKEPKAL GHFMQQAPHD WYFDASLGGT QVAGRIVEKG KEVGKERKLV GYRMRGNSAY KTVLDKSLVG NTELSQCSM IIEIPYTQTV DADFRAQVQA GLPKVSINLP VKETITASNK DEQMLFDRFV AIDLGERGLG YAVFDAKTLE L QESGHRPI KAITNLLNRT HHYEQRPNQR QKFQAKFNVN LSELRENTVG DVCHQINRIC AYYNAFPVLE YMVPDRLDKQ LK SVYESVT NRYIWSSTDA HKSARVQFWL GGETWEHPYL KSAKDKKPLV LSPGRGASGK GTSQTCSCCG RNPFDLIKDM KPR AKIAVV DGKAKLENSE LKLFERNLES KDDMLARRHR NERAGMEQPL TPGNYTVDEI KALLRANLRR APKNRRTKDT TVSE YHCVF SDCGKTMHAD ENAAVNIGGK FIADIEK |
-分子 #2: sgRNA
| 分子 | 名称: sgRNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  uncultured archaeon (環境試料) uncultured archaeon (環境試料) |
| 分子量 | 理論値: 36.147414 KDa |
| 配列 | 文字列: GGAUACCACC CGUGCAUUUC UGGAUCAAUG AUCCGUACCU CAAUGUCCGG GCGCGCAGCU AGAGCGACCU GAAGAAAUUC AGGUUGGGU UUGAGGGGAA AUUAGGUGCG CUU |
-分子 #3: target DNA (target strand)
| 分子 | 名称: target DNA (target strand) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.080494 KDa |
| 配列 | 文字列: (DA)(DG)(DG)(DT)(DT)(DG)(DC)(DC)(DA)(DA) (DG)(DC)(DG)(DC)(DA)(DC)(DC)(DT)(DA)(DA) (DT)(DT)(DT)(DC)(DC)(DC)(DA)(DT)(DT) (DT)(DT)(DA)(DG) |
-分子 #4: target DNA (non target strand)
| 分子 | 名称: target DNA (non target strand) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.218596 KDa |
| 配列 | 文字列: (DC)(DT)(DA)(DA)(DA)(DA)(DT)(DG)(DG)(DG) (DA)(DA)(DA)(DT)(DT)(DA)(DG)(DG)(DT)(DG) (DC)(DG)(DC)(DT)(DT)(DG)(DG)(DC)(DA) (DA)(DC)(DC)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 2.6 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7v94: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)