+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
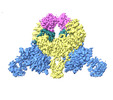
| タイトル | CryoEM structure of DDB1-VprBP-Vpr-UNG2(94-313) complex | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | E3 ligase / HIV accessory protein / restriction factors / LIGASE / TRANSFERASE-DNA BINDING PROTEIN-VIRAL PROTEIN complex | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cell competition in a multicellular organism / histone H2AT120 kinase activity / symbiont-mediated arrest of host cell cycle during G2/M transition / base-excision repair, AP site formation via deaminated base removal / uracil-DNA glycosylase / depyrimidination / Displacement of DNA glycosylase by APEX1 / single strand break repair / V(D)J recombination / positive regulation by virus of viral protein levels in host cell ...cell competition in a multicellular organism / histone H2AT120 kinase activity / symbiont-mediated arrest of host cell cycle during G2/M transition / base-excision repair, AP site formation via deaminated base removal / uracil-DNA glycosylase / depyrimidination / Displacement of DNA glycosylase by APEX1 / single strand break repair / V(D)J recombination / positive regulation by virus of viral protein levels in host cell / isotype switching / spindle assembly involved in female meiosis / uracil DNA N-glycosylase activity / epigenetic programming in the zygotic pronuclei / UV-damage excision repair / biological process involved in interaction with symbiont / regulation of mitotic cell cycle phase transition / WD40-repeat domain binding / Cul4A-RING E3 ubiquitin ligase complex / Cul4-RING E3 ubiquitin ligase complex / Cul4B-RING E3 ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / negative regulation of reproductive process / negative regulation of developmental process / cullin family protein binding / viral release from host cell / ribosomal small subunit binding / ectopic germ cell programmed cell death / positive regulation of viral genome replication / ubiquitin-like ligase-substrate adaptor activity / proteasomal protein catabolic process / somatic hypermutation of immunoglobulin genes / positive regulation of gluconeogenesis / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / post-translational protein modification / B cell differentiation / Chromatin modifications during the maternal to zygotic transition (MZT) / Recognition of DNA damage by PCNA-containing replication complex / nuclear estrogen receptor binding / DNA Damage Recognition in GG-NER / nucleotide-excision repair / symbiont-mediated activation of host apoptosis / Dual Incision in GG-NER / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex / Formation of Incision Complex in GG-NER / regulation of circadian rhythm / base-excision repair / protein homooligomerization / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / virion component / viral penetration into host nucleus / Wnt signaling pathway / fibrillar center / positive regulation of protein catabolic process / Antigen processing: Ubiquitination & Proteasome degradation / cellular response to UV / rhythmic process / site of double-strand break / Neddylation / host cell / ubiquitin-dependent protein catabolic process / monoatomic ion transmembrane transport / protein-macromolecule adaptor activity / damaged DNA binding / host extracellular space / proteasome-mediated ubiquitin-dependent protein catabolic process / chromosome, telomeric region / non-specific serine/threonine protein kinase / protein ubiquitination / protein serine kinase activity / DNA repair / apoptotic process / DNA-templated transcription / centrosome / DNA damage response / symbiont entry into host cell / negative regulation of apoptotic process / regulation of DNA-templated transcription / protein-containing complex binding / nucleolus / host cell nucleus / negative regulation of transcription by RNA polymerase II / protein-containing complex / mitochondrion / extracellular space / DNA binding / extracellular exosome / nucleoplasm / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Wang D / Xu J | ||||||||||||||||||
| 資金援助 |  中国, 5件 中国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural insights into the HIV-1 Vpr mediated ubiquitination through the Cullin-RING E3 ubiquitin ligase 著者: Wang D / Xu J / Liu Q / Xiang Y | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31766.map.gz emd_31766.map.gz | 111.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31766-v30.xml emd-31766-v30.xml emd-31766.xml emd-31766.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31766.png emd_31766.png | 118.7 KB | ||
| Filedesc metadata |  emd-31766.cif.gz emd-31766.cif.gz | 7.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31766 http://ftp.pdbj.org/pub/emdb/structures/EMD-31766 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31766 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31766 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31766_validation.pdf.gz emd_31766_validation.pdf.gz | 465.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31766_full_validation.pdf.gz emd_31766_full_validation.pdf.gz | 464.9 KB | 表示 | |
| XML形式データ |  emd_31766_validation.xml.gz emd_31766_validation.xml.gz | 6.7 KB | 表示 | |
| CIF形式データ |  emd_31766_validation.cif.gz emd_31766_validation.cif.gz | 7.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31766 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31766 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31766 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31766 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7v7cMC  7v7bC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31766.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31766.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0742 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DDB1-VprBP-Vpr-UNG2(94-313) complex
| 全体 | 名称: DDB1-VprBP-Vpr-UNG2(94-313) complex |
|---|---|
| 要素 |
|
-超分子 #1: DDB1-VprBP-Vpr-UNG2(94-313) complex
| 超分子 | 名称: DDB1-VprBP-Vpr-UNG2(94-313) complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 0.66 kDa/nm |
-分子 #1: DDB1- and CUL4-associated factor 1
| 分子 | 名称: DDB1- and CUL4-associated factor 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 169.194781 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MTTVVVHVDS KAELTTLLEQ WEKEHGSGQD MVPILTRMSQ LIEKETEEYR KGDPDPFDDR HPGRADPECM LGHLLRILFK NDDFMNALV NAYVMTSREP PLNTAACRLL LDIMPGLETA VVFQEKEGIV ENLFKWAREA DQPLRTYSTG LLGGAMENQD I AANYRDEN ...文字列: MTTVVVHVDS KAELTTLLEQ WEKEHGSGQD MVPILTRMSQ LIEKETEEYR KGDPDPFDDR HPGRADPECM LGHLLRILFK NDDFMNALV NAYVMTSREP PLNTAACRLL LDIMPGLETA VVFQEKEGIV ENLFKWAREA DQPLRTYSTG LLGGAMENQD I AANYRDEN SQLVAIVLRR LRELQLQEVA LRQENKRPSP RKLSSEPLLP LDEEAVDMDY GDMAVDVVDG DQEEASGDME IS FHLDSGH KTSSRVNSTT KPEDGGLKKN KSAKQGDREN FRKAKQKLGF SSSDPDRMFV ELSNSSWSEM SPWVIGTNYT LYP MTPAIE QRLILQYLTP LGEYQELLPI FMQLGSRELM MFYIDLKQTN DVLLTFEALK HLASLLLHNK FATEFVAHGG VQKL LEIPR PSMAATGVSM CLYYLSYNQD AMERVCMHPH NVLSDVVNYT LWLMECSHAS GCCHATMFFS ICFSFRAVLE LFDRY DGLR RLVNLISTLE ILNLEDQGAL LSDDEIFASR QTGKHTCMAL RKYFEAHLAI KLEQVKQSLQ RTEGGILVHP QPPYKA CSY THEQIVEMME FLIEYGPAQL YWEPAEVFLK LSCVQLLLQL ISIACNWKTY YARNDTVRFA LDVLAILTVV PKIQLQL AE SVDVLDEAGS TVSTVGISII LGVAEGEFFI HDAEIQKSAL QIIINCVCGP DNRISSIGKF ISGTPRRKLP QNPKSSEH T LAKMWNVVQS NNGIKVLLSL LSIKMPITDA DQIRALACKA LVGLSRSSTV RQIISKLPLF SSCQIQQLMK EPVLQDKRS DHVKFCKYAA ELIERVSGKP LLIGTDVSLA RLQKADVVAQ SRISFPEKEL LLLIRNHLIS KGLGETATVL TKEADLPMTA ASHSSAFTP VTAAASPVSL PRTPRIANGI ATRLGSHAAV GASAPSAPTA HPQPRPPQGP LALPGPSYAG NSPLIGRISF I RERPSPCN GRKIRVLRQK SDHGAYSQSP AIKKQLDRHL PSPPTLDSII TEYLREQHAR CKNPVATCPP FSLFTPHQCP EP KQRRQAP INFTSRLNRR ASFPKYGGVD GGCFDRHLIF SRFRPISVFR EANEDESGFT CCAFSARERF LMLGTCTGQL KLY NVFSGQ EEASYNCHNS AITHLEPSRD GSLLLTSATW SQPLSALWGM KSVFDMKHSF TEDHYVEFSK HSQDRVIGTK GDIA HIYDI QTGNKLLTLF NPDLANNYKR NCATFNPTDD LVLNDGVLWD VRSAQAIHKF DKFNMNISGV FHPNGLEVII NTEIW DLRT FHLLHTVPAL DQCRVVFNHT GTVMYGAMLQ ADDEDDLMEE RMKSPFGSSF RTFNATDYKP IATIDVKRNI FDLCTD TKD CYLAVIENQG SMDALNMDTV CRLYEVGRQR LAEDEDEEED QEEEEQEEED DDEDDDDTDD LDELDTDQLL EAELEED DN NENAGEDGDN DFSPSDEELA NLLEEGEDGE DEDSDADEEV ELILGDTDSS DNSDLEDDII LSLNE UniProtKB: DDB1- and CUL4-associated factor 1 |
-分子 #2: DNA damage-binding protein 1
| 分子 | 名称: DNA damage-binding protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 127.097469 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSYNYVVTAQ KPTAVNGCVT GHFTSAEDLN LLIAKNTRLE IYVVTAEGLR PVKEVGMYGK IAVMELFRPK GESKDLLFIL TAKYNACIL EYKQSGESID IITRAHGNVQ DRIGRPSETG IIGIIDPECR MIGLRLYDGL FKVIPLDRDN KELKAFNIRL E ELHVIDVK ...文字列: MSYNYVVTAQ KPTAVNGCVT GHFTSAEDLN LLIAKNTRLE IYVVTAEGLR PVKEVGMYGK IAVMELFRPK GESKDLLFIL TAKYNACIL EYKQSGESID IITRAHGNVQ DRIGRPSETG IIGIIDPECR MIGLRLYDGL FKVIPLDRDN KELKAFNIRL E ELHVIDVK FLYGCQAPTI CFVYQDPQGR HVKTYEVSLR EKEFNKGPWK QENVEAEASM VIAVPEPFGG AIIIGQESIT YH NGDKYLA IAPPIIKQST IVCHNRVDPN GSRYLLGDME GRLFMLLLEK EEQMDGTVTL KDLRVELLGE TSIAECLTYL DNG VVFVGS RLGDSQLVKL NVDSNEQGSY VVAMETFTNL GPIVDMCVVD LERQGQGQLV TCSGAFKEGS LRIIRNGIGI HEHA SIDLP GIKGLWPLRS DPNRETDDTL VLSFVGQTRV LMLNGEEVEE TELMGFVDDQ QTFFCGNVAH QQLIQITSAS VRLVS QEPK ALVSEWKEPQ AKNISVASCN SSQVVVAVGR ALYYLQIHPQ ELRQISHTEM EHEVACLDIT PLGDSNGLSP LCAIGL WTD ISARILKLPS FELLHKEMLG GEIIPRSILM TTFESSHYLL CALGDGALFY FGLNIETGLL SDRKKVTLGT QPTVLRT FR SLSTTNVFAC SDRPTVIYSS NHKLVFSNVN LKEVNYMCPL NSDGYPDSLA LANNSTLTIG TIDEIQKLHI RTVPLYES P RKICYQEVSQ CFGVLSSRIE VQDTSGGTTA LRPSASTQAL SSSVSSSKLF SSSTAPHETS FGEEVEVHNL LIIDQHTFE VLHAHQFLQN EYALSLVSCK LGKDPNTYFI VGTAMVYPEE AEPKQGRIVV FQYSDGKLQT VAEKEVKGAV YSMVEFNGKL LASINSTVR LYEWTTEKEL RTECNHYNNI MALYLKTKGD FILVGDLMRS VLLLAYKPME GNFEEIARDF NPNWMSAVEI L DDDNFLGA ENAFNLFVCQ KDSAATTDEE RQHLQEVGLF HLGEFVNVFC HGSLVMQNLG ETSTPTQGSV LFGTVNGMIG LV TSLSESW YNLLLDMQNR LNKVIKSVGK IEHSFWRSFH TERKTEPATG FIDGDLIESF LDISRPKMQE VVANLQYDDG SGM KREATA DDLIKVVEEL TRIH UniProtKB: DNA damage-binding protein 1 |
-分子 #3: Protein Vpr
| 分子 | 名称: Protein Vpr / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 11.396878 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEQAPEDQGP QREPYNEWTL ELLEELKSEA VRHFPRIWLH NLGQHIYETY GDTWAGVEAI IRILQQLLFI HFRIGCRHSR IGVTRQRRA RNGASRS UniProtKB: Protein Vpr |
-分子 #4: Uracil-DNA glycosylase
| 分子 | 名称: Uracil-DNA glycosylase / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO / EC番号: uracil-DNA glycosylase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.136654 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: FGESWKKHLS GEFGKPYFIK LMGFVAEERK HYTVYPPPHQ VFTWTQMCDI KDVKVVILGQ DPYHGPNQAH GLCFSVQRPV PPPPSLENI YKELSTDIED FVHPGHGDLS GWAKQGVLLL NAVLTVRAHQ ANSHKERGWE QFTDAVVSWL NQNSNGLVFL L WGSYAQKK ...文字列: FGESWKKHLS GEFGKPYFIK LMGFVAEERK HYTVYPPPHQ VFTWTQMCDI KDVKVVILGQ DPYHGPNQAH GLCFSVQRPV PPPPSLENI YKELSTDIED FVHPGHGDLS GWAKQGVLLL NAVLTVRAHQ ANSHKERGWE QFTDAVVSWL NQNSNGLVFL L WGSYAQKK GSAIDRKRHH VLQTAHPSPL SVYRGFFGCR HFSKTNELLQ KSGKKPIDWK EL UniProtKB: Uracil-DNA glycosylase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 145568 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7v7c: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















