+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30592 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural insights into membrane remodeling by SNX1 | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Coat complex / Membrane deformation / LIPID BINDING PROTEIN / helical assembly / PROTEIN TRANSPORT | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報retromer, tubulation complex / lamellipodium morphogenesis / leptin receptor binding / early endosome to Golgi transport / transferrin receptor binding / epidermal growth factor receptor binding / phosphatidylinositol binding / insulin receptor binding / intracellular protein transport / receptor internalization ...retromer, tubulation complex / lamellipodium morphogenesis / leptin receptor binding / early endosome to Golgi transport / transferrin receptor binding / epidermal growth factor receptor binding / phosphatidylinositol binding / insulin receptor binding / intracellular protein transport / receptor internalization / positive regulation of protein catabolic process / lamellipodium / early endosome membrane / lysosome / protein heterodimerization activity / Golgi apparatus / protein homodimerization activity / cytosol 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
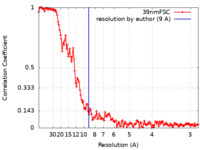
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 9.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Zhang Y / Pang X | ||||||||||||||||||
| 資金援助 |  中国, 5件 中国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Structural insights into membrane remodeling by SNX1. 著者: Yan Zhang / Xiaoyun Pang / Jian Li / Jiashu Xu / Victor W Hsu / Fei Sun /   要旨: The sorting nexin (SNX) family of proteins deform the membrane to generate transport carriers in endosomal pathways. Here, we elucidate how a prototypic member, SNX1, acts in this process. Performing ...The sorting nexin (SNX) family of proteins deform the membrane to generate transport carriers in endosomal pathways. Here, we elucidate how a prototypic member, SNX1, acts in this process. Performing cryoelectron microscopy, we find that SNX1 assembles into a protein lattice that consists of helical rows of SNX1 dimers wrapped around tubular membranes in a crosslinked fashion. We also visualize the details of this structure, which provides a molecular understanding of how various parts of SNX1 contribute to its ability to deform the membrane. Moreover, we have compared the SNX1 structure with a previously elucidated structure of an endosomal coat complex formed by retromer coupled to a SNX, which reveals how the molecular organization of the SNX in this coat complex is affected by retromer. The comparison also suggests insight into intermediary stages of assembly that results in the formation of the retromer-SNX coat complex on the membrane. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30592.map.gz emd_30592.map.gz | 54.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30592-v30.xml emd-30592-v30.xml emd-30592.xml emd-30592.xml | 14.4 KB 14.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30592_fsc.xml emd_30592_fsc.xml | 13.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30592.png emd_30592.png | 123.7 KB | ||
| Filedesc metadata |  emd-30592.cif.gz emd-30592.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30592 http://ftp.pdbj.org/pub/emdb/structures/EMD-30592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30592 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30592_validation.pdf.gz emd_30592_validation.pdf.gz | 470.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30592_full_validation.pdf.gz emd_30592_full_validation.pdf.gz | 470.3 KB | 表示 | |
| XML形式データ |  emd_30592_validation.xml.gz emd_30592_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_30592_validation.cif.gz emd_30592_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30592 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30592 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30592.map.gz / 形式: CCP4 / 大きさ: 59.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30592.map.gz / 形式: CCP4 / 大きさ: 59.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.42 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : sorting nexin 1
| 全体 | 名称: sorting nexin 1 |
|---|---|
| 要素 |
|
-超分子 #1: sorting nexin 1
| 超分子 | 名称: sorting nexin 1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: sorting nexin 1 in membrane-bound state |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Sorting nexin-1
| 分子 | 名称: Sorting nexin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.740887 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPLGSPEFMA SGGGGCSASE RLPPPFPGMD PESEGAAGGS EPEAGDSDTE GEDIFTGAAA ATKPQSPKKT TSLFPIKNGS KENGIHEDQ DQEPQDLFAD ATVELSLDST QNNQKTMPGK TLTSHPPQEA TNSPKPQPSY EELEEEQEDQ FDLTVGITDP E KIGDGMNA ...文字列: GPLGSPEFMA SGGGGCSASE RLPPPFPGMD PESEGAAGGS EPEAGDSDTE GEDIFTGAAA ATKPQSPKKT TSLFPIKNGS KENGIHEDQ DQEPQDLFAD ATVELSLDST QNNQKTMPGK TLTSHPPQEA TNSPKPQPSY EELEEEQEDQ FDLTVGITDP E KIGDGMNA YVAYKVTTQT SLPMFRSRQF AVKRRFSDFL GLYEKLSEKH SQNGFIVPPP PEKSLIGMTK VKVGKEDSSS AE FLEKRRA ALERYLQRIV NHPTMLQDPD VREFLEKEEL PRAVGTQALS GAGLLKMFNK ATDAVSKMTI KMNESDIWFE EKL QEVECE EQRLRKLHAV VETLVNHRKE LALNTALFAK SLAMLGSSED NTALSRALSQ LAEVEEKIEQ LHQEQANNDF FLLA ELLSD YIRLLAIVRA AFDQRMKTWQ RWQDAQATLQ KKRESEARLL WANKPDKLQQ AKDEITEWES RVTQYERDFE RISTV VRKE VTRFEKEKSK DFKNHVMKYL ETLLHSQQQL AKYWEAFLPE AKAIS UniProtKB: Sorting nexin-1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50 mM HEPES, pH7.4, 100 mM NaCl |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 詳細: blot for 3.5 seconds with a blot force 2 before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 2-28 / 撮影したグリッド数: 2 / 実像数: 501 / 平均露光時間: 2.0 sec. / 平均電子線量: 25.0 e/Å2 詳細: Images were collected in movie mode at 16 frames per second |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 59000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | The temperature was kept at 300K, time step was 1 fs, and secondary structure restraints was also included. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-7d6d: |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)