+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM map of a de novo designed octahedral nanocage with programmable volume; design cage_O4_34 | |||||||||
 マップデータ マップデータ | DeepEMhancer sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | octahedral nanocage / octahedra / nanocage / nanomaterial / computational design / de novo / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
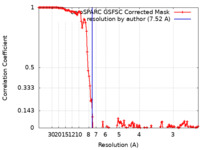
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.52 Å | |||||||||
 データ登録者 データ登録者 | Kibler RD / Borst AJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Blueprinting expandable nanomaterials with standardized protein building blocks. 要旨: A wooden house frame consists of many different lumber pieces, but because of the regularity of these building blocks, the structure can be designed using straightforward geometrical principles. The ...A wooden house frame consists of many different lumber pieces, but because of the regularity of these building blocks, the structure can be designed using straightforward geometrical principles. The design of multicomponent protein assemblies in comparison has been much more complex, largely due to the irregular shapes of protein structures . Here we describe extendable linear, curved, and angled protein building blocks, as well as inter-block interactions that conform to specified geometric standards; assemblies designed using these blocks inherit their extendability and regular interaction surfaces, enabling them to be expanded or contracted by varying the number of modules, and reinforced with secondary struts. Using X-ray crystallography and electron microscopy, we validate nanomaterial designs ranging from simple polygonal and circular oligomers that can be concentrically nested, up to large polyhedral nanocages and unbounded straight "train track" assemblies with reconfigurable sizes and geometries that can be readily blueprinted. Because of the complexity of protein structures and sequence-structure relationships, it has not been previously possible to build up large protein assemblies by deliberate placement of protein backbones onto a blank 3D canvas; the simplicity and geometric regularity of our design platform now enables construction of protein nanomaterials according to "back of an envelope" architectural blueprints. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29915.map.gz emd_29915.map.gz | 196.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29915-v30.xml emd-29915-v30.xml emd-29915.xml emd-29915.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29915_fsc.xml emd_29915_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29915.png emd_29915.png | 114.1 KB | ||
| その他 |  emd_29915_additional_1.map.gz emd_29915_additional_1.map.gz emd_29915_half_map_1.map.gz emd_29915_half_map_1.map.gz emd_29915_half_map_2.map.gz emd_29915_half_map_2.map.gz | 98.2 MB 192.4 MB 192.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29915 http://ftp.pdbj.org/pub/emdb/structures/EMD-29915 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29915 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29915 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29915_validation.pdf.gz emd_29915_validation.pdf.gz | 682.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29915_full_validation.pdf.gz emd_29915_full_validation.pdf.gz | 681.7 KB | 表示 | |
| XML形式データ |  emd_29915_validation.xml.gz emd_29915_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  emd_29915_validation.cif.gz emd_29915_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29915 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29915 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29915 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29915 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29915.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29915.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.248 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_29915_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_29915_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_29915_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : cage_O4_34
| 全体 | 名称: cage_O4_34 |
|---|---|
| 要素 |
|
-超分子 #1: cage_O4_34
| 超分子 | 名称: cage_O4_34 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The smallest size of a de novo designed octahedral nanocage with programmable volume |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: cage_O4_34
| 分子 | 名称: cage_O4_34 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEEKREIAEF AREMMEEISE RVERYVEDPV LATAVRGRAI VESAAVFAKT IGGDIEKLDE AREEVERAAE EALRRREEGA DVSELVAELI RETSRQIAEI AEATIKATDD PEVLEEISEF AEERSRRLSE YAERHVTNPI LAATVVALAE VLSAVVRARS YGAPEEVGEK ...文字列: MEEKREIAEF AREMMEEISE RVERYVEDPV LATAVRGRAI VESAAVFAKT IGGDIEKLDE AREEVERAAE EALRRREEGA DVSELVAELI RETSRQIAEI AEATIKATDD PEVLEEISEF AEERSRRLSE YAERHVTNPI LAATVVALAE VLSAVVRARS YGAPEEVGEK AVKEVREASE EALERYKEGA DESELIAEIM EATAEAVGKI AEAAIEATDD PEKRRRIARW AREQMRRLSR RAEELVEDPV LAMTVFARAQ ILAAAVFGKA VGMPEIVSEH ARRMVRLAGE AALALKKEGA DVSRLVAMVG RVTSIAVGMI AKATVSDSPV EITKELIRAF KEITEELIKA GVDGKHLNEA VRLGSEAVNE LVEEAVKEGT PVEEVTESAI KGLEAVTEMA IEAMKAGGDP LEMARIVAEL AEKAIDVVAK MGAPGTYMLA MIMAASVAHR RIVEAAIKAG APGEVLEEAV RLAGRVQVRG VEKAAELGTP KTLVEAAGEL GLATVRRMAE LAIEAGGDRE RMEEIIREVE EAMERVIERI EGGSGGSWGH HHHHHG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 25mM Tris, 300mM NaCl |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TUNDRA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / 平均電子線量: 42.6 e/Å2 |
| 電子線 | 加速電圧: 100 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 17.0 µm / 最小 デフォーカス(公称値): 8.0 µm |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)