+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Human Mitochondrial DNA Polymerase Gamma | |||||||||
 マップデータ マップデータ | Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Human Mitochondrial DNA Polymerase Gamma | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mitochondrial DNA Polymerase / DNA Proofreading / PolG / REPLICATION / REPLICATION-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報gamma DNA polymerase complex / mitochondrial chromosome / Strand-asynchronous mitochondrial DNA replication / mitochondrial DNA replication / positive regulation of DNA-directed DNA polymerase activity / DNA replication proofreading / single-stranded DNA 3'-5' DNA exonuclease activity / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ / DNA metabolic process / DNA polymerase processivity factor activity ...gamma DNA polymerase complex / mitochondrial chromosome / Strand-asynchronous mitochondrial DNA replication / mitochondrial DNA replication / positive regulation of DNA-directed DNA polymerase activity / DNA replication proofreading / single-stranded DNA 3'-5' DNA exonuclease activity / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ / DNA metabolic process / DNA polymerase processivity factor activity / 付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; その他の炭素-酸素リアーゼ / mitochondrial nucleoid / 5'-deoxyribose-5-phosphate lyase activity / base-excision repair, gap-filling / DNA polymerase binding / 3'-5' exonuclease activity / Transcriptional activation of mitochondrial biogenesis / base-excision repair / DNA-templated DNA replication / protease binding / double-stranded DNA binding / in utero embryonic development / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / mitochondrial matrix / intracellular membrane-bounded organelle / chromatin binding / protein-containing complex / mitochondrion / DNA binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
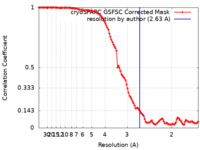
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.63 Å | |||||||||
 データ登録者 データ登録者 | Nayak AR / Buchel G / Herbine KH / Sarfallah A / Sokolova VO / Zamudio-Ochoa A / Temiakov D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural basis for DNA proofreading. 著者: Gina Buchel / Ashok R Nayak / Karl Herbine / Azadeh Sarfallah / Viktoriia O Sokolova / Angelica Zamudio-Ochoa / Dmitry Temiakov /  要旨: DNA polymerase (DNAP) can correct errors in DNA during replication by proofreading, a process critical for cell viability. However, the mechanism by which an erroneously incorporated base ...DNA polymerase (DNAP) can correct errors in DNA during replication by proofreading, a process critical for cell viability. However, the mechanism by which an erroneously incorporated base translocates from the polymerase to the exonuclease site and the corrected DNA terminus returns has remained elusive. Here, we present an ensemble of nine high-resolution structures representing human mitochondrial DNA polymerase Gamma, Polγ, captured during consecutive proofreading steps. The structures reveal key events, including mismatched base recognition, its dissociation from the polymerase site, forward translocation of DNAP, alterations in DNA trajectory, repositioning and refolding of elements for primer separation, DNAP backtracking, and displacement of the mismatched base into the exonuclease site. Altogether, our findings suggest a conserved 'bolt-action' mechanism of proofreading based on iterative cycles of DNAP translocation without dissociation from the DNA, facilitating primer transfer between catalytic sites. Functional assays and mutagenesis corroborate this mechanism, connecting pathogenic mutations to crucial structural elements in proofreading steps. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29746.map.gz emd_29746.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29746-v30.xml emd-29746-v30.xml emd-29746.xml emd-29746.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29746_fsc.xml emd_29746_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29746.png emd_29746.png | 157.8 KB | ||
| Filedesc metadata |  emd-29746.cif.gz emd-29746.cif.gz | 7.7 KB | ||
| その他 |  emd_29746_half_map_1.map.gz emd_29746_half_map_1.map.gz emd_29746_half_map_2.map.gz emd_29746_half_map_2.map.gz | 10.3 MB 10.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29746 http://ftp.pdbj.org/pub/emdb/structures/EMD-29746 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29746 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29746 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29746_validation.pdf.gz emd_29746_validation.pdf.gz | 637.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29746_full_validation.pdf.gz emd_29746_full_validation.pdf.gz | 636.8 KB | 表示 | |
| XML形式データ |  emd_29746_validation.xml.gz emd_29746_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_29746_validation.cif.gz emd_29746_validation.cif.gz | 24.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29746 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29746 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29746 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29746 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8g5jMC  8g5iC  8g5kC  8g5lC  8g5mC  8g5nC  8g5oC  8g5pC  8t7eC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29746.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29746.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Human Mitochondrial DNA Polymerase Gamma | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.826 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_29746_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_29746_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Huma...
| 全体 | 名称: Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Human Mitochondrial DNA Polymerase Gamma |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Huma...
| 超分子 | 名称: Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Human Mitochondrial DNA Polymerase Gamma タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Human mitochondrial DNA polymerase PolG (wild type) assembled on a DNA-DNA scaffold |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 362 KDa |
-分子 #1: DNA polymerase subunit gamma-1
| 分子 | 名称: DNA polymerase subunit gamma-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 139.730703 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSRLLWRKVA GATVGPGPVP APGRWVSSSV PASDPSDGQR RRQQQQQQQQ QQQQQPQQPQ VLSSEGGQLR HNPLDIQMLS RGLHEQIFG QGGEMPGEAA VRRSVEHLQK HGLWGQPAVP LPDVELRLPP LYGDNLDQHF RLLAQKQSLP YLEAANLLLQ A QLPPKPPA ...文字列: MSRLLWRKVA GATVGPGPVP APGRWVSSSV PASDPSDGQR RRQQQQQQQQ QQQQQPQQPQ VLSSEGGQLR HNPLDIQMLS RGLHEQIFG QGGEMPGEAA VRRSVEHLQK HGLWGQPAVP LPDVELRLPP LYGDNLDQHF RLLAQKQSLP YLEAANLLLQ A QLPPKPPA WAWAEGWTRY GPEGEAVPVA IPEERALVFD VEVCLAEGTC PTLAVAISPS AWYSWCSQRL VEERYSWTSQ LS PADLIPL EVPTGASSPT QRDWQEQLVV GHNVSFDRAH IREQYLIQGS RMRFLDTMSM HMAISGLSSF QRSLWIAAKQ GKH KVQPPT KQGQKSQRKA RRGPAISSWD WLDISSVNSL AEVHRLYVGG PPLEKEPREL FVKGTMKDIR ENFQDLMQYC AQDV WATHE VFQQQLPLFL ERCPHPVTLA GMLEMGVSYL PVNQNWERYL AEAQGTYEEL QREMKKSLMD LANDACQLLS GERYK EDPW LWDLEWDLQE FKQKKAKKVK KEPATASKLP IEGAGAPGDP MDQEDLGPCS EEEEFQQDVM ARACLQKLKG TTELLP KRP QHLPGHPGWY RKLCPRLDDP AWTPGPSLLS LQMRVTPKLM ALTWDGFPLH YSERHGWGYL VPGRRDNLAK LPTGTTL ES AGVVCPYRAI ESLYRKHCLE QGKQQLMPQE AGLAEEFLLT DNSAIWQTVE ELDYLEVEAE AKMENLRAAV PGQPLALT A RGGPKDTQPS YHHGNGPYND VDIPGCWFFK LPHKDGNSCN VGSPFAKDFL PKMEDGTLQA GPGGASGPRA LEINKMISF WRNAHKRISS QMVVWLPRSA LPRAVIRHPD YDEEGLYGAI LPQVVTAGTI TRRAVEPTWL TASNARPDRV GSELKAMVQA PPGYTLVGA DVDSQELWIA AVLGDAHFAG MHGCTAFGWM TLQGRKSRGT DLHSKTATTV GISREHAKIF NYGRIYGAGQ P FAERLLMQ FNHRLTQQEA AEKAQQMYAA TKGLRWYRLS DEGEWLVREL NLPVDRTEGG WISLQDLRKV QRETARKSQW KK WEVVAER AWKGGTESEM FNKLESIATS DIPRTPVLGC CISRALEPSA VQEEFMTSRV NWVVQSSAVD YLHLMLVAMK WLF EEFAID GRFCISIHDE VRYLVREEDR YRAALALQIT NLLTRCMFAY KLGLNDLPQS VAFFSAVDID RCLRKEVTMD CKTP SNPTG MERRYGIPQG EALDIYQIIE LTKGSLEKRS QPGP UniProtKB: DNA polymerase subunit gamma-1 |
-分子 #2: DNA polymerase subunit gamma-2, mitochondrial
| 分子 | 名称: DNA polymerase subunit gamma-2, mitochondrial / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 54.991 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRSRVAVRAC HKVCRCLLSG FGGRVDAGQP ELLTERSSPK GGHVKSHAEL EGNGEHPEAP GSGEGSEALL EICQRRHFLS GSKQQLSRD SLLSGCHPGF GPLGVELRKN LAAEWWTSVV VFREQVFPVD ALHHKPGPLL PGDSAFRLVS AETLREILQD K ELSKEQLV ...文字列: MRSRVAVRAC HKVCRCLLSG FGGRVDAGQP ELLTERSSPK GGHVKSHAEL EGNGEHPEAP GSGEGSEALL EICQRRHFLS GSKQQLSRD SLLSGCHPGF GPLGVELRKN LAAEWWTSVV VFREQVFPVD ALHHKPGPLL PGDSAFRLVS AETLREILQD K ELSKEQLV AFLENVLKTS GKLRENLLHG ALEHYVNCLD LVNKRLPYGL AQIGVCFHPV FDTKQIRNGV KSIGEKTEAS LV WFTPPRT SNQWLDFWLR HRLQWWRKFA MSPSNFSSSD CQDEEGRKGN KLYYNFPWGK ELIETLWNLG DHELLHMYPG NVS KLHGRD GRKNVVPCVL SVNGDLDRGM LAYLYDSFQL TENSFTRKKN LHRKVLKLHP CLAPIKVALD VGRGPTLELR QVCQ GLFNE LLENGISVWP GYLETMQSSL EQLYSKYDEM SILFTVLVTE TTLENGLIHL RSRDTTMKEM MHISKLKDFL IKYIS SAKN V UniProtKB: DNA polymerase subunit gamma-2 |
-分子 #3: Mismatched Primer DNA
| 分子 | 名称: Mismatched Primer DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 6.209018 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DG)(DA)(DC)(DA)(DG)(DT)(DC) (DT)(DG)(DC)(DG)(DG)(DC)(DG)(DC)(DG)(DA) |
-分子 #4: Template DNA
| 分子 | 名称: Template DNA / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.894083 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DC)(DA)(DC)(DG)(DC)(DG)(DC) (DG)(DC)(DC)(DG)(DC)(DA)(DG)(DA)(DC)(DT) (DG)(DT)(DC)(DT)(DT)(DC) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.52 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 詳細: 10 mM Tris-Hcl pH 7.9, 100 mM Nacl, 10 mM DTT, and 2 mM MgCl2 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | 2 uM complex of the wild type PolG and DNA-DNA scaffold containing a mismatched base |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 10572 / 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 18.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8g5j: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)