+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Hybrid aspen cellulose synthase-8 bound to UDP | |||||||||
 マップデータ マップデータ | autosharpened map local refinement in cryosparc | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cellulose / cell wall / UDP / PLANT PROTEIN / Transferase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellulose synthase (UDP-forming) / cellulose synthase (UDP-forming) activity / cellulose biosynthetic process / cell wall organization / zinc ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Verma P / Zimmer J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Insights into substrate coordination and glycosyl transfer of poplar cellulose synthase-8. 著者: Preeti Verma / Albert L Kwansa / Ruoya Ho / Yaroslava G Yingling / Jochen Zimmer /  要旨: Cellulose is an abundant cell wall component of land plants. It is synthesized from UDP-activated glucose molecules by cellulose synthase, a membrane-integrated processive glycosyltransferase. ...Cellulose is an abundant cell wall component of land plants. It is synthesized from UDP-activated glucose molecules by cellulose synthase, a membrane-integrated processive glycosyltransferase. Cellulose synthase couples the elongation of the cellulose polymer with its translocation across the plasma membrane. Here, we present substrate and product-bound cryogenic electron microscopy structures of the homotrimeric cellulose synthase isoform-8 (CesA8) from hybrid aspen (poplar). UDP-glucose binds to a conserved catalytic pocket adjacent to the entrance to a transmembrane channel. The substrate's glucosyl unit is coordinated by conserved residues of the glycosyltransferase domain and amphipathic interface helices. Site-directed mutagenesis of a conserved gating loop capping the active site reveals its critical function for catalytic activity. Molecular dynamics simulations reveal prolonged interactions of the gating loop with the substrate molecule, particularly across its central conserved region. These transient interactions likely facilitate the proper positioning of the substrate molecule for glycosyl transfer and cellulose translocation. HIGHLIGHTS: Cryo-EM structures of substrate and product bound poplar cellulose synthase provide insights into substrate selectivitySite directed mutagenesis signifies a critical function of the ...HIGHLIGHTS: Cryo-EM structures of substrate and product bound poplar cellulose synthase provide insights into substrate selectivitySite directed mutagenesis signifies a critical function of the gating loop for catalysisMolecular dynamics simulations support persistent gating loop - substrate interactionsGating loop helps in positioning the substrate molecule to facilitate cellulose elongationConserved cellulose synthesis substrate binding mechanism across the kingdoms. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29678.map.gz emd_29678.map.gz | 223.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29678-v30.xml emd-29678-v30.xml emd-29678.xml emd-29678.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_29678.png emd_29678.png | 68.2 KB | ||
| Filedesc metadata |  emd-29678.cif.gz emd-29678.cif.gz | 7.1 KB | ||
| その他 |  emd_29678_half_map_1.map.gz emd_29678_half_map_1.map.gz emd_29678_half_map_2.map.gz emd_29678_half_map_2.map.gz | 226.8 MB 226.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29678 http://ftp.pdbj.org/pub/emdb/structures/EMD-29678 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29678 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29678 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29678_validation.pdf.gz emd_29678_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29678_full_validation.pdf.gz emd_29678_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_29678_validation.xml.gz emd_29678_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_29678_validation.cif.gz emd_29678_validation.cif.gz | 19.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29678 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29678 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29678 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29678 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8g27MC  8g2jC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29678.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29678.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | autosharpened map local refinement in cryosparc | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map A
| ファイル | emd_29678_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
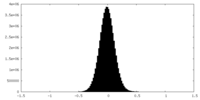
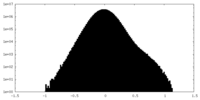
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_29678_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
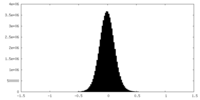
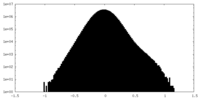
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homotrimeric cellulose synthase complex
| 全体 | 名称: Homotrimeric cellulose synthase complex |
|---|---|
| 要素 |
|
-超分子 #1: Homotrimeric cellulose synthase complex
| 超分子 | 名称: Homotrimeric cellulose synthase complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Cellulose synthase
| 分子 | 名称: Cellulose synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号: cellulose synthase (UDP-forming) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 112.483023 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHHHH HHHMMESGAP ICHTCGEQVG HDANGDLFVA CHECNYHICK SCFEYEIKEG RKVCLRCGSP YDENLLDDVE KKGSGNQST MASHLNNSQD VGIHARHISS VSTVDSEMND EYGNPIWKNR VESWKDKRNK KKKSNTKPET EPAQVPPEQQ M ENKPSAEA ...文字列: MHHHHHHHHH HHHMMESGAP ICHTCGEQVG HDANGDLFVA CHECNYHICK SCFEYEIKEG RKVCLRCGSP YDENLLDDVE KKGSGNQST MASHLNNSQD VGIHARHISS VSTVDSEMND EYGNPIWKNR VESWKDKRNK KKKSNTKPET EPAQVPPEQQ M ENKPSAEA SEPLSIVYPI PRNKLTPYRA VIIMRLIILG LFFHYRITNP VDSAFGLWLT SVICEIWFAF SWVLDQFPKW KP VNRETFI ERLSARYERE GEPSQLAAVD FFVSTVDPLK EPPLITANTV LSILAVDYPV DKVSCYVSDD GAAMLTFESL VET AEFARK WVPFCKKFSI EPRAPEFYFS QKIDYLKDKV QPSFVKERRA MKRDYEEYKV RVNALVAKAQ KTPDEGWTMQ DGTP WPGNN TRDHPGMIQV FLGNTGARDI EGNELPRLVY VSREKRPGYQ HHKKAGAENA LVRVSAVLTN APYILNLDCD HYVNN SKAV REAMCILMDP QVGRDVCYVQ FPQRFDGIDR SDRYANRNIV FFDVNMKGLD GIQGPMYVGT GCVFNRQALY GYGPPS MPR LRKGKESSSC FSCCCPTKKK PAQDPAEVYR DAKREDLNAA IFNLTEIDNY DDYERSMLIS QLSFEKTFGL SPVFIES TL MENGGVPESA NSSTLIKEAI HVIGCGFEEK TEWGKEIGWI YGSVTEDILS GFKMHCRGWR SIYCMPVRPA FKGSAPIN L SDRLHQVLRW ALGSVEIFFS RHCPFWYGYG GGRLKWLQRL AYINTIVYPF TSLPLIAYCT IPAVCLLTGK FIIPTLSNL ASMLFLGLFI SIIVTAVLEL RWSGVSIEDL WRNEQFWVIG GVSAHLFAVF QGFLKMLAGI DTNFTVTAKA ADDTEFGELY MVKWTTLLI PPTTLLIINI VGVVAGFSDA LNKGYEAWGP LFGKVFFAFW VILHLYPFLK GLMGRQNRTP TIVVLWSVLL T SVFSLVWV KINPFVNKVD NTLAGETCIS IDC UniProtKB: Cellulose synthase |
-分子 #3: URIDINE-5'-DIPHOSPHATE
| 分子 | 名称: URIDINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 3 / 式: UDP |
|---|---|
| 分子量 | 理論値: 404.161 Da |
| Chemical component information |  ChemComp-UDP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 15.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)