登録情報 データベース : EMDB / ID : EMD-29423タイトル Structure of Escherichia coli CedA in complex with transcription initiation complex 複合体 : Escherichia coli CedA in complex with initiation complexタンパク質・ペプチド : x 6種DNA : x 2種リガンド : x 3種 / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Escherichia coli (大腸菌)手法 / / 解像度 : 2.76 Å Liu M / Vassyliev N / Nudler E 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) GM126891 Howard Hughes Medical Institute (HHMI) Other private Blavatnik Family Foundation
ジャーナル : Nat Struct Mol Biol / 年 : 2024タイトル : General transcription factor from Escherichia coli with a distinct mechanism of action.著者 : Nikita Vasilyev / Mengjie M J Liu / Vitaly Epshtein / Ilya Shamovsky / Evgeny Nudler / 要旨 : Gene expression in Escherichia coli is controlled by well-established mechanisms that activate or repress transcription. Here, we identify CedA as an unconventional transcription factor specifically ... Gene expression in Escherichia coli is controlled by well-established mechanisms that activate or repress transcription. Here, we identify CedA as an unconventional transcription factor specifically associated with the RNA polymerase (RNAP) σ holoenzyme. Structural and biochemical analysis of CedA bound to RNAP reveal that it bridges distant domains of β and σ subunits to stabilize an open-promoter complex. CedA does so without contacting DNA. We further show that cedA is strongly induced in response to amino acid starvation, oxidative stress and aminoglycosides. CedA provides a basal level of tolerance to these clinically relevant antibiotics, as well as to rifampicin and peroxide. Finally, we show that CedA modulates transcription of hundreds of bacterial genes, which explains its pleotropic effect on cell physiology and pathogenesis. 履歴 登録 2023年1月11日 - ヘッダ(付随情報) 公開 2024年1月10日 - マップ公開 2024年1月10日 - 更新 2025年6月4日 - 現状 2025年6月4日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Nat Struct Mol Biol / 年: 2024
ジャーナル: Nat Struct Mol Biol / 年: 2024
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_29423.map.gz
emd_29423.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-29423-v30.xml
emd-29423-v30.xml emd-29423.xml
emd-29423.xml EMDBヘッダ
EMDBヘッダ emd_29423.png
emd_29423.png emd-29423.cif.gz
emd-29423.cif.gz emd_29423_half_map_1.map.gz
emd_29423_half_map_1.map.gz emd_29423_half_map_2.map.gz
emd_29423_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-29423
http://ftp.pdbj.org/pub/emdb/structures/EMD-29423 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29423
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29423 emd_29423_validation.pdf.gz
emd_29423_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_29423_full_validation.pdf.gz
emd_29423_full_validation.pdf.gz emd_29423_validation.xml.gz
emd_29423_validation.xml.gz emd_29423_validation.cif.gz
emd_29423_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29423
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29423 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29423
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29423
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
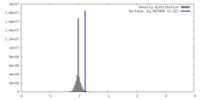
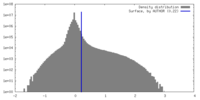
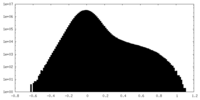
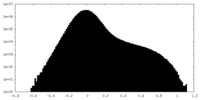
マップ ダウンロード / ファイル: emd_29423.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_29423.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)