+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
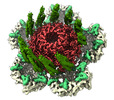
| Title | portal vertex of HK97 phage | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Prohead I / icosahedral symmetry / HK97 / phage / capsid / VIRUS | |||||||||
| Function / homology | Phage portal protein, HK97 / Bacteriophage/Gene transfer agent portal protein / Phage portal protein / symbiont genome ejection through host cell envelope, long flexible tail mechanism / viral capsid / Portal protein Function and homology information Function and homology information | |||||||||
| Biological species |  Escherichia phage HK97 (virus) Escherichia phage HK97 (virus) | |||||||||
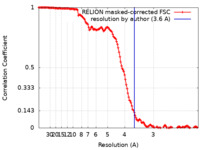
| Method | single particle reconstruction / cryo EM / Resolution: 3.6 Å | |||||||||
 Authors Authors | Huet A / Oh B / Maurer J / Duda RL / Conway JF | |||||||||
| Funding support |  United States, 2 items United States, 2 items
| |||||||||
 Citation Citation |  Journal: Sci Adv / Year: 2023 Journal: Sci Adv / Year: 2023Title: A symmetry mismatch unraveled: How phage HK97 scaffold flexibly accommodates a 12-fold pore at a 5-fold viral capsid vertex. Authors: Alexis Huet / Bonnie Oh / Josh Maurer / Robert L Duda / James F Conway /  Abstract: Tailed bacteriophages and herpesviruses use a transient scaffold to assemble icosahedral capsids with hexameric capsomers on the faces and pentameric capsomers at all but one vertex where a 12-fold ...Tailed bacteriophages and herpesviruses use a transient scaffold to assemble icosahedral capsids with hexameric capsomers on the faces and pentameric capsomers at all but one vertex where a 12-fold portal is thought to nucleate the assembly. How does the scaffold orchestrate this step? We have determined the portal vertex structure of the bacteriophage HK97 procapsid, where the scaffold is a domain of the major capsid protein. The scaffold forms rigid helix-turn-strand structures on the interior surfaces of all capsomers and is further stabilized around the portal, forming trimeric coiled-coil towers, two per surrounding capsomer. These 10 towers bind identically to 10 of 12 portal subunits, adopting a pseudo-12-fold organization that explains how the symmetry mismatch is managed at this early step. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_29392.map.gz emd_29392.map.gz | 228.3 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-29392-v30.xml emd-29392-v30.xml emd-29392.xml emd-29392.xml | 17 KB 17 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_29392_fsc.xml emd_29392_fsc.xml | 14.2 KB | Display |  FSC data file FSC data file |
| Images |  emd_29392.png emd_29392.png | 187.6 KB | ||
| Filedesc metadata |  emd-29392.cif.gz emd-29392.cif.gz | 5.8 KB | ||
| Others |  emd_29392_half_map_1.map.gz emd_29392_half_map_1.map.gz emd_29392_half_map_2.map.gz emd_29392_half_map_2.map.gz | 193.9 MB 193.9 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29392 http://ftp.pdbj.org/pub/emdb/structures/EMD-29392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29392 | HTTPS FTP |
-Related structure data
| Related structure data |  8fqlMC  8fqkC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_29392.map.gz / Format: CCP4 / Size: 244.1 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_29392.map.gz / Format: CCP4 / Size: 244.1 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #1
| File | emd_29392_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_29392_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Escherichia phage HK97
| Entire | Name:  Escherichia phage HK97 (virus) Escherichia phage HK97 (virus) |
|---|---|
| Components |
|
-Supramolecule #1: Escherichia phage HK97
| Supramolecule | Name: Escherichia phage HK97 / type: virus / ID: 1 / Parent: 0 / Macromolecule list: all / Details: Prohead I expressed from plasmid in E.Coli / NCBI-ID: 2681617 / Sci species name: Escherichia phage HK97 / Virus type: VIRUS-LIKE PARTICLE / Virus isolate: OTHER / Virus enveloped: No / Virus empty: Yes |
|---|---|
| Host (natural) | Organism:  |
| Molecular weight | Theoretical: 17.7 MDa |
| Virus shell | Shell ID: 1 / Name: Prohead I / Diameter: 500.0 Å / T number (triangulation number): 7 |
-Macromolecule #1: Portal protein
| Macromolecule | Name: Portal protein / type: protein_or_peptide / ID: 1 / Number of copies: 12 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Escherichia phage HK97 (virus) Escherichia phage HK97 (virus) |
| Molecular weight | Theoretical: 47.318488 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MEEPKYTIDL RTNNGWWARL KSWFVGGRLV TPNQGSQTGP VSAHGYLGDS SINDERILQI STVWRCVSLI STLTACLPLD VFETDQNDN RKKVDLSNPL ARLLRYSPNQ YMTAQEFREA MTMQLCFYGN AYALVDRNSA GDVISLLPLQ SANMDVKLVG K KVVYRYQR ...String: MEEPKYTIDL RTNNGWWARL KSWFVGGRLV TPNQGSQTGP VSAHGYLGDS SINDERILQI STVWRCVSLI STLTACLPLD VFETDQNDN RKKVDLSNPL ARLLRYSPNQ YMTAQEFREA MTMQLCFYGN AYALVDRNSA GDVISLLPLQ SANMDVKLVG K KVVYRYQR DSEYADFSQK EIFHLKGFGF TGLVGLSPIA FACKSAGVAV AMEDQQRDFF ANGAKSPQIL STGEKVLTEQ QR SQVEENF KEIAGGPVKK RLWILEAGFS TSAIGVTPQD AEMMASRKFQ VSELARFFGV PPHLVGDVEK STSWGSGIEQ QNL GFLQYT LQPYISRWEN SIQRWLIPSK DVGRLHAEHN LDGLLRGDSA SRAAFMKAMG ESGLRTINEM RRTDNMPPLP GGDV AMRQA QYVPITDLGT NKEPRNNGA UniProtKB: Portal protein |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 10 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.5 Component:
| |||||||||
| Grid | Model: Quantifoil R2/1 / Material: COPPER / Mesh: 400 / Support film - Material: CARBON / Support film - topology: HOLEY | |||||||||
| Vitrification | Cryogen name: ETHANE-PROPANE / Chamber humidity: 100 % / Chamber temperature: 293 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON III (4k x 4k) / Detector mode: COUNTING / Number grids imaged: 2 / Number real images: 2909 / Average electron dose: 60.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Calibrated defocus max: 4.0 µm / Calibrated defocus min: 1.0 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 4.0 µm / Nominal defocus min: 1.0 µm / Nominal magnification: 75000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Refinement | Space: REAL / Protocol: FLEXIBLE FIT |
|---|---|
| Output model |  PDB-8fql: |
 Movie
Movie Controller
Controller






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)