+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DNA replication fork binding triggers structural changes in the PriA DNA helicase that regulate the PriA-PriB replication restart pathway in E. coli | |||||||||
 マップデータ マップデータ | final sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PriA / PriB / replication restart / E. coli / HELICASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pre-primosome complex / DnaB-DnaC-DnaT-PriA-PriC complex / DnaB-DnaC-DnaT-PriA-PriB complex / plasmid maintenance / primosome complex / DNA replication, synthesis of primer / DNA 3'-5' helicase / 3'-5' DNA helicase activity / replication fork processing / DNA replication initiation ...pre-primosome complex / DnaB-DnaC-DnaT-PriA-PriC complex / DnaB-DnaC-DnaT-PriA-PriB complex / plasmid maintenance / primosome complex / DNA replication, synthesis of primer / DNA 3'-5' helicase / 3'-5' DNA helicase activity / replication fork processing / DNA replication initiation / response to gamma radiation / helicase activity / response to radiation / DNA-templated DNA replication / double-strand break repair / single-stranded DNA binding / DNA recombination / DNA replication / response to antibiotic / ATP hydrolysis activity / DNA binding / zinc ion binding / ATP binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
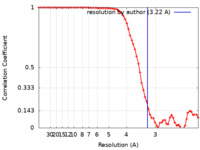
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.22 Å | |||||||||
 データ登録者 データ登録者 | Duckworth AT / Ducos PL / McMillan SD / Satyshur KA / Blumenthal KH / Deorio HR / Larson JA / Sandler SJ / Grant T / Keck JL | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Replication fork binding triggers structural changes in the PriA helicase that govern DNA replication restart in E. coli. 著者: Alexander T Duckworth / Peter L Ducos / Sarah D McMillan / Kenneth A Satyshur / Katelien H Blumenthal / Haley R Deorio / Joseph A Larson / Steven J Sandler / Timothy Grant / James L Keck /  要旨: Bacterial replisomes often dissociate from replication forks before chromosomal replication is complete. To avoid the lethal consequences of such situations, bacteria have evolved replication restart ...Bacterial replisomes often dissociate from replication forks before chromosomal replication is complete. To avoid the lethal consequences of such situations, bacteria have evolved replication restart pathways that reload replisomes onto prematurely terminated replication forks. To understand how the primary replication restart pathway in E. coli (PriA-PriB) selectively acts on replication forks, we determined the cryogenic-electron microscopy structure of a PriA/PriB/replication fork complex. Replication fork specificity arises from extensive PriA interactions with each arm of the branched DNA. These interactions reshape the PriA protein to create a pore encircling single-stranded lagging-strand DNA while also exposing a surface of PriA onto which PriB docks. Together with supporting biochemical and genetic studies, the structure reveals a switch-like mechanism for replication restart initiation in which restructuring of PriA directly couples replication fork recognition to PriA/PriB complex formation to ensure robust and high-fidelity replication re-initiation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28959.map.gz emd_28959.map.gz | 25.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28959-v30.xml emd-28959-v30.xml emd-28959.xml emd-28959.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28959_fsc.xml emd_28959_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28959.png emd_28959.png | 122.1 KB | ||
| Filedesc metadata |  emd-28959.cif.gz emd-28959.cif.gz | 6.9 KB | ||
| その他 |  emd_28959_half_map_1.map.gz emd_28959_half_map_1.map.gz emd_28959_half_map_2.map.gz emd_28959_half_map_2.map.gz | 25 MB 25 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28959 http://ftp.pdbj.org/pub/emdb/structures/EMD-28959 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28959 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28959 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28959_validation.pdf.gz emd_28959_validation.pdf.gz | 1004.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28959_full_validation.pdf.gz emd_28959_full_validation.pdf.gz | 1003.9 KB | 表示 | |
| XML形式データ |  emd_28959_validation.xml.gz emd_28959_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_28959_validation.cif.gz emd_28959_validation.cif.gz | 17.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28959 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28959 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28959 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28959 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fakMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28959.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28959.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.079 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: unfiltered / unsharpened half map 1
| ファイル | emd_28959_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered / unsharpened half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: unfiltered / unsharpened half map 2
| ファイル | emd_28959_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered / unsharpened half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of PriA, PriB, and replication fork
| 全体 | 名称: Complex of PriA, PriB, and replication fork |
|---|---|
| 要素 |
|
-超分子 #1: Complex of PriA, PriB, and replication fork
| 超分子 | 名称: Complex of PriA, PriB, and replication fork / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 134 KDa |
-分子 #1: Primosomal replication protein N
| 分子 | 名称: Primosomal replication protein N / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.459194 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTNRLVLSGT VCRAPLRKVS PSGIPHCQFV LEHRSVQEEA GFHRQAWCQM PVIVSGHENQ AITHSITVGS RITVQGFISC HKAKNGLSK MVLHAEQIEL IDSGD UniProtKB: Replication restart protein PriB |
-分子 #3: Primosomal protein N'
| 分子 | 名称: Primosomal protein N' / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 81.76582 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPVAHVALPV PLPRTFDYLL PEGMTVKAGC RVRVPFGKQQ ERIGIVVSVS DASELPLNEL KAVVEVLDSE PVFTHSVWRL LLWAADYYH HPIGDVLFHA LPILLRQGRP AANAPMWYWF ATEQGQAVDL NSLKRSPKQQ QALAALRQGK IWRDQVATLE F NDAALQAL ...文字列: MPVAHVALPV PLPRTFDYLL PEGMTVKAGC RVRVPFGKQQ ERIGIVVSVS DASELPLNEL KAVVEVLDSE PVFTHSVWRL LLWAADYYH HPIGDVLFHA LPILLRQGRP AANAPMWYWF ATEQGQAVDL NSLKRSPKQQ QALAALRQGK IWRDQVATLE F NDAALQAL RKKGLCDLAS ETPEFSDWRT NYAVSGERLR LNTEQATAVG AIHSAADTFS AWLLAGVTGS GKTEVYLSVL EN VLAQGKQ ALVMVPEIGL TPQTIARFRE RFNAPVEVLH SGLNDSERLS AWLKAKNGEA AIVIGTRSAL FTPFKNLGVI VID EEHDSS YKQQEGWRYH ARDLAVYRAH SEQIPIILGS ATPALETLCN VQQKKYRLLR LTRRAGNARP AIQHVLDLKG QKVQ AGLAP ALITRMRQHL QADNQVILFL NRRGFAPALL CHDCGWIAEC PRCDHYYTLH QAQHHLRCHH CDSQRPVPRQ CPSCG STHL VPVGLGTEQL EQTLAPLFPG VPISRIDRDT TSRKGALEQQ LAEVHRGGAR ILIGTQMLAK GHHFPDVTLV ALLDVD GAL FSADFRSAER FAQLYTQVAG RAGRAGKQGE VVLQTHHPEH PLLQTLLYKG YDAFAEQALA ERRMMQLPPW TSHVIVR AE DHNNQHAPLF LQQLRNLILS SPLADEKLWV LGPVPALAPK RGGRWRWQIL LQHPSRVRLQ HIINGTLALI NTIPDSRK V KWVLDVDPIE G UniProtKB: Replication restart protein PriA |
-分子 #2: DNA (5'-D(P*CP*AP*GP*AP*CP*TP*CP*AP*TP*TP*TP*AP*GP*CP*CP*CP*TP*TP...
| 分子 | 名称: DNA (5'-D(P*CP*AP*GP*AP*CP*TP*CP*AP*TP*TP*TP*AP*GP*CP*CP*CP*TP*TP*AP*TP*CP*CP*G)-3') タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.206812 KDa |
| 配列 | 文字列: (DG)(DC)(DC)(DG)(DC)(DA)(DG)(DA)(DC)(DT) (DC)(DA)(DT)(DT)(DT)(DA)(DG)(DC)(DC)(DC) (DT)(DT)(DA)(DT)(DC)(DC)(DG)(DT)(DA) (DT)(DT)(DG)(DC)(DG)(DG)(DT)(DC)(DT)(DC) (DG) |
-分子 #4: DNA (5'-D(P*CP*GP*GP*AP*TP*AP*AP*GP*GP*GP*CP*TP*GP*AP*GP*CP*AP*CP...
| 分子 | 名称: DNA (5'-D(P*CP*GP*GP*AP*TP*AP*AP*GP*GP*GP*CP*TP*GP*AP*GP*CP*AP*CP*GP*CP*CP*GP*A)-3') タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.399983 KDa |
| 配列 | 文字列: (DC)(DG)(DA)(DG)(DA)(DC)(DC)(DG)(DC)(DA) (DA)(DT)(DA)(DC)(DG)(DG)(DA)(DT)(DA)(DA) (DG)(DG)(DG)(DC)(DT)(DG)(DA)(DG)(DC) (DA)(DC)(DG)(DC)(DC)(DG)(DA)(DC)(DG)(DA) (DA) |
-分子 #5: DNA (5'-D(P*TP*CP*GP*GP*CP*GP*TP*GP*CP*TP*C)-3')
| 分子 | 名称: DNA (5'-D(P*TP*CP*GP*GP*CP*GP*TP*GP*CP*TP*C)-3') / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.567944 KDa |
| 配列 | 文字列: (DT)(DT)(DC)(DG)(DT)(DC)(DG)(DG)(DC)(DG) (DT)(DG)(DC)(DT)(DC) |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil / 材質: GOLD / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 撮影したグリッド数: 1 / #0 - 実像数: 1600 / #0 - 平均電子線量: 100.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: FEI FALCON III (4k x 4k) #1 - 検出モード: INTEGRATING / #1 - 撮影したグリッド数: 1 / #1 - 実像数: 1200 / #1 - 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8fak: |
 ムービー
ムービー コントローラー
コントローラー




 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)