+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of alkane 1-monooxygenase AlkB-AlkG complex from Fontimonas thermophila | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Electron transfer complex / iron-sulfur cluster / histidine-diiron center / hydrophobic alkane binding pocket / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Fontimonas thermophila (バクテリア) Fontimonas thermophila (バクテリア) | |||||||||
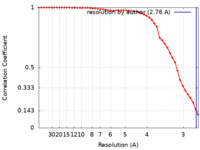
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.76 Å | |||||||||
 データ登録者 データ登録者 | Chai J / Guo G / McSweeney S / Shanklin J / Liu Q | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structural basis for enzymatic terminal C-H bond functionalization of alkanes. 著者: Jin Chai / Gongrui Guo / Sean M McSweeney / John Shanklin / Qun Liu /  要旨: Alkane monooxygenase (AlkB) is a widely occurring integral membrane metalloenzyme that catalyzes the initial step in the functionalization of recalcitrant alkanes with high terminal selectivity. AlkB ...Alkane monooxygenase (AlkB) is a widely occurring integral membrane metalloenzyme that catalyzes the initial step in the functionalization of recalcitrant alkanes with high terminal selectivity. AlkB enables diverse microorganisms to use alkanes as their sole carbon and energy source. Here we present the 48.6-kDa cryo-electron microscopy structure of a natural fusion from Fontimonas thermophila between AlkB and its electron donor AlkG at 2.76 Å resolution. The AlkB portion contains six transmembrane helices with an alkane entry tunnel within its transmembrane domain. A dodecane substrate is oriented by hydrophobic tunnel-lining residues to present a terminal C-H bond toward a diiron active site. AlkG, an [Fe-4S] rubredoxin, docks via electrostatic interactions and sequentially transfers electrons to the diiron center. The archetypal structural complex presented reveals the basis for terminal C-H selectivity and functionalization within this broadly distributed evolutionary class of enzymes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28890.map.gz emd_28890.map.gz | 4.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28890-v30.xml emd-28890-v30.xml emd-28890.xml emd-28890.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28890_fsc.xml emd_28890_fsc.xml | 4.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28890.png emd_28890.png | 122.8 KB | ||
| Filedesc metadata |  emd-28890.cif.gz emd-28890.cif.gz | 5.4 KB | ||
| その他 |  emd_28890_half_map_1.map.gz emd_28890_half_map_1.map.gz emd_28890_half_map_2.map.gz emd_28890_half_map_2.map.gz | 7.4 MB 7.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28890 http://ftp.pdbj.org/pub/emdb/structures/EMD-28890 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28890 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28890 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8f6tMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28890.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28890.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.332 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28890_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
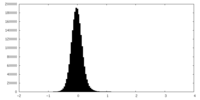
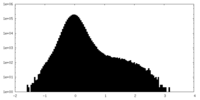
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28890_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
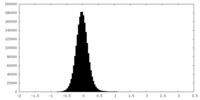
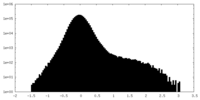
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Electron transfer complex of AlkB and AlkG
| 全体 | 名称: Electron transfer complex of AlkB and AlkG |
|---|---|
| 要素 |
|
-超分子 #1: Electron transfer complex of AlkB and AlkG
| 超分子 | 名称: Electron transfer complex of AlkB and AlkG / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Fontimonas thermophila (バクテリア) Fontimonas thermophila (バクテリア) |
-分子 #1: Alkane 1-monooxygenase
| 分子 | 名称: Alkane 1-monooxygenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: alkane 1-monooxygenase |
|---|---|
| 由来(天然) | 生物種:  Fontimonas thermophila (バクテリア) Fontimonas thermophila (バクテリア) |
| 分子量 | 理論値: 52.942164 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMSTPTLD AGTLAWNDGK RYLWLLSPFI PVLGLIGLGL FLYTDIGLFT WSGPLLIYGL IPLLDWLVGE DRNNPPEAAV AQLENDRYY RAIVYAYLPT QYAVTVLGTW VAVTADLAIW EYIGLVLSVG AVNGIGINTA HELGHKRENL DRWLAKLTLA P VAYGHFFV ...文字列: SNAMSTPTLD AGTLAWNDGK RYLWLLSPFI PVLGLIGLGL FLYTDIGLFT WSGPLLIYGL IPLLDWLVGE DRNNPPEAAV AQLENDRYY RAIVYAYLPT QYAVTVLGTW VAVTADLAIW EYIGLVLSVG AVNGIGINTA HELGHKRENL DRWLAKLTLA P VAYGHFFV EHNRGHHKNV ATPEDPASSK MGESFWAFLP RTVIGSLRSA WAIEKARLQR NKQSVWSLDN ENLQAWAMTI VL FGALTAC LGWPALLFLV LQAAYGASLL EVINYIEHYG LLRQKLPDGR YERCQPRHSW NSNHIVTNLF LYQLQRHSDH HAN PTRRFQ ALRHFDDSPQ LPSGYASMLI PAYVPWLWFR LMDPLVARHY GGDLTKANLY PPKRAALLAR WHRPRPDARR ADTQ PTDAT ATPADAAASP GGRYQCTDCG YIYDEAIGCP REGFPPGTPW SQIPDDWSCP DCAVRDKVDF RKLPAA UniProtKB: Alkane 1-monooxygenase |
-分子 #2: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 2 / コピー数: 3 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-分子 #3: DODECANE
| 分子 | 名称: DODECANE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: D12 |
|---|---|
| 分子量 | 理論値: 170.335 Da |
| Chemical component information |  ChemComp-D12: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)