+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Herpes simplex virus 1 DNA polymerase holoenzyme bound to DNA template and primer, dNTP-free (editing mode) | |||||||||
 マップデータ マップデータ | HSV-1 polymerase holoenzyme bound by template and primer DNA, dNTP-free sample (editing conformation) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | herpes simplex virus / replication / DNA polymerase / holoenzyme / editing complex / VIRAL PROTEIN-DNA complex | |||||||||
| 生物種 |   Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) / Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) /   Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) / synthetic construct (人工物) Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) / synthetic construct (人工物) | |||||||||
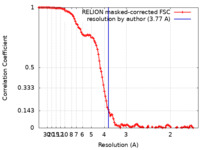
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.77 Å | |||||||||
 データ登録者 データ登録者 | Pan J / Abraham J / Coen DM / Shankar S / Yang P / Hogle J | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: Viral DNA polymerase structures reveal mechanisms of antiviral drug resistance. 著者: Sundaresh Shankar / Junhua Pan / Pan Yang / Yuemin Bian / Gábor Oroszlán / Zishuo Yu / Purba Mukherjee / David J Filman / James M Hogle / Mrinal Shekhar / Donald M Coen / Jonathan Abraham /    要旨: DNA polymerases are important drug targets, and many structural studies have captured them in distinct conformations. However, a detailed understanding of the impact of polymerase conformational ...DNA polymerases are important drug targets, and many structural studies have captured them in distinct conformations. However, a detailed understanding of the impact of polymerase conformational dynamics on drug resistance is lacking. We determined cryoelectron microscopy (cryo-EM) structures of DNA-bound herpes simplex virus polymerase holoenzyme in multiple conformations and interacting with antivirals in clinical use. These structures reveal how the catalytic subunit Pol and the processivity factor UL42 bind DNA to promote processive DNA synthesis. Unexpectedly, in the absence of an incoming nucleotide, we observed Pol in multiple conformations with the closed state sampled by the fingers domain. Drug-bound structures reveal how antivirals may selectively bind enzymes that more readily adopt the closed conformation. Molecular dynamics simulations and the cryo-EM structure of a drug-resistant mutant indicate that some resistance mutations modulate conformational dynamics rather than directly impacting drug binding, thus clarifying mechanisms that drive drug selectivity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28664.map.gz emd_28664.map.gz | 117 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28664-v30.xml emd-28664-v30.xml emd-28664.xml emd-28664.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28664_fsc.xml emd_28664_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28664.png emd_28664.png | 60.1 KB | ||
| Filedesc metadata |  emd-28664.cif.gz emd-28664.cif.gz | 7.4 KB | ||
| その他 |  emd_28664_half_map_1.map.gz emd_28664_half_map_1.map.gz emd_28664_half_map_2.map.gz emd_28664_half_map_2.map.gz | 98.2 MB 98.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28664 http://ftp.pdbj.org/pub/emdb/structures/EMD-28664 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28664 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28664 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28664_validation.pdf.gz emd_28664_validation.pdf.gz | 845.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28664_full_validation.pdf.gz emd_28664_full_validation.pdf.gz | 845.4 KB | 表示 | |
| XML形式データ |  emd_28664_validation.xml.gz emd_28664_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_28664_validation.cif.gz emd_28664_validation.cif.gz | 24.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28664 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28664 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28664 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28664 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28664.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28664.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HSV-1 polymerase holoenzyme bound by template and primer DNA, dNTP-free sample (editing conformation) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||
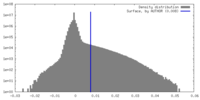
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: HSV-1 polymerase holoenzyme bound by template and primer...
| ファイル | emd_28664_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HSV-1 polymerase holoenzyme bound by template and primer DNA, dNTP-free sample (editing conformation) half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
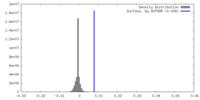
| 密度ヒストグラム |
-ハーフマップ: HSV-1 polymerase holoenzyme bound by template and primer...
| ファイル | emd_28664_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HSV-1 polymerase holoenzyme bound by template and primer DNA, dNTP-free sample (editing conformation) half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HSV-1 polymerase holoenzyme UL30:UL42 in complex with template an...
| 全体 | 名称: HSV-1 polymerase holoenzyme UL30:UL42 in complex with template and primer DNA (no mismatch), dNTP free |
|---|---|
| 要素 |
|
-超分子 #1: HSV-1 polymerase holoenzyme UL30:UL42 in complex with template an...
| 超分子 | 名称: HSV-1 polymerase holoenzyme UL30:UL42 in complex with template and primer DNA (no mismatch), dNTP free タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Herpes simplex virus type 1 polymerase holoenzyme UL30:UL42 in complex with template and primer DNA strands (no mismatch), dNTP free |
|---|---|
| 由来(天然) | 生物種:   Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) |
| 分子量 | 理論値: 197 KDa |
-分子 #1: HSV-1 UL30
| 分子 | 名称: HSV-1 UL30 / タイプ: protein_or_peptide / ID: 1 詳細: herpes simplex virus type 1 (KOS strain) DNA polymerase catalytic subunit UL30 with its N-terminal 42 residues deleted and replaced by an N-terminal poly-histidine tag 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:   Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス)株: KOS |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHNFYN PYLAPVGTQQ KPTGPTQRHT YYSECDEFRF IAPRVLDEDA PPEKRAGVHD GHLKRAPKVY CGGDERDVLR VGSGGFWPRR SRLWGGVDHA PAGFNPTVTV FHVYDILENV EHAYGMRAAQ FHARFMDAIT PTGTVITLLG LTPEGHRVAV HVYGTRQYFY ...文字列: HHHHHHNFYN PYLAPVGTQQ KPTGPTQRHT YYSECDEFRF IAPRVLDEDA PPEKRAGVHD GHLKRAPKVY CGGDERDVLR VGSGGFWPRR SRLWGGVDHA PAGFNPTVTV FHVYDILENV EHAYGMRAAQ FHARFMDAIT PTGTVITLLG LTPEGHRVAV HVYGTRQYFY MNKEEVDRHL QCRAPRDLCE RMAAALRESP GASFRGISAD HFEAEVVERT DVYYYETRPA LFYRVYVRSG RVLSYLCDNF CPAIKKYEGG VDATTRFILD NPGFVTFGWY RLKPGRNNTL AQPRAPMAFG TSSDVEFNCT ADNLAIEGGM SDLPAYKLMC FDIECKAGGE DELAFPVAGH PEDLVIQISC LLYDLSTTAL EHVLLFSLGS CDLPESHLNE LAARGLPTPV VLEFDSEFEM LLAFMTLVKQ YGPEFVTGYN IINFDWPFLL AKLTDIYKVP LDGYGRMNGR GVFRVWDIGQ SHFQKRSKIK VNGMVNIDMY GIITDKIKLS SYKLNAVAEA VLKDKKKDLS YRDIPAYYAT GPAQRGVIGE YCIQDSLLVG QLFFKFLPHL ELSAVARLAG INITRTIYDG QQIRVFTCLL RLADQKGFIL PDTQGRFRGA GGEAPKRPAA AREDEERPEE EGEDEDEREE GGGEREPEGA RETAGRHVGY QGARVLDPTS GFHVNPVVVF DFASLYPSII QAHNLCFSTL SLRADAVAHL EAGKDYLEIE VGGRRLFFVK AHVRESLLSI LLRDWLAMRK QIRSRIPQSS PEEAVLLDKQ QAAIKVVCNS VYGFTGVQHG LLPCLHVAAT VTTIGREMLL ATREYVHARW AAFEQLLADF PEAADMRAPG PYSMRIIYGD TDSIFVLCRG LTAAGLTAMG DKMASHISRA LFLPPIKLEC EKTFTKLLLI AKKKYIGVIY GGKMLIKGVD LVRKNNCAFI NRTSRALVDL LFYDDTVSGA AAALAERPAE EWLARPLPEG LQAFGAVLVD AHRRITDPER DIQDFVLTAE LSRHPRAYTN KRLAHLTVYY KLMARRAQVP SIKDRIPYVI VAQTREVEET VARLAALREL DAAAPGDEPA PPAALPSPAK RPRETPSHAD PPGGASKPRK LLVSELAEDP AYAIAHGVAL NTDYYFSHLL GAACVTFKAL FGNNAKITES LLKRFIPEVW HPPDDVAARL RAAGFGAVGA GATAEETRRM LHRAFDTLA GENBANK: GENBANK: AFE62858 |
-分子 #2: HSV-1 UL42
| 分子 | 名称: HSV-1 UL42 / タイプ: protein_or_peptide / ID: 2 詳細: Herpes simplex virus type 1 DNA polymerase processivity factor UL42 residues 1-340 (UL42delC340) followed by a PreScission protease cleavage site and a maltose binding protein (MBP) tag 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス)株: KOS |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTDSPGGVAP ASPVEDASDA SLGQPEEGAP CQVVLQGAEL NGILQAFAPL RTSLLDSLLV MGDRGILIHN TIFGEQVFLP LEHSQFSRYR WRGPTAAFLS LVDQKRSLLS VFRANQYPDL RRVELAITGQ APFRTLVQRI WTTTSDGEAV ELASETLMKR ELTSFVVLVP ...文字列: MTDSPGGVAP ASPVEDASDA SLGQPEEGAP CQVVLQGAEL NGILQAFAPL RTSLLDSLLV MGDRGILIHN TIFGEQVFLP LEHSQFSRYR WRGPTAAFLS LVDQKRSLLS VFRANQYPDL RRVELAITGQ APFRTLVQRI WTTTSDGEAV ELASETLMKR ELTSFVVLVP QGTPDVQLRL TRPQLTKVLN ATGADSATPT TFELGVNGKF SVFTTSTCVT FAAREEGVSS STSTQVQILS NALTKAGQAA ANAKTVYGEN THRTFSVVVD DCSMRAVLRR LQVAGGTLKF FLTTPVPSLC VTATGPNAVS AVFLLKPQKI CLDWLGHSQG SPSAGSSASR GENBANK: GENBANK: AFE62870 |
-分子 #3: Template DNA (50-mer)
| 分子 | 名称: Template DNA (50-mer) / タイプ: dna / ID: 3 / 詳細: tempalte DNA / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: CACACACACA CACACACAGA TCCCCGGGTA CCGAGCTCGA ATTCGTAATC |
-分子 #4: Primer DNA (32-mer)
| 分子 | 名称: Primer DNA (32-mer) / タイプ: dna / ID: 4 / 詳細: 3'-deoxyl primer DNA strand / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: GATTACGAAT TCGAGCTCGG TACCCGGGGA T(DOC) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 25 mM HEPES, pH 7.5, 150 mM NaCl, 2 mM tris(2-carboxyethyl)phosphine (TCEP) |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa 詳細: grids were glow discharged in Pelco easiGlow at 15 mA for 30 seconds under 0.39 mBar (i.e. 39 Pa) |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: 3 microliters of sample were blotted for 3 seconds with filter paper saturated under 100% humidity prior to plunging.. |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 77.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 6173 / 平均露光時間: 1.5 sec. / 平均電子線量: 53.11 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 60606 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | rigid body, minimization_global, local_grid_search, adp refinement |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 45.88 / 当てはまり具合の基準: correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)