+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Yeast NuA4 Histone Acetyltransferase Complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Histone Acetyltransferase / NuA4 / Yeast / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RHOB GTPase cycle / RHOA GTPase cycle / cellular bud neck contractile ring / mitotic actomyosin contractile ring contraction / piccolo histone acetyltransferase complex / vacuole inheritance / ascospore wall assembly / TTT Hsp90 cochaperone complex / actin cortical patch / SLIK (SAGA-like) complex ...RHOB GTPase cycle / RHOA GTPase cycle / cellular bud neck contractile ring / mitotic actomyosin contractile ring contraction / piccolo histone acetyltransferase complex / vacuole inheritance / ascospore wall assembly / TTT Hsp90 cochaperone complex / actin cortical patch / SLIK (SAGA-like) complex / Swr1 complex / kinetochore assembly / Ino80 complex / SAGA complex / SWI/SNF complex / establishment of cell polarity / DNA repair-dependent chromatin remodeling / NuA4 histone acetyltransferase complex / actin filament bundle / positive regulation of macroautophagy / protein secretion / chromosome organization / Ub-specific processing proteases / actin filament / structural constituent of cytoskeleton / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / endocytosis / transcription corepressor activity / nucleosome / actin cytoskeleton / chromatin organization / protein-containing complex assembly / histone binding / chromatin remodeling / DNA repair / DNA-templated transcription / chromatin binding / regulation of transcription by RNA polymerase II / regulation of DNA-templated transcription / chromatin / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / ATP hydrolysis activity / ATP binding / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
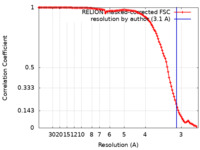
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Patel AB / Zukin SA / Nogales E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Structure and flexibility of the yeast NuA4 histone acetyltransferase complex. 著者: Stefan A Zukin / Matthew R Marunde / Irina K Popova / Katarzyna M Soczek / Eva Nogales / Avinash B Patel /  要旨: The NuA4 protein complex acetylates histones H4 and H2A to activate both transcription and DNA repair. We report the 3.1-Å resolution cryo-electron microscopy structure of the central hub of NuA4, ...The NuA4 protein complex acetylates histones H4 and H2A to activate both transcription and DNA repair. We report the 3.1-Å resolution cryo-electron microscopy structure of the central hub of NuA4, which flexibly tethers the histone acetyltransferase (HAT) and Trimer Independent of NuA4 involved in Transcription Interactions with Nucleosomes (TINTIN) modules. The hub contains the large Tra1 subunit and a core that includes Swc4, Arp4, Act1, Eaf1, and the C-terminal region of Epl1. Eaf1 stands out as the primary scaffolding factor that interacts with the Tra1, Swc4, and Epl1 subunits and contributes the conserved HSA helix to the Arp module. Using nucleosome-binding assays, we find that the HAT module, which is anchored to the core through Epl1, recognizes H3K4me3 nucleosomes with hyperacetylated H3 tails, while the TINTIN module, anchored to the core via Eaf1, recognizes nucleosomes that have hyperacetylated H2A and H4 tails. Together with the known interaction of Tra1 with site-specific transcription factors, our data suggest a model in which Tra1 recruits NuA4 to specific genomic sites then allowing the flexible HAT and TINTIN modules to select nearby nucleosomes for acetylation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28575.map.gz emd_28575.map.gz | 9.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28575-v30.xml emd-28575-v30.xml emd-28575.xml emd-28575.xml | 25.1 KB 25.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28575_fsc.xml emd_28575_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28575.png emd_28575.png | 58.9 KB | ||
| マスクデータ |  emd_28575_msk_1.map emd_28575_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28575.cif.gz emd-28575.cif.gz | 10 KB | ||
| その他 |  emd_28575_half_map_1.map.gz emd_28575_half_map_1.map.gz emd_28575_half_map_2.map.gz emd_28575_half_map_2.map.gz | 98.3 MB 98.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28575 http://ftp.pdbj.org/pub/emdb/structures/EMD-28575 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28575 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28575 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28575_validation.pdf.gz emd_28575_validation.pdf.gz | 686.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28575_full_validation.pdf.gz emd_28575_full_validation.pdf.gz | 686 KB | 表示 | |
| XML形式データ |  emd_28575_validation.xml.gz emd_28575_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_28575_validation.cif.gz emd_28575_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28575 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28575 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28575 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28575 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8escMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28575.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28575.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3404 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28575_msk_1.map emd_28575_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28575_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28575_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NuA4
| 全体 | 名称: NuA4 |
|---|---|
| 要素 |
|
-超分子 #1: NuA4
| 超分子 | 名称: NuA4 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.3 MDa |
-分子 #1: Transcription-associated protein 1
| 分子 | 名称: Transcription-associated protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 433.677281 KDa |
| 配列 | 文字列: MSLTEQIEQF ASRFRDDDAT LQSRYSTLSE LYDIMELLNS PEDYHFFLQA VIPLLLNQLK EVPISYDAHS PEQKLRNSML DIFNRCLMN QTFQPYAMEV LEFLLSVLPK ENEENGILCM KVLTTLFKSF KSILQDKLDS FIRIIIQIYK NTPNLINQTF Y EAGKAEQG ...文字列: MSLTEQIEQF ASRFRDDDAT LQSRYSTLSE LYDIMELLNS PEDYHFFLQA VIPLLLNQLK EVPISYDAHS PEQKLRNSML DIFNRCLMN QTFQPYAMEV LEFLLSVLPK ENEENGILCM KVLTTLFKSF KSILQDKLDS FIRIIIQIYK NTPNLINQTF Y EAGKAEQG DLDSPKEPQA DELLDEFSKN DEEKDFPSKQ SSTEPRFENS TSSNGLRSSM FSFKILSECP ITMVTLYSSY KQ LTSTSLP EFTPLIMNLL NIQIKQQQEA REQAESRGEH FTSISTEIIN RPAYCDFILA QIKATSFLAY VFIRGYAPEF LQD YVNFVP DLIIRLLQDC PSELSSARKE LLHATRHILS TNYKKLFLPK LDYLFDERIL IGNGFTMHET LRPLAYSTVA DFIH NIRSE LQLSEIEKTI KIYTGYLLDE SLALTVQIMS AKLLLNLVER ILKLGKENPQ EAPRAKKLLM IIIDSYMNRF KTLNR QYDT IMKYYGRYET HKKEKAEKLK NSIQDNDKES EEFMRKVLEP SDDDHLMPQP KKEDINDSPD VEMTESDKVV KNDVEM FDI KNYAPILLLP TPTNDPIKDA FYLYRTLMSF LKTIIHDLKV FNPPPNEYTV ANPKLWASVS RVFSYEEVIV FKDLFHE CI IGLKFFKDHN EKLSPETTKK HFDISMPSLP VSATKDAREL MDYLAFMFMQ MDNATFNEII EQELPFVYER MLEDSGLL H VAQSFLTSEI TSPNFAGILL RFLKGKLKDL GNVDFNTSNV LIRLFKLSFM SVNLFPNINE VVLLPHLNDL ILNSLKYST TAEEPLVYFY LIRTLFRSIG GGRFENLYRS IKPILQVLLQ SLNQMILTAR LPHERELYVE LCITVPVRLS VLAPYLPFLM KPLVFALQQ YPDLVSQGLR TLELCIDNLT AEYFDPIIEP VIDDVSKALF NLLQPQPFNH AISHNVVRIL GKLGGRNRQF L KPPTDLTE KTELDIDAIA DFKINGMPED VPLSVTPGIQ SALNILQSYK SDIHYRKSAY KYLTCVLLLM TKSSAEFPTN YT ELLKTAV NSIKLERIGI EKNFDLEPTV NKRDYSNQEN LFLRLLESVF YATSIKELKD DAMDLLNNLL DHFCLLQVNT TLL NKRNYN GTFNIDLKNP NFMLDSSLIL DAIPFALSYY IPEVREVGVL AYKRIYEKSC LIYGEELALS HSFIPELAKQ FIHL CYDET YYNKRGGVLG IKVLIDNVKS SSVFLKKYQY NLANGLLFVL KDTQSEAPSA ITDSAEKLLI DLLSITFADV KEEDL GNKV LENTLTDIVC ELSNANPKVR NACQKSLHTI SNLTGIPIVK LMDHSKQFLL SPIFAKPLRA LPFTMQIGNV DAITFC LSL PNTFLTFNEE LFRLLQESIV LADAEDESLS TNIQKTTEYS TSEQLVQLRI ACIKLLAIAL KNEEFATAQQ GNIRIRI LA VFFKTMLKTS PEIINTTYEA LKGSLAENSK LPKELLQNGL KPLLMNLSDH QKLTVPGLDA LSKLLELLIA YFKVEIGR K LLDHLTAWCR VEVLDTLFGQ DLAEQMPTKI IVSIINIFHL LPPQADMFLN DLLLKVMLLE RKLRLQLDSP FRTPLARYL NRFHNPVTEY FKKNMTLRQL VLFMCNIVQR PEAKELAEDF EKELDNFYDF YISNIPKNQV RVVSFFTNMV DLFNTMVITN GDEWLKKKG NMILKLKDML NLTLKTIKEN SFYIDHLQLN QSIAKFQALY LRFTELSERD QNPLLLDFID FSFSNGIKAS Y SLKKFIFH NIIASSNKEK QNNFINDATL FVLSDKCLDA RIFVLKNVIN STLIYEVATS GSLKSYLVED KKPKWLELLH NK IWKNSNA ILAYDVLDHH DLFRFELLQL SAIFIKADPE IIAEIKKDII KFCWNFIKLE DTLIKQSAYL VTSYFISKFD FPI KVVTQV FVALLRSSHV EARYLVKQSL DVLTPVLHER MNAAGTPDTW INWVKRVMVE NSSSQNNILY QFLISHPDLF FNSR DLFIS NIIHHMNKIT FMSNSNSDSH TLAIDLASLI LYWENKTLEI TNVNNTKTDS DGDVVMSDSK SDINPVEADT TAIIV DANN NSPISLHLRE ACTAFLIRYV CASNHRAIET ELGLRAINIL SELISDKHWT NVNVKLVYFE KFLIFQDLDS ENILYY CMN ALDVLYVFFK NKTKEWIMEN LPTIQNLLEK CIKSDHHDVQ EALQKVLQVI MKAIKAQGVS VIIEEESPGK TFIQMLT SV ITQDLQETSS VTAGVTLAWV LFMNFPDNIV PLLTPLMKTF SKLCKDHLSI SQPKDAMALE EARITTKLLE KVLYILSL K VSLLGDSRRP FLSTVALLID HSMDQNFLRK IVNMSRSWIF NTEIFPTVKE KAAILTKMLA FEIRGEPSLS KLFYEIVLK LFDQEHFNNT EITVRMEQPF LVGTRVEDIG IRKRFMTILD NSLERDIKER LYYVIRDQNW EFIADYPWLN QALQLLYGSF NREKELSLK NIYCLSPPSI LQEYLPENAE MVTEVNDLEL SNFVKGHIAS MQGLCRIISS DFIDSLIEIF YQDPKAIHRA W VTLFPQVY KSIPKNEKYG FVRSIITLLS KPYHTRQISS RTNVINMLLD SISKIESLEL PPHLVKYLAI SYNAWYQSIN IL ESIQSNT SIDNTKIIEA NEDALLELYV NLQEEDMFYG LWRRRAKYTE TNIGLSYEQI GLWDKAQQLY EVAQVKARSG ALP YSQSEY ALWEDNWIQC AEKLQHWDVL TELAKHEGFT DLLLECGWRV ADWNSDRDAL EQSVKSVMDV PTPRRQMFKT FLAL QNFAE SRKGDQEVRK LCDEGIQLSL IKWVSLPIRY TPAHKWLLHG FQQYMEFLEA TQIYANLHTT TVQNLDSKAQ EIKRI LQAW RDRLPNTWDD VNMWNDLVTW RQHAFQVINN AYLPLIPALQ QSNSNSNINT HAYRGYHEIA WVINRFAHVA RKHNMP DVC ISQLARIYTL PNIEIQEAFL KLREQAKCHY QNMNELTTGL DVISNTNLVY FGTVQKAEFF TLKGMFLSKL RAYEEAN QA FATAVQIDLN LAKAWAQWGF FNDRRLSEEP NNISFASNAI SCYLQAAGLY KNSKIRELLC RILWLISIDD ASGMLTNA F DSFRGEIPVW YWITFIPQLL TSLSHKEANM VRHILIRIAK SYPQALHFQL RTTKEDFAVI QRQTMAVMGD KPDTNDRNG RRQPWEYLQE LNNILKTAYP LLALSLESLV AQINDRFKST TDEDLFRLIN VLLIDGTLNY NRLPFPRKNP KLPENTEKNL VKFSTTLLA PYIRPKFNAD FIDNKPDYET YIKRLRYWRR RLENKLDRAS KKENLEVLCP HLSNFHHQKF EDIEIPGQYL L NKDNNVHF IKIARFLPTV DFVRGTHSSY RRLMIRGHDG SVHSFAVQYP AVRHSRREER MFQLYRLFNK SLSKNVETRR RS IQFNLPI AIPLSPQVRI MNDSVSFTTL HEIHNEFCKK KGFDPDDIQD FMADKLNAAH DDALPAPDMT ILKVEIFNSI QTM FVPSNV LKDHFTSLFT QFEDFWLFRK QFASQYSSFV FMSYMMMINN RTPHKIHVDK TSGNVFTLEM LPSRFPYERV KPLL KNHDL SLPPDSPIFH NNEPVPFRLT PNIQSLIGDS ALEGIFAVNL FTISRALIEP DNELNTYLAL FIRDEIISWF SNLHR PIIE NPQLREMVQT NVDLIIRKVA QLGHLNSTPT VTTQFILDCI GSAVSPRNLA RTDVNFMPWF UniProtKB: SAGA complex/NuA4 acetyltransferase complex subunit TRA1 |
-分子 #2: Actin-related protein 4
| 分子 | 名称: Actin-related protein 4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 54.894684 KDa |
| 配列 | 文字列: MSNAALQVYG GDEVSAVVID PGSYTTNIGY SGSDFPQSIL PSVYGKYTAD EGNKKIFSEQ SIGIPRKDYE LKPIIENGLV IDWDTAQEQ WQWALQNELY LNSNSGIPAL LTEPVWNSTE NRKKSLEVLL EGMQFEACYL APTSTCVSFA AGRPNCLVVD I GHDTCSVS ...文字列: MSNAALQVYG GDEVSAVVID PGSYTTNIGY SGSDFPQSIL PSVYGKYTAD EGNKKIFSEQ SIGIPRKDYE LKPIIENGLV IDWDTAQEQ WQWALQNELY LNSNSGIPAL LTEPVWNSTE NRKKSLEVLL EGMQFEACYL APTSTCVSFA AGRPNCLVVD I GHDTCSVS PIVDGMTLSK STRRNFIAGK FINHLIKKAL EPKEIIPLFA IKQRKPEFIK KTFDYEVDKS LYDYANNRGF FQ ECKETLC HICPTKTLEE TKTELSSTAK RSIESPWNEE IVFDNETRYG FAEELFLPKE DDIPANWPRS NSGVVKTWRN DYV PLKRTK PSGVNKSDKK VTPTEEKEQE AVSKSTSPAA NSADTPNETG KRPLEEEKPP KENNELIGLA DLVYSSIMSS DVDL RATLA HNVVLTGGTS SIPGLSDRLM TELNKILPSL KFRILTTGHT IERQYQSWLG GSILTSLGTF HQLWVGKKEY EEVGV ERLL NDRFR UniProtKB: Actin-related protein 4 |
-分子 #3: Actin
| 分子 | 名称: Actin / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.735547 KDa |
| 配列 | 文字列: MDSEVAALVI DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGIMVGMGQK DSYVGDEAQS KRGILTLRYP IEHGIVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP MNPKSNREKM TQIMFETFNV PAFYVSIQAV LSLYSSGRTT GIVLDSGDGV T HVVPIYAG ...文字列: MDSEVAALVI DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGIMVGMGQK DSYVGDEAQS KRGILTLRYP IEHGIVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP MNPKSNREKM TQIMFETFNV PAFYVSIQAV LSLYSSGRTT GIVLDSGDGV T HVVPIYAG FSLPHAILRI DLAGRDLTDY LMKILSERGY SFSTTAEREI VRDIKEKLCY VALDFEQEMQ TAAQSSSIEK SY ELPDGQV ITIGNERFRA PEALFHPSVL GLESAGIDQT TYNSIMKCDV DVRKELYGNI VMSGGTTMFP GIAERMQKEI TAL APSSMK VKIIAPPERK YSVWIGGSIL ASLTTFQQMW ISKQEYDESG PSIVHHKCF UniProtKB: Actin |
-分子 #4: Enhancer of polycomb-like protein 1
| 分子 | 名称: Enhancer of polycomb-like protein 1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 96.889867 KDa |
| 配列 | 文字列: MPTPSNAIEI NDGSHKSGRS TRRSGSRSAH DDGLDSFSKG DSGAGASAGS SNSRFRHRKI SVKQHLKIYL PNDLKHLDKD ELQQREVVE IETGVEKNEE KEVHLHRILQ MGSGHTKHKD YIPTPDASMT WNEYDKFYTG SFQETTSYIK FSATVEDCCG T NYNMDERD ...文字列: MPTPSNAIEI NDGSHKSGRS TRRSGSRSAH DDGLDSFSKG DSGAGASAGS SNSRFRHRKI SVKQHLKIYL PNDLKHLDKD ELQQREVVE IETGVEKNEE KEVHLHRILQ MGSGHTKHKD YIPTPDASMT WNEYDKFYTG SFQETTSYIK FSATVEDCCG T NYNMDERD ETFLNEQVNK GSSDILTEDE FEILCSSFEH AIHERQPFLS MDPESILSFE ELKPTLIKSD MADFNLRNQL NH EINSHKT HFITQFDPVS QMNTRPLIQL IEKFGSKIYD YWRERKIEVN GYEIFPQLKF ERPGEKEEID PYVCFRRREV RHP RKTRRI DILNSQRLRA LHQELKNAKD LALLVAKREN VSLNWINDEL KIFDQRVKIK NLKRSLNISG EDDDLINHKR KRPT IVTVE QREAELRKAE LKRAAAAAAA AKAKNNKRNN QLEDKSSRLT KQQQQQLLQQ QQQQQQNALK TENGKQLANA SSSST SQPI TSHVYVKLPS SKIPDIVLED VDALLNSKEK NARKFVQEKM EKRKIEDADV FFNLTDDPFN PVFDMSLPKN FSTSNV PFA SIASSKFQID RSFYSSHLPE YLKGISDDIR IYDSNGRSRN KDNYNLDTKR IKKTELYDPF QENLEIHSRE YPIKFRK RV GRSNIKYVDR MPNFTTSSTK SACSLMDFVD FDSIEKEQYS REGSNDTDSI NVYDSKYDEF VRLYDKWKYD SPQNEYGI K FSDEPARLNQ ISNDTQVIRF GTMLGTKSYE QLREATIKYR RDYITRLKQK HIQHLQQQQQ QQQQQQQQAQ QQKQKSQNN NSNSSNSLKK LNDSLINSEA KQNSSITQKN SS UniProtKB: Enhancer of polycomb-like protein 1 |
-分子 #5: Chromatin modification-related protein EAF1
| 分子 | 名称: Chromatin modification-related protein EAF1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 112.667008 KDa |
| 配列 | 文字列: MSSRPSSAVP NSASLSEDQS SDRSKFPKAD DLIDERDRKL TELYCVSRLN QLLELTDENK LRKEIDAFLK KNDIRRGIRF DEASLPKLL HTAATPITKK KLKDVNLINV PNQRLSDSKM SRELPENSEN VSVKSESHFV PSHDNSIREN MMDSLRPAEK T GGMWNKRP ...文字列: MSSRPSSAVP NSASLSEDQS SDRSKFPKAD DLIDERDRKL TELYCVSRLN QLLELTDENK LRKEIDAFLK KNDIRRGIRF DEASLPKLL HTAATPITKK KLKDVNLINV PNQRLSDSKM SRELPENSEN VSVKSESHFV PSHDNSIREN MMDSLRPAEK T GGMWNKRP LESTMGGEEE RHEKRQKMQS QSLESSNNSE MASLPISPRP PVPNALAHYT YYENIEYPPA DPTEVQPAVK FK DPLIKNI MAKEIDTSDH YNENNVDALE TVFLLMNDYI PSKIPQALPL AELKYMSQTL PLINLIPRAH KALTTNIINN ALN EARITV VGSRIEELRR LGLWSLRQPK RFIDPWKQHN THQNILLEEA KWMQADFKEG HKYKVAICTA MAQAIKDYWT YGEI CCVKR KTLLPGKENK LSDDGRISEK SGRPSDTSRN DSDISIAGKD DIGIIANVDD ITEKESAAAN DNDENGKNEA GAKSD FDFA DGLLSQEGAH DQIISSIDTK LLLKKPSSSS EVVLIQHEVA ASSALIETEE SKKELAPPFK LSIFVDELNT FEKTLI QDL PLYNGINEER PKKDDSLPFI PISKSVVSLD DNGFYKLLER QLIDEEPSIS QLSKRRGMFY GNRRNHYLRP PAVPSLR YL QNRTPTIWLS EDDQELVKNI NTYGYNWELI SAHMTHRLTY SYLSNIERRT PWQCFERFVQ LNERFNFSDL KGPRAHSA Q QWLIEAHKFQ QRQNRRISPL GVNTESIQRG HRRLRWASMF EAIRKCMKKR ENTPRPNPTQ PRKPLDCKNM KVPTPAEMS LLKAQRDEAL RRDIQLRRTV KNRLQQRQQQ SQQAHSSRAQ SPIPSNGKSS SNLARNGQAS APRPNQKQYT EQDIIESYSR KLLEQKPDI GPEMALKAAK NYYRTLREQQ QQLKQHQIQQ QRQQLQEESS HVQQLQQLQP GSQAPPPKSS PSQSSLSNIS N INSAPRIK SPTPQEILQR FQKQ UniProtKB: Chromatin modification-related protein EAF1 |
-分子 #6: SWR1-complex protein 4
| 分子 | 名称: SWR1-complex protein 4 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 55.297684 KDa |
| 配列 | 文字列: MSSSDIFDVL NIKQKSRSPT NGQVSVPSSS AANRPKPQVT GMQRELFNLL GENQPPVVIK SGNNFKEKML STSKPSPWSF VEFKANNSV TLRHWVKGSK ELIGDTPKES PYSKFNQHLS IPSFTKEEYE AFMNENEGTQ KSVESEKNHN ENFTNEKKDE S KNSWSFEE ...文字列: MSSSDIFDVL NIKQKSRSPT NGQVSVPSSS AANRPKPQVT GMQRELFNLL GENQPPVVIK SGNNFKEKML STSKPSPWSF VEFKANNSV TLRHWVKGSK ELIGDTPKES PYSKFNQHLS IPSFTKEEYE AFMNENEGTQ KSVESEKNHN ENFTNEKKDE S KNSWSFEE IEYLFNLCKK YDLRWFLIFD RYSYNNSRTL EDLKEKFYYT CRNYFKASDP SNPLLSSLNF SAEKEIERKK YL QRLLSRS AAEIAEEEAL VVESKKFEMA AKRTLAERES LLRLLDSPHS DQTITQYLTS QGMSQLYNAL LADKTRKRKH DLN IPENPW MKQQQQFAQH RQLQQLNVKK SEVKENLSPK KTKRQRQEMQ TALKRKSESA YAEQLLKDFN SDERKALGVI THGE KLSPG VYLRSTKLST FKPALQNKIL AILQELSLPS RPVMPSFDVM ERQEELLKKI NTLIDLKKHV DKYEAGMSIT K UniProtKB: SWR1-complex protein 4 |
-分子 #7: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #8: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 8 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)