+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
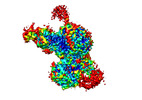
| タイトル | Cryo-EM structure of PRC2 in complex with the long isoform of AEBP2 | ||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | Polycomb repressive complex 2 Histone methyltransferase / GENE REGULATION | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hepatocyte homeostasis / cellular response to trichostatin A / regulation of gliogenesis / negative regulation of striated muscle cell differentiation / regulation of kidney development / [histone H3]-lysine27 N-trimethyltransferase / sex chromatin / CAF-1 complex / negative regulation of keratinocyte differentiation / histone H3K27 trimethyltransferase activity ...hepatocyte homeostasis / cellular response to trichostatin A / regulation of gliogenesis / negative regulation of striated muscle cell differentiation / regulation of kidney development / [histone H3]-lysine27 N-trimethyltransferase / sex chromatin / CAF-1 complex / negative regulation of keratinocyte differentiation / histone H3K27 trimethyltransferase activity / negative regulation of retinoic acid receptor signaling pathway / response to tetrachloromethane / cerebellar cortex development / primary miRNA binding / random inactivation of X chromosome / regulatory ncRNA-mediated heterochromatin formation / skeletal muscle satellite cell maintenance involved in skeletal muscle regeneration / histone H3K27 methyltransferase activity / facultative heterochromatin formation / positive regulation of cell cycle G1/S phase transition / NuRD complex / NURF complex / regulation of cell fate specification / negative regulation of stem cell population maintenance / DNA replication-dependent chromatin assembly / ESC/E(Z) complex / Transcription of E2F targets under negative control by p107 (RBL1) and p130 (RBL2) in complex with HDAC1 / regulation of stem cell differentiation / RSC-type complex / negative regulation of stem cell differentiation / pronucleus / chromatin silencing complex / protein-lysine N-methyltransferase activity / Polo-like kinase mediated events / cardiac muscle hypertrophy in response to stress / Transcription of E2F targets under negative control by DREAM complex / G1 to G0 transition / positive regulation of dendrite development / histone H3 methyltransferase activity / synaptic transmission, GABAergic / DNA methylation-dependent constitutive heterochromatin formation / histone methyltransferase activity / lncRNA binding / negative regulation of G1/S transition of mitotic cell cycle / ATPase complex / spinal cord development / negative regulation of gene expression, epigenetic / Sin3-type complex / G1/S-Specific Transcription / positive regulation of stem cell population maintenance / oligodendrocyte differentiation / Transcriptional Regulation by E2F6 / negative regulation of transcription elongation by RNA polymerase II / RNA Polymerase I Transcription Initiation / histone deacetylase complex / G0 and Early G1 / negative regulation of cell differentiation / positive regulation of protein serine/threonine kinase activity / subtelomeric heterochromatin formation / ribonucleoprotein complex binding / pericentric heterochromatin / Transcriptional regulation of brown and beige adipocyte differentiation by EBF2 / Cyclin E associated events during G1/S transition / positive regulation of epithelial to mesenchymal transition / RNA polymerase II core promoter sequence-specific DNA binding / Cyclin A:Cdk2-associated events at S phase entry / nucleosome binding / : / keratinocyte differentiation / protein localization to chromatin / Regulation of TP53 Activity through Acetylation / Deposition of new CENPA-containing nucleosomes at the centromere / negative regulation of cytokine production involved in inflammatory response / positive regulation of GTPase activity / positive regulation of MAP kinase activity / B cell differentiation / SUMOylation of chromatin organization proteins / enzyme activator activity / negative regulation of cell migration / liver regeneration / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / hippocampus development / Regulation of PTEN gene transcription / PRC2 methylates histones and DNA / transcription corepressor binding / Regulation of endogenous retroelements by KRAB-ZFP proteins / Defective pyroptosis / Regulation of endogenous retroelements by Piwi-interacting RNAs (piRNAs) / HDACs deacetylate histones / stem cell differentiation / transcription coregulator activity / promoter-specific chromatin binding / negative regulation of transforming growth factor beta receptor signaling pathway / regulation of circadian rhythm / brain development / protein-DNA complex / protein modification process / PKMTs methylate histone lysines / chromatin DNA binding / cellular response to hydrogen peroxide 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||
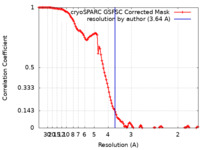
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.64 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Boudes M / Zhang Q / Flanigan SF / Davidovich C | ||||||||||||||||||||||||
| 資金援助 |  オーストラリア, 7件 オーストラリア, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: To be updated 著者: Boudes M / Zhang Q / Flanigan SF / Davidovich C | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28547.map.gz emd_28547.map.gz | 88.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28547-v30.xml emd-28547-v30.xml emd-28547.xml emd-28547.xml | 29.8 KB 29.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28547_fsc.xml emd_28547_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28547.png emd_28547.png | 73.3 KB | ||
| Filedesc metadata |  emd-28547.cif.gz emd-28547.cif.gz | 8.8 KB | ||
| その他 |  emd_28547_additional_1.map.gz emd_28547_additional_1.map.gz emd_28547_half_map_1.map.gz emd_28547_half_map_1.map.gz emd_28547_half_map_2.map.gz emd_28547_half_map_2.map.gz | 168.1 MB 165.1 MB 165.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28547 http://ftp.pdbj.org/pub/emdb/structures/EMD-28547 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28547 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28547 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28547_validation.pdf.gz emd_28547_validation.pdf.gz | 938.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28547_full_validation.pdf.gz emd_28547_full_validation.pdf.gz | 937.8 KB | 表示 | |
| XML形式データ |  emd_28547_validation.xml.gz emd_28547_validation.xml.gz | 20.8 KB | 表示 | |
| CIF形式データ |  emd_28547_validation.cif.gz emd_28547_validation.cif.gz | 27 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28547 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28547 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28547 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28547 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8eqvMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28547.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28547.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
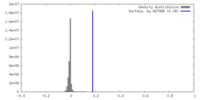
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_28547_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
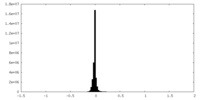
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28547_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
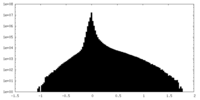
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28547_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
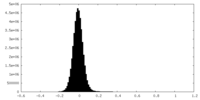
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of PRC2 with long isoform of AEBP2
| 全体 | 名称: Ternary complex of PRC2 with long isoform of AEBP2 |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of PRC2 with long isoform of AEBP2
| 超分子 | 名称: Ternary complex of PRC2 with long isoform of AEBP2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Histone-binding protein RBBP4
| 分子 | 名称: Histone-binding protein RBBP4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 47.709527 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MADKEAAFDD AVEERVINEE YKIWKKNTPF LYDLVMTHAL EWPSLTAQWL PDVTRPEGKD FSIHRLVLGT HTSDEQNHLV IASVQLPND DAQFDASHYD SEKGEFGGFG SVSGKIEIEI KINHEGEVNR ARYMPQNPCI IATKTPSSDV LVFDYTKHPS K PDPSGECN ...文字列: MADKEAAFDD AVEERVINEE YKIWKKNTPF LYDLVMTHAL EWPSLTAQWL PDVTRPEGKD FSIHRLVLGT HTSDEQNHLV IASVQLPND DAQFDASHYD SEKGEFGGFG SVSGKIEIEI KINHEGEVNR ARYMPQNPCI IATKTPSSDV LVFDYTKHPS K PDPSGECN PDLRLRGHQK EGYGLSWNPN LSGHLLSASD DHTICLWDIS AVPKEGKVVD AKTIFTGHTA VVEDVSWHLL HE SLFGSVA DDQKLMIWDT RSNNTSKPSH SVDAHTAEVN CLSFNPYSEF ILATGSADKT VALWDLRNLK LKLHSFESHK DEI FQVQWS PHNETILASS GTDRRLNVWD LSKIGEEQSP EDAEDGPPEL LFIHGGHTAK ISDFSWNPNE PWVICSVSED NIMQ VWQMA ENIYNDEDPE GSVDPEGQGS UniProtKB: Histone-binding protein RBBP4 |
-分子 #2: Zinc finger protein AEBP2
| 分子 | 名称: Zinc finger protein AEBP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 54.535496 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAAITDMAD LEELSRLSPL PPGSPGSAAR GRAEPPEEEE EEEEEEEEAE AEAVAALLLN GGSGGGGGGG GGGVGGGEAE TMSEPSPES ASQAGEDEDE EEDDEEEEDE SSSSGGGEEE SSAESLVGSS GGSSSDETRS LSPGAASSSS GDGDGKEGLE E PKGPRGSQ ...文字列: MAAAITDMAD LEELSRLSPL PPGSPGSAAR GRAEPPEEEE EEEEEEEEAE AEAVAALLLN GGSGGGGGGG GGGVGGGEAE TMSEPSPES ASQAGEDEDE EEDDEEEEDE SSSSGGGEEE SSAESLVGSS GGSSSDETRS LSPGAASSSS GDGDGKEGLE E PKGPRGSQ GGGGGGSSSS SVVSSGGDEG YGTGGGGSSA TSGGRRGSLE MSSDGEPLSR MDSEDSISST IMDVDSTISS GR STPAMMN GQGSTTSSSK NIAYNCCWDQ CQACFNSSPD LADHIRSIHV DGQRGGVFVC LWKGCKVYNT PSTSQSWLQR HML THSGDK PFKCVVGGCN ASFASQGGLA RHVPTHFSQQ NSSKVSSQPK AKEESPSKAG MNKRRKLKNK RRRSLPRPHD FFDA QTLDA IRHRAICFNL SAHIESLGKG HSVVFHSTVI AKRKEDSGKI KLLLHWMPED ILPDVWVNES ERHQLKTKVV HLSKL PKDT ALLLDPNIYR TMPQKRLKRT LIRKVFNLYL SKQ UniProtKB: Zinc finger protein AEBP2 |
-分子 #3: Polycomb protein EED
| 分子 | 名称: Polycomb protein EED / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 50.267691 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSEREVSTAP AGTDMPAAKK QKLSSDENSN PDLSGDENDD AVSIESGTNT ERPDTPTNTP NAPGRKSWGK GKWKSKKCKY SFKCVNSLK EDHNQPLFGV QFNWHSKEGD PLVFATVGSN RVTLYECHSQ GEIRLLQSYV DADADENFYT CAWTYDSNTS H PLLAVAGS ...文字列: MSEREVSTAP AGTDMPAAKK QKLSSDENSN PDLSGDENDD AVSIESGTNT ERPDTPTNTP NAPGRKSWGK GKWKSKKCKY SFKCVNSLK EDHNQPLFGV QFNWHSKEGD PLVFATVGSN RVTLYECHSQ GEIRLLQSYV DADADENFYT CAWTYDSNTS H PLLAVAGS RGIIRIINPI TMQCIKHYVG HGNAINELKF HPRDPNLLLS VSKDHALRLW NIQTDTLVAI FGGVEGHRDE VL SADYDLL GEKIMSCGMD HSLKLWRINS KRMMNAIKES YDYNPNKTNR PFISQKIHFP DFSTRDIHRN YVDCVRWLGD LIL SKSCEN AIVCWKPGKM EDDIDKIKPS ESNVTILGRF DYSQCDIWYM RFSMDFWQKM LALGNQVGKL YVWDLEVEDP HKAK CTTLT HHKCGAAIRQ TSFSRDSSIL IAVCDDASIW RWDRLR UniProtKB: Polycomb protein EED |
-分子 #4: Histone-lysine N-methyltransferase EZH2
| 分子 | 名称: Histone-lysine N-methyltransferase EZH2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO / EC番号: [histone H3]-lysine27 N-trimethyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 85.492297 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGQTGKKSEK GPVCWRKRVK SEYMRLRQLK RFRRADEVKS MFSSNRQKIL ERTEILNQEW KQRRIQPVHI LTSVSSLRGT RECSVTSDL DFPTQVIPLK TLNAVASVPI MYSWSPLQQN FMVEDETVLH NIPYMGDEVL DQDGTFIEEL IKNYDGKVHG D RECGFIND ...文字列: MGQTGKKSEK GPVCWRKRVK SEYMRLRQLK RFRRADEVKS MFSSNRQKIL ERTEILNQEW KQRRIQPVHI LTSVSSLRGT RECSVTSDL DFPTQVIPLK TLNAVASVPI MYSWSPLQQN FMVEDETVLH NIPYMGDEVL DQDGTFIEEL IKNYDGKVHG D RECGFIND EIFVELVNAL GQYNDDDDDD DGDDPEEREE KQKDLEDHRD DKESRPPRKF PSDKIFEAIS SMFPDKGTAE EL KEKYKEL TEQQLPGALP PECTPNIDGP NAKSVQREQS LHSFHTLFCR RCFKYDCFLH PFHATPNTYK RKNTETALDN KPC GPQCYQ HLEGAKEFAA ALTAERIKTP PKRPGGRRRG RLPNNSSRPS TPTINVLESK DTDSDREAGT ETGGENNDKE EEEK KDETS SSSEANSRCQ TPIKMKPNIE PPENVEWSGA EASMFRVLIG TYYDNFCAIA RLIGTKTCRQ VYEFRVKESS IIAPA PAED VDTPPRKKKR KHRLWAAHCR KIQLKKDGSS NHVYNYQPCD HPRQPCDSSC PCVIAQNFCE KFCQCSSECQ NRFPGC RCK AQCNTKQCPC YLAVRECDPD LCLTCGAADH WDSKNVSCKN CSIQRGSKKH LLLAPSDVAG WGIFIKDPVQ KNEFISE YC GEIISQDEAD RRGKVYDKYM CSFLFNLNND FVVDATRKGN KIRFANHSVN PNCYAKVMMV NGDHRIGIFA KRAIQTGE E LFFDYRYSQA DALKYVGIER EMEIP UniProtKB: Histone-lysine N-methyltransferase EZH2 |
-分子 #5: Polycomb protein SUZ12
| 分子 | 名称: Polycomb protein SUZ12 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 83.181922 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAPQKHGGGG GGGSGPSAGS GGGGFGGSAA VAAATASGGK SGGGSCGGGG SYSASSSSSA AAAAGAAVLP VKKPKMEHVQ ADHELFLQA FEKPTQIYRF LRTRNLIAPI FLHRTLTYMS HRNSRTNIKR KTFKVDDMLS KVEKMKGEQE SHSLSAHLQL T FTGFFHKN ...文字列: MAPQKHGGGG GGGSGPSAGS GGGGFGGSAA VAAATASGGK SGGGSCGGGG SYSASSSSSA AAAAGAAVLP VKKPKMEHVQ ADHELFLQA FEKPTQIYRF LRTRNLIAPI FLHRTLTYMS HRNSRTNIKR KTFKVDDMLS KVEKMKGEQE SHSLSAHLQL T FTGFFHKN DKPSPNSENE QNSVTLEVLL VKVCHKKRKD VSCPIRQVPT GKKQVPLNPD LNQTKPGNFP SLAVSSNEFE PS NSHMVKS YSLLFRVTRP GRREFNGMIN GETNENIDVN EELPARRKRN REDGEKTFVA QMTVFDKNRR LQLLDGEYEV AMQ EMEECP ISKKRATWET ILDGKRLPPF ETFSQGPTLQ FTLRWTGETN DKSTAPIAKP LATRNSESLH QENKPGSVKP TQTI AVKES LTTDLQTRKE KDTPNENRQK LRIFYQFLYN NNTRQQTEAR DDLHCPWCTL NCRKLYSLLK HLKLCHSRFI FNYVY HPKG ARIDVSINEC YDGSYAGNPQ DIHRQPGFAF SRNGPVKRTP ITHILVCRPK RTKASMSEFL ESEDGEVEQQ RTYSSG HNR LYFHSDTCLP LRPQEMEVDS EDEKDPEWLR EKTITQIEEF SDVNEGEKEV MKLWNLHVMK HGFIADNQMN HACMLFV EN YGQKIIKKNL CRNFMLHLVS MHDFNLISIM SIDKAVTKLR EMQQKLEKGE SASPANEEIT EEQNGTANGF SEINSKEK A LETDSVSGVS KQSKKQKL UniProtKB: Polycomb protein SUZ12 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.4 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 200 mM NaCl, 20 mM HEPES pH 7.5, 1 mM TCEP, 0.01% NP-40 | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 詳細: 0.24 mBar, 120 s, 10 mAmp | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting 3 seconds. | |||||||||||||||
| 詳細 | Prior to cryo-EM sample preparation, complexes were PEGylated at 0.9 mg/mL with 5 mM MS(PEG)4 Methyl-PEG-NHS-Ester (ThermoFisher Scientific) for 2 h at 4oC. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum ER / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)