+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P nanoparticle by localized reconstruction | |||||||||
 マップデータ マップデータ | BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P nanoparticle by localized reconstruction - Main Map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 10.4 Å | |||||||||
 データ登録者 データ登録者 | Antanasijevic A / Zhang YN / Zhu J / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Single-component multilayered self-assembling protein nanoparticles presenting glycan-trimmed uncleaved prefusion optimized envelope trimmers as HIV-1 vaccine candidates. 著者: Yi-Nan Zhang / Jennifer Paynter / Aleksandar Antanasijevic / Joel D Allen / Mor Eldad / Yi-Zong Lee / Jeffrey Copps / Maddy L Newby / Linling He / Deborah Chavez / Pat Frost / Anna Goodroe / ...著者: Yi-Nan Zhang / Jennifer Paynter / Aleksandar Antanasijevic / Joel D Allen / Mor Eldad / Yi-Zong Lee / Jeffrey Copps / Maddy L Newby / Linling He / Deborah Chavez / Pat Frost / Anna Goodroe / John Dutton / Robert Lanford / Christopher Chen / Ian A Wilson / Max Crispin / Andrew B Ward / Jiang Zhu /   要旨: Uncleaved prefusion-optimized (UFO) design can stabilize diverse HIV-1 envelope glycoproteins (Envs). Single-component, self-assembling protein nanoparticles (1c-SApNP) can display 8 or 20 native- ...Uncleaved prefusion-optimized (UFO) design can stabilize diverse HIV-1 envelope glycoproteins (Envs). Single-component, self-assembling protein nanoparticles (1c-SApNP) can display 8 or 20 native-like Env trimers as vaccine candidates. We characterize the biophysical, structural, and antigenic properties of 1c-SApNPs that present the BG505 UFO trimer with wildtype and modified glycans. For 1c-SApNPs, glycan trimming improves recognition of the CD4 binding site without affecting broadly neutralizing antibodies (bNAbs) to major glycan epitopes. In mice, rabbits, and nonhuman primates, glycan trimming increases the frequency of vaccine responders (FVR) and steers antibody responses away from immunodominant glycan holes and glycan patches. The mechanism of vaccine-induced immunity is examined in mice. Compared with the UFO trimer, the multilayered E2p and I3-01v9 1c-SApNPs show 420 times longer retention in lymph node follicles, 20-32 times greater presentation on follicular dendritic cell dendrites, and up-to-4 times stronger germinal center reactions. These findings can inform future HIV-1 vaccine development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28543.map.gz emd_28543.map.gz | 16.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28543-v30.xml emd-28543-v30.xml emd-28543.xml emd-28543.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28543.png emd_28543.png | 51.4 KB | ||
| その他 |  emd_28543_half_map_1.map.gz emd_28543_half_map_1.map.gz emd_28543_half_map_2.map.gz emd_28543_half_map_2.map.gz | 16.9 MB 16.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28543 http://ftp.pdbj.org/pub/emdb/structures/EMD-28543 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28543 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28543 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28543_validation.pdf.gz emd_28543_validation.pdf.gz | 701.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28543_full_validation.pdf.gz emd_28543_full_validation.pdf.gz | 700.6 KB | 表示 | |
| XML形式データ |  emd_28543_validation.xml.gz emd_28543_validation.xml.gz | 10.2 KB | 表示 | |
| CIF形式データ |  emd_28543_validation.cif.gz emd_28543_validation.cif.gz | 11.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28543 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28543 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28543 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28543 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28543.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28543.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P nanoparticle by localized reconstruction - Main Map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P...
| ファイル | emd_28543_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
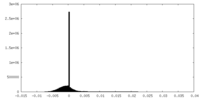
| 注釈 | BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P nanoparticle by localized reconstruction - Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P...
| ファイル | emd_28543_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
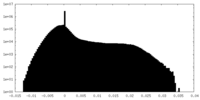
| 注釈 | BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P nanoparticle by localized reconstruction - Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BG505 UFO map obtained by localized reconstruction
| 全体 | 名称: BG505 UFO map obtained by localized reconstruction |
|---|---|
| 要素 |
|
-超分子 #1: BG505 UFO map obtained by localized reconstruction
| 超分子 | 名称: BG505 UFO map obtained by localized reconstruction / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all 詳細: The map was generated by localized reconstruction of the BG505 UFO part of the BG505 UFO-10GS-I3-01v9-L7P nanoparticle dataset |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: BG505 UFO-10GS-I3-01v9-L7P - Antigenic portion
| 分子 | 名称: BG505 UFO-10GS-I3-01v9-L7P - Antigenic portion / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARSRAEN LWVTVYYGVP VWKDAETTLF CASDAKAYDT EKHNVWATHA CVPTDPNPQE IHLENVTEEF NMWKNNMVEQ MHTDIISLWD QSLKPCVKLT PLCVTLQCTN VTNNITDDMR GELKNCSFNM TTELRDKKQK ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARSRAEN LWVTVYYGVP VWKDAETTLF CASDAKAYDT EKHNVWATHA CVPTDPNPQE IHLENVTEEF NMWKNNMVEQ MHTDIISLWD QSLKPCVKLT PLCVTLQCTN VTNNITDDMR GELKNCSFNM TTELRDKKQK VYSLFYRLDV VQINENQGNR SNNSNKEYRL INCNTSAITQ ACPKVSFEPI PIHYCAPAGF AILKCKDKKF NGTGPCPSVS TVQCTHGIKP VVSTQLLLNG SLAEEEVMIR SENITNNAKN ILVQFNTPVQ INCTRPNNNT RKSIRIGPGQ AFYATGDIIG DIRQAHCNVS KATWNETLGK VVKQLRKHFG NNTIIRFANS SGGDLEVTTH SFNCGGEFFY CNTSGLFNST WISNTSVQGS NSTGSNDSIT LPCRIKQIIN MWQRIGQAMY APPIQGVIRC VSNITGLILT RDGGSTNSTT ETFRPGGGDM RDNWRSELYK YKVVKIEPLG VAPTRCKRRV VGGGGGSGGG GSAVGIGAVF LGFLGAAGST MGAASMTLTV QARNLLSGNP DWLPDMTVWG IKQLQARVLA VERYLRDQQL LGIWGCSGKL ICCTNVPWNS SWSNRNLSEI WDNMTWLQWD KEISNYTQII YGLLEESQNQ QEKNEQDLLA LDASGGGGSG GGGSMKMEEL FKKHKIVAVL RANSVEEAKM KALAVFVGGV HLIEITFTVP DADTVIKELS FLKELGAIIG AGTVTSVEQC RKAVESGAEF IVSPHLDEEI SQFCKEKGVF YMPGVMTPTE LVKAMKLGHT ILKLFPGEVV GPQFVKAMKG PFPNVKFVPT GGVNLDNVCE WFKAGVLAVG VGSALVKGTI AEVAAKAAAF VEKIRGCTEG GGGSSPAVDI GDRLDELEKA LEALSAEDGH DDVGQRLESL LRRWNSRRAD GSAKFVAAWT LKAAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: TBS buffer prepared from a 10X stock | |||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting time varied between 3 and 7 seconds.. | |||||||||
| 詳細 | The nanoparticle was expressed in ExpiCHO cells and purified using a combination of immuno-affinity chromatography and SEC. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-48 / 撮影したグリッド数: 1 / 実像数: 1200 / 平均露光時間: 12.0 sec. / 平均電子線量: 50.4 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)