+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a potent anti-malarial antibody L9 in complex with Plasmodium falciparum circumsporozoite protein (PfCSP)(dominant class) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Anti-Malarial / PfCSP / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell surface binding / symbiont entry into host / entry into host cell by a symbiont-containing vacuole / heparan sulfate proteoglycan binding / side of membrane / cell surface / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
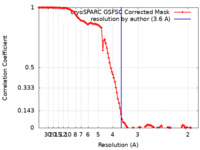
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Tripathi P / Kwong PD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Cryo-EM structures of anti-malarial antibody L9 with circumsporozoite protein reveal trimeric L9 association and complete 27-residue epitope. 著者: Prabhanshu Tripathi / Michael F Bender / Haotian Lei / Lais Da Silva Pereira / Chen-Hsiang Shen / Brian Bonilla / Marlon Dillon / Li Ou / Marie Pancera / Lawrence T Wang / Baoshan Zhang / ...著者: Prabhanshu Tripathi / Michael F Bender / Haotian Lei / Lais Da Silva Pereira / Chen-Hsiang Shen / Brian Bonilla / Marlon Dillon / Li Ou / Marie Pancera / Lawrence T Wang / Baoshan Zhang / Facundo D Batista / Azza H Idris / Robert A Seder / Peter D Kwong /  要旨: Monoclonal antibody L9 recognizes the Plasmodium falciparum circumsporozoite protein (PfCSP) and is highly protective following controlled human malaria challenge. To gain insight into its function, ...Monoclonal antibody L9 recognizes the Plasmodium falciparum circumsporozoite protein (PfCSP) and is highly protective following controlled human malaria challenge. To gain insight into its function, we determined cryoelectron microscopy (cryo-EM) structures of L9 in complex with full-length PfCSP and assessed how this recognition influenced protection by wild-type and mutant L9s. Cryo-EM reconstructions at 3.6- and 3.7-Å resolution revealed L9 to recognize PfCSP as an atypical trimer. Each of the three L9s in the trimer directly recognized an Asn-Pro-Asn-Val (NPNV) tetrapeptide on PfCSP and interacted homotypically to facilitate L9-trimer assembly. We analyzed peptides containing different repeat tetrapeptides for binding to wild-type and mutant L9s to delineate epitope and homotypic components of L9 recognition; we found both components necessary for potent malaria protection. Last, we found the 27-residue stretch recognized by L9 to be highly conserved in P. falciparum isolates, suggesting the newly revealed complete L9 epitope to be an attractive vaccine target. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28192.map.gz emd_28192.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28192-v30.xml emd-28192-v30.xml emd-28192.xml emd-28192.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28192_fsc.xml emd_28192_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28192.png emd_28192.png | 36.8 KB | ||
| Filedesc metadata |  emd-28192.cif.gz emd-28192.cif.gz | 6.6 KB | ||
| その他 |  emd_28192_half_map_1.map.gz emd_28192_half_map_1.map.gz emd_28192_half_map_2.map.gz emd_28192_half_map_2.map.gz | 116.2 MB 116.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28192 http://ftp.pdbj.org/pub/emdb/structures/EMD-28192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28192 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28192_validation.pdf.gz emd_28192_validation.pdf.gz | 848 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28192_full_validation.pdf.gz emd_28192_full_validation.pdf.gz | 847.5 KB | 表示 | |
| XML形式データ |  emd_28192_validation.xml.gz emd_28192_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_28192_validation.cif.gz emd_28192_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28192 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28192 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ek1MC  8ekaC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28192.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28192.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.92 Å | ||||||||||||||||||||||||||||||||||||
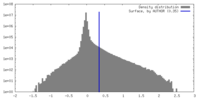
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28192_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
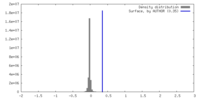
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28192_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
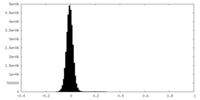
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PfCSP in complex with antibody L9
| 全体 | 名称: PfCSP in complex with antibody L9 |
|---|---|
| 要素 |
|
-超分子 #1: PfCSP in complex with antibody L9
| 超分子 | 名称: PfCSP in complex with antibody L9 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: L9 Fab heavy chain
| 分子 | 名称: L9 Fab heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.475436 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVKLVESGGG VVQPGRSLRL SCEASGFIFS TYGMHWVRQA PGKGLEWVAV IWFDGSNIYY ADSVKGRFTI SRDNSKNTVF MQMDSLRAE DTAVYYCHRN FYDGSGPFDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS ...文字列: QVKLVESGGG VVQPGRSLRL SCEASGFIFS TYGMHWVRQA PGKGLEWVAV IWFDGSNIYY ADSVKGRFTI SRDNSKNTVF MQMDSLRAE DTAVYYCHRN FYDGSGPFDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV EPKSCDKTH |
-分子 #2: L9 Fab light chain
| 分子 | 名称: L9 Fab light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.597254 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPST LSASVGDRVT ITCRASQFIS RWLAWYQQKP GKAPKLLIYK ASSLESGVPS RFSGSGSETH FTLTISSLQP DDVATYYCQ EYTSYGRTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPST LSASVGDRVT ITCRASQFIS RWLAWYQQKP GKAPKLLIYK ASSLESGVPS RFSGSGSETH FTLTISSLQP DDVATYYCQ EYTSYGRTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #3: Circumsporozoite protein
| 分子 | 名称: Circumsporozoite protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: isolate 3D7 |
| 分子量 | 理論値: 37.807488 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QEYQSYGSSS NTRVLNELNY DNAGTNLYNE LEMNYYGKQE NWYSLSSNSA SLGENDDGNN EDNEKLRKPK HKKLKQPADG NPDPNANPN VDPNANPNVD PNANPNVDPN ANPNANPNAN PNANPNANPN ANPNANPNAN PNANPNANPN ANPNANPNAN P NANPNANP ...文字列: QEYQSYGSSS NTRVLNELNY DNAGTNLYNE LEMNYYGKQE NWYSLSSNSA SLGENDDGNN EDNEKLRKPK HKKLKQPADG NPDPNANPN VDPNANPNVD PNANPNVDPN ANPNANPNAN PNANPNANPN ANPNANPNAN PNANPNANPN ANPNANPNAN P NANPNANP NANPNANPNV DPNANPNANP NANPNANPNA NPNANPNANP NANPNANPNA NPNANPNANP NANPNANPNA NP NANPNAN PNANPNKNNQ GNGQGHNMPN DPNRNVDENA NANSAVKNNN NEEPSDKHIK EYLNKIQNSL STEWSPCSVT CGN GIQVRI KPGSANKPKD ELDYANDIEK KICKMEKCS UniProtKB: Circumsporozoite protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 63.7 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 45000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 147.9 |
|---|---|
| 得られたモデル |  PDB-8ek1: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)