+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of focused PtuA(dimer) and PtuB(monomer) complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PtuA / IMMUNE SYSTEM | |||||||||
| 機能・相同性 | Retron Ec78 putative HNH endonuclease-like / TIGR02646 family protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
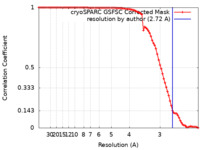
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.72 Å | |||||||||
 データ登録者 データ登録者 | Shen ZF / Fu TM | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: PtuA and PtuB assemble into an inflammasome-like oligomer for anti-phage defense. 著者: Yuanyuan Li / Zhangfei Shen / Mengyuan Zhang / Xiao-Yuan Yang / Sean P Cleary / Jiale Xie / Ila A Marathe / Marius Kostelic / Jacelyn Greenwald / Anthony D Rish / Vicki H Wysocki / Chong Chen ...著者: Yuanyuan Li / Zhangfei Shen / Mengyuan Zhang / Xiao-Yuan Yang / Sean P Cleary / Jiale Xie / Ila A Marathe / Marius Kostelic / Jacelyn Greenwald / Anthony D Rish / Vicki H Wysocki / Chong Chen / Qiang Chen / Tian-Min Fu / Yamei Yu /   要旨: Escherichia coli Septu system, an anti-phage defense system, comprises two components: PtuA and PtuB. PtuA contains an ATPase domain, while PtuB is predicted to function as a nuclease. Here we show ...Escherichia coli Septu system, an anti-phage defense system, comprises two components: PtuA and PtuB. PtuA contains an ATPase domain, while PtuB is predicted to function as a nuclease. Here we show that PtuA and PtuB form a stable complex with a 6:2 stoichiometry. Cryo-electron microscopy structure of PtuAB reveals a distinctive horseshoe-like configuration. PtuA adopts a hexameric arrangement, organized as an asymmetric trimer of dimers, contrasting the ring-like structure by other ATPases. Notably, the three pairs of PtuA dimers assume distinct conformations and fulfill unique roles in recruiting PtuB. Our functional assays have further illuminated the importance of the oligomeric assembly of PtuAB in anti-phage defense. Moreover, we have uncovered that ATP molecules can directly bind to PtuA and inhibit the activities of PtuAB. Together, the assembly and function of the Septu system shed light on understanding other ATPase-containing systems in bacterial immunity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28048.map.gz emd_28048.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28048-v30.xml emd-28048-v30.xml emd-28048.xml emd-28048.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28048_fsc.xml emd_28048_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28048.png emd_28048.png | 41.6 KB | ||
| マスクデータ |  emd_28048_msk_1.map emd_28048_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28048.cif.gz emd-28048.cif.gz | 6.5 KB | ||
| その他 |  emd_28048_half_map_1.map.gz emd_28048_half_map_1.map.gz emd_28048_half_map_2.map.gz emd_28048_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28048 http://ftp.pdbj.org/pub/emdb/structures/EMD-28048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28048 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28048_validation.pdf.gz emd_28048_validation.pdf.gz | 896.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28048_full_validation.pdf.gz emd_28048_full_validation.pdf.gz | 896.4 KB | 表示 | |
| XML形式データ |  emd_28048_validation.xml.gz emd_28048_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_28048_validation.cif.gz emd_28048_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28048 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28048 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28048 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28048 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28048.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28048.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28048_msk_1.map emd_28048_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28048_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28048_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 6 ptuA form as a complex
| 全体 | 名称: 6 ptuA form as a complex |
|---|---|
| 要素 |
|
-超分子 #1: 6 ptuA form as a complex
| 超分子 | 名称: 6 ptuA form as a complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: PtuA
| 分子 | 名称: PtuA / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.189656 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRIDKLSLLN FRCFKQLDIT FDEHITILVA PNGAGKTTVL DAVRLALFPF IRGFDASLYV KDKSLAIRTE DLRLIYRQEA LNMEMSSPA KITATGEWAS GKTATWMLDK RGEQPPHEDK MAAQLTRWGE QLQKRVREEH SLQQVELPLM LYLGTARLWY Q ERYEKQPT ...文字列: MRIDKLSLLN FRCFKQLDIT FDEHITILVA PNGAGKTTVL DAVRLALFPF IRGFDASLYV KDKSLAIRTE DLRLIYRQEA LNMEMSSPA KITATGEWAS GKTATWMLDK RGEQPPHEDK MAAQLTRWGE QLQKRVREEH SLQQVELPLM LYLGTARLWY Q ERYEKQPT EQRLDNSAFS RLSGYDDCLS ATSNYKQFEQ WYSWLWLSYR EHQITQLESP SAKLKEGVRV QRMKEAIQAI QQ AINCLTQ QVTGWHDLEY SASHNQQLVM SHPQYGKIPL SQLSDGLRNA VAMVADIAFR CVKLNPHLQN DAALKTQGIV LID EVDMFL HPAWQQQIIQ SLRSAFPQIQ FIVTTHSPQV LSTVKRESIR LLEQDENGNG KALMPLGATY GEPSNDVLQS VMGV DPQPA VKEKADLQKL TGWVDQGKYD EPKTQQLMVA LEVALGEKHP QLQRLQRSIA RQRLLKGKAQ |
-分子 #2: PtuB
| 分子 | 名称: PtuB / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 28.136291 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRHVIKTQLG TVALLTAHEN PPQDADQSTR RWRNFRRDKA AVMVQLINEQ YHLCCYSEIR SDLRGLGYHI EHVENKSQHP ERTFDYQNL AASALDSGEN GGLSSLKGKN AFGGHAQGKQ DVVDMAKFIH CHIRDCSRYF AYLSDGRIVP ADELNAQETE N AQYTIDLL ...文字列: MRHVIKTQLG TVALLTAHEN PPQDADQSTR RWRNFRRDKA AVMVQLINEQ YHLCCYSEIR SDLRGLGYHI EHVENKSQHP ERTFDYQNL AASALDSGEN GGLSSLKGKN AFGGHAQGKQ DVVDMAKFIH CHIRDCSRYF AYLSDGRIVP ADELNAQETE N AQYTIDLL NLNSGFLQTE RRNHWEELEQ LFDEHIEKDW DLQQLLQLDL VSTPDHKLHE FFSITRQFFQ QEAEQVLQSH AP ALI UniProtKB: TIGR02646 family protein |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)