+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of P74-26 tail-like tubes | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | tail tube protein / bacteriophage / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Phage tail tube protein-like / Phage tail tube protein / Major capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Oshimavirus P7426 (ウイルス) Oshimavirus P7426 (ウイルス) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.81 Å | |||||||||
 データ登録者 データ登録者 | Agnello E / Pajak J / Liu X / Kelch B | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2023 ジャーナル: J Biol Chem / 年: 2023タイトル: Conformational dynamics control assembly of an extremely long bacteriophage tail tube. 著者: Emily Agnello / Joshua Pajak / Xingchen Liu / Brian A Kelch /  要旨: Tail tube assembly is an essential step in the lifecycle of long-tailed bacteriophages. Limited structural and biophysical information has impeded an understanding of assembly and stability of their ...Tail tube assembly is an essential step in the lifecycle of long-tailed bacteriophages. Limited structural and biophysical information has impeded an understanding of assembly and stability of their long, flexible tail tubes. The hyperthermophilic phage P74-26 is particularly intriguing as it has the longest tail of any known virus (nearly 1 μm) and is the most thermostable known phage. Here, we use structures of the P74-26 tail tube along with an in vitro system for studying tube assembly kinetics to propose the first molecular model for the tail tube assembly of long-tailed phages. Our high-resolution cryo-EM structure provides insight into how the P74-26 phage assembles through flexible loops that fit into neighboring rings through tight "ball-and-socket"-like interactions. Guided by this structure, and in combination with mutational, light scattering, and molecular dynamics simulations data, we propose a model for the assembly of conserved tube-like structures across phage and other entities possessing tail tube-like proteins. We propose that formation of a full ring promotes the adoption of a tube elongation-competent conformation among the flexible loops and their corresponding sockets, which is further stabilized by an adjacent ring. Tail assembly is controlled by the cooperative interaction of dynamic intraring and interring contacts. Given the structural conservation among tail tube proteins and tail-like structures, our model can explain the mechanism of high-fidelity assembly of long, stable tubes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28042.map.gz emd_28042.map.gz | 53.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28042-v30.xml emd-28042-v30.xml emd-28042.xml emd-28042.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28042.png emd_28042.png | 125.4 KB | ||
| Filedesc metadata |  emd-28042.cif.gz emd-28042.cif.gz | 5.7 KB | ||
| その他 |  emd_28042_half_map_1.map.gz emd_28042_half_map_1.map.gz emd_28042_half_map_2.map.gz emd_28042_half_map_2.map.gz | 95.1 MB 95.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28042 http://ftp.pdbj.org/pub/emdb/structures/EMD-28042 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28042 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28042 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28042_validation.pdf.gz emd_28042_validation.pdf.gz | 931.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28042_full_validation.pdf.gz emd_28042_full_validation.pdf.gz | 931.3 KB | 表示 | |
| XML形式データ |  emd_28042_validation.xml.gz emd_28042_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_28042_validation.cif.gz emd_28042_validation.cif.gz | 16 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28042 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28042 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28042 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28042 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8edxMC  8ed0C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28042.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28042.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28042_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
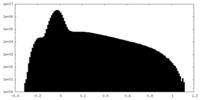
| 投影像・断面図 |
| ||||||||||||
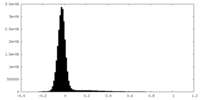
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28042_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
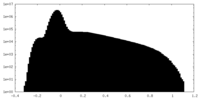
| 投影像・断面図 |
| ||||||||||||
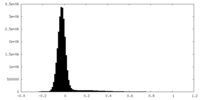
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : In vitro-assembled tail-like tubes of tail tube protein gp93
| 全体 | 名称: In vitro-assembled tail-like tubes of tail tube protein gp93 |
|---|---|
| 要素 |
|
-超分子 #1: In vitro-assembled tail-like tubes of tail tube protein gp93
| 超分子 | 名称: In vitro-assembled tail-like tubes of tail tube protein gp93 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Oshimavirus P7426 (ウイルス) Oshimavirus P7426 (ウイルス) |
-分子 #1: Tail Tube Protein gp93
| 分子 | 名称: Tail Tube Protein gp93 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Oshimavirus P7426 (ウイルス) Oshimavirus P7426 (ウイルス) |
| 分子量 | 理論値: 37.955738 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRGVDTFLAF KEQADLKTPA TLASLAAGDF LAFNSESLSG RQQVIQSRAI RRMPMRQIAY TANGTVEAGG AVEFTTSNYV LKKLLPLIF HSKTGQEDDP DGDGATFTLV NGGVLTPFTA FVGFDGPEGK YVRRFFGAKV NQATFSARVN DMLNLNLDVQ A IGKDILQP ...文字列: MRGVDTFLAF KEQADLKTPA TLASLAAGDF LAFNSESLSG RQQVIQSRAI RRMPMRQIAY TANGTVEAGG AVEFTTSNYV LKKLLPLIF HSKTGQEDDP DGDGATFTLV NGGVLTPFTA FVGFDGPEGK YVRRFFGAKV NQATFSARVN DMLNLNLDVQ A IGKDILQP GDPGWVNVTP VYPGGDEEYA YVFYQARVLI KAGDMADLAE LPVESFDLTI NHNLNTNRYR LGSIYRQSLD EG VTEVTGT FTLDAAVKSI SGPALNLTGG TAHDPAFLEK VALYGKYAAL KLEFIDPTRE VAEGVPCRLT IHLPFVRLEE PDF QVRDPG VITGSARFNA YETISVTHVA KF UniProtKB: Major capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. 詳細: Grids were glow discharged at 25 mA for 60 sec with negative polarity. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 38.4312 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 45000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 使用したクラス数: 1 想定した対称性 - らせんパラメータ - Δz: 40.0 Å 想定した対称性 - らせんパラメータ - ΔΦ: -44 ° 想定した対称性 - らせんパラメータ - 軸対称性: C3 (3回回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 2.81 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 395357 |
|---|---|
| Segment selection | 選択した数: 619907 |
| 初期モデル | モデルのタイプ: NONE / 詳細: Ab initio map generated in cryoSPARC |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)