+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
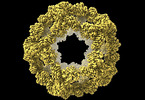
| タイトル | Structure of a nanoparticle with icosahedral symmetry | |||||||||
 マップデータ マップデータ | Post-processed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Protein design / nanoparticle / DE NOVO PROTEIN | |||||||||
| 機能・相同性 | 4-hydroxy-2-oxoglutarate aldolase / (R,S)-4-hydroxy-2-oxoglutarate aldolase activity / 2-dehydro-3-deoxy-phosphogluconate aldolase / 2-dehydro-3-deoxy-phosphogluconate aldolase activity / KDPG/KHG aldolase / KDPG and KHG aldolase / Aldolase-type TIM barrel / 4-Hydroxy-2-oxoglutarate aldolase / 2-dehydro-3-deoxyphosphogluconate aldolase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Thermotoga maritima MSB8 (バクテリア) Thermotoga maritima MSB8 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | McCarthy S / Gonen S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Heliyon / 年: 2022 ジャーナル: Heliyon / 年: 2022タイトル: Improved interface packing and design opportunities revealed by CryoEM analysis of a designed protein nanocage. 著者: Stephen McCarthy / Shane Gonen /  要旨: Symmetric protein assemblies play important roles in nature which makes them an attractive target for engineering. symmetric protein complexes can be created through computational protein design to ...Symmetric protein assemblies play important roles in nature which makes them an attractive target for engineering. symmetric protein complexes can be created through computational protein design to tailor their properties from first principles, and recently several protein nanocages have been created by bringing together protein components through hydrophobic interactions. Accurate experimental structures of newly-developed proteins are essential to validate their design, improve assembly stability, and tailor downstream applications. We describe the CryoEM structure of the nanocage I3-01, at an overall resolution of 3.5 Å. I3-01, comprising 60 aldolase subunits arranged with icosahedral symmetry, has resisted high-resolution characterization. Some key differences between the refined structure and the original design are identified, such as improved packing of hydrophobic sidechains, providing insight to the resistance of I3-01 to high-resolution averaging. Based on our analysis, we suggest factors important in the design and structural processing of new assemblies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28027.map.gz emd_28027.map.gz | 17.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28027-v30.xml emd-28027-v30.xml emd-28027.xml emd-28027.xml | 13.4 KB 13.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28027.png emd_28027.png | 128.3 KB | ||
| Filedesc metadata |  emd-28027.cif.gz emd-28027.cif.gz | 5.1 KB | ||
| その他 |  emd_28027_half_map_1.map.gz emd_28027_half_map_1.map.gz emd_28027_half_map_2.map.gz emd_28027_half_map_2.map.gz | 140.9 MB 140.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28027 http://ftp.pdbj.org/pub/emdb/structures/EMD-28027 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28027 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28027 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ed3MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28027.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28027.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||
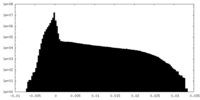
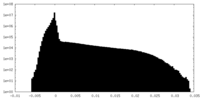
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half-map 1
| ファイル | emd_28027_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
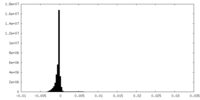
| 密度ヒストグラム |
-ハーフマップ: Half-map 2
| ファイル | emd_28027_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
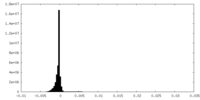
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : I3-01
| 全体 | 名称: I3-01 |
|---|---|
| 要素 |
|
-超分子 #1: I3-01
| 超分子 | 名称: I3-01 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima MSB8 (バクテリア) Thermotoga maritima MSB8 (バクテリア) |
-分子 #1: Designed I3-01 icosahedron
| 分子 | 名称: Designed I3-01 icosahedron / タイプ: protein_or_peptide / ID: 1 / コピー数: 60 / 光学異性体: LEVO / EC番号: 2-dehydro-3-deoxy-phosphogluconate aldolase |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima MSB8 (バクテリア) / 株: ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8 Thermotoga maritima MSB8 (バクテリア) / 株: ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8 |
| 分子量 | 理論値: 21.660584 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEELFKKHKI VAVLRANSVE EAKKKALAVF LGGVHLIEIT FTVPDADTVI KELSFLKEMG AIIGAGTVTS VEQCRKAVES GAEFIVSPH LDEEISQFCK EKGVFYMPGV MTPTELVKAM KLGHTILKLF PGEVVGPQFV KAMKGPFPNV KFVPTGGVNL D NVCEWFKA ...文字列: MEELFKKHKI VAVLRANSVE EAKKKALAVF LGGVHLIEIT FTVPDADTVI KELSFLKEMG AIIGAGTVTS VEQCRKAVES GAEFIVSPH LDEEISQFCK EKGVFYMPGV MTPTELVKAM KLGHTILKLF PGEVVGPQFV KAMKGPFPNV KFVPTGGVNL D NVCEWFKA GVLAVGVGSA LVKGTPVEVA EKAKAFVEKI RGC UniProtKB: 4-Hydroxy-2-oxoglutarate aldolase / 2-dehydro-3-deoxyphosphogluconate aldolase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 2.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Stochastic gradient descent from 2D averages |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 147349 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8ed3: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)