+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Vigna radiata supercomplex I+III2 (full bridge) | |||||||||
 マップデータ マップデータ | Vigna radiata supercomplex I III2 (SC I III2) composite map of full supercomplex (bridged class 1). | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | respiratory supercomplex / nadh-cyt c oxidoreductase / membrane complex / ELECTRON TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報TIM22 mitochondrial import inner membrane insertion complex / mitochondrial processing peptidase / P450-containing electron transport chain / protein insertion into mitochondrial inner membrane / NADH dehydrogenase complex / respiratory chain complex III / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / mitochondrial electron transport, ubiquinol to cytochrome c / NADH dehydrogenase activity ...TIM22 mitochondrial import inner membrane insertion complex / mitochondrial processing peptidase / P450-containing electron transport chain / protein insertion into mitochondrial inner membrane / NADH dehydrogenase complex / respiratory chain complex III / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / mitochondrial electron transport, ubiquinol to cytochrome c / NADH dehydrogenase activity / acyl binding / electron transport coupled proton transport / acyl carrier activity / NADH:ubiquinone reductase (H+-translocating) / mitochondrial respiratory chain complex I assembly / mitochondrial electron transport, NADH to ubiquinone / respiratory chain complex I / NADH dehydrogenase (ubiquinone) activity / quinone binding / ATP synthesis coupled electron transport / aerobic respiration / chloroplast / respiratory electron transport chain / electron transport chain / mitochondrial membrane / metalloendopeptidase activity / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / mRNA processing / 4 iron, 4 sulfur cluster binding / response to oxidative stress / electron transfer activity / mitochondrial inner membrane / mRNA binding / heme binding / protein-containing complex binding / mitochondrion / proteolysis / metal ion binding / nucleus / membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Vigna radiata (リョクトウ) Vigna radiata (リョクトウ) | |||||||||
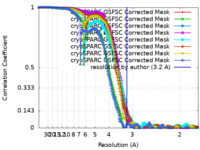
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Maldonado M / Letts JA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2023 ジャーナル: Nat Plants / 年: 2023タイトル: Plant-specific features of respiratory supercomplex I + III from Vigna radiata. 著者: M Maldonado / Z Fan / K M Abe / J A Letts /  要旨: The last steps of cellular respiration-an essential metabolic process in plants-are carried out by mitochondrial oxidative phosphorylation. This process involves a chain of multi-subunit membrane ...The last steps of cellular respiration-an essential metabolic process in plants-are carried out by mitochondrial oxidative phosphorylation. This process involves a chain of multi-subunit membrane protein complexes (complexes I-V) that form higher-order assemblies called supercomplexes. Although supercomplexes are the most physiologically relevant form of the oxidative phosphorylation complexes, their functions and structures remain mostly unknown. Here we present the cryogenic electron microscopy structure of the supercomplex I + III from Vigna radiata (mung bean). The structure contains the full subunit complement of complex I, including a newly assigned, plant-specific subunit. It also shows differences in the mitochondrial processing peptidase domain of complex III relative to a previously determined supercomplex with complex IV. The supercomplex interface, while reminiscent of that in other organisms, is plant specific, with a major interface involving complex III's mitochondrial processing peptidase domain and no participation of complex I's bridge domain. The complex I structure suggests that the bridge domain sets the angle between the enzyme's two arms, limiting large-scale conformational changes. Moreover, complex I's catalytic loops and its response in active-to-deactive assays suggest that, in V. radiata, the resting complex adopts a non-canonical state and can sample deactive- or open-like conformations even in the presence of substrate. This study widens our understanding of the possible conformations and behaviour of complex I and supercomplex I + III. Further studies of complex I and its supercomplexes in diverse organisms are needed to determine the universal and clade-specific mechanisms of respiration. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27934.map.gz emd_27934.map.gz | 81.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27934-v30.xml emd-27934-v30.xml emd-27934.xml emd-27934.xml | 83.2 KB 83.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27934_fsc.xml emd_27934_fsc.xml emd_27934_fsc_2.xml emd_27934_fsc_2.xml emd_27934_fsc_3.xml emd_27934_fsc_3.xml emd_27934_fsc_4.xml emd_27934_fsc_4.xml emd_27934_fsc_5.xml emd_27934_fsc_5.xml emd_27934_fsc_6.xml emd_27934_fsc_6.xml emd_27934_fsc_7.xml emd_27934_fsc_7.xml emd_27934_fsc_8.xml emd_27934_fsc_8.xml | 19.9 KB 19.9 KB 19.9 KB 19.9 KB 19.9 KB 19.9 KB 20 KB 19.9 KB | 表示 表示 表示 表示 表示 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27934.png emd_27934.png | 86.8 KB | ||
| マスクデータ |  emd_27934_msk_1.map emd_27934_msk_1.map | 824 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-27934.cif.gz emd-27934.cif.gz | 16.9 KB | ||
| その他 |  emd_27934_additional_1.map.gz emd_27934_additional_1.map.gz emd_27934_half_map_1.map.gz emd_27934_half_map_1.map.gz emd_27934_half_map_2.map.gz emd_27934_half_map_2.map.gz | 779.1 MB 764 MB 764 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27934 http://ftp.pdbj.org/pub/emdb/structures/EMD-27934 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27934 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27934 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27934_validation.pdf.gz emd_27934_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27934_full_validation.pdf.gz emd_27934_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_27934_validation.xml.gz emd_27934_validation.xml.gz | 8.5 KB | 表示 | |
| CIF形式データ |  emd_27934_validation.cif.gz emd_27934_validation.cif.gz | 5.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27934 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27934 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27934 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27934 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8e73MC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27934.map.gz / 形式: CCP4 / 大きさ: 87.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27934.map.gz / 形式: CCP4 / 大きさ: 87.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Vigna radiata supercomplex I III2 (SC I III2) composite map of full supercomplex (bridged class 1). | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27934_msk_1.map emd_27934_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Vigna radiata supercomplex I III2 bridged class 1
| ファイル | emd_27934_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Vigna radiata supercomplex I III2 bridged class 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Vigna radiata supercomplex I III2 bridged class 1 half map B
| ファイル | emd_27934_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Vigna radiata supercomplex I III2 bridged class 1_half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Vigna radiata supercomplex I III2 bridged class 1 half map A
| ファイル | emd_27934_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Vigna radiata supercomplex I III2 bridged class 1_half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Vigna radiata supercomplex I+III2 (full bridge)
+超分子 #1: Vigna radiata supercomplex I+III2 (full bridge)
+分子 #1: MPP-beta
+分子 #2: MPP-alpha (protomer 1)
+分子 #3: COB (cyt b)
+分子 #4: CYC1 (cyt c1)
+分子 #5: UCR1 (Rieske iron-sulfur protein subunit)
+分子 #6: QCR7
+分子 #7: QCR8
+分子 #8: QCR6
+分子 #9: QCR9
+分子 #10: QCR10 (UCRY)
+分子 #11: MPP-alpha (protomer 2)
+分子 #12: Nad1
+分子 #13: Nad2
+分子 #14: Nad3
+分子 #15: Nad4
+分子 #16: Nad4L
+分子 #17: Nad5
+分子 #18: Nad6
+分子 #19: NDUA1
+分子 #20: NDUA2
+分子 #21: NDUA3
+分子 #22: NDUA5
+分子 #23: NDUA6
+分子 #24: NDUA7
+分子 #25: NDUA8
+分子 #26: NDUA9
+分子 #27: NDUA11
+分子 #28: NDUA12
+分子 #29: NDUA13
+分子 #30: NDUAB1-beta
+分子 #31: NDUAB1-alpha
+分子 #32: NDUB2
+分子 #33: NDUB3
+分子 #34: NDUB4
+分子 #35: NDUB7
+分子 #36: NDUB8
+分子 #37: NDUB9
+分子 #38: NDUB10
+分子 #39: NDUB11
+分子 #40: NDUFX
+分子 #41: NDUC2
+分子 #42: NDUP2
+分子 #43: NDUCA1
+分子 #44: NDUCA2
+分子 #45: NDUCAL2
+分子 #46: NDUS1
+分子 #47: NDUS2
+分子 #48: NDUS3
+分子 #49: NDUS4
+分子 #50: NDUS5
+分子 #51: NDUS6
+分子 #52: NDUS7
+分子 #53: NDUS8
+分子 #54: NDUP1
+分子 #55: NDUP4
+分子 #56: NDUB6
+分子 #57: NDUV1
+分子 #58: NDUV2
+分子 #59: NDUX1
+分子 #60: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
+分子 #61: CARDIOLIPIN
+分子 #62: PROTOPORPHYRIN IX CONTAINING FE
+分子 #63: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #64: HEME C
+分子 #65: UBIQUINONE-10
+分子 #66: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE
+分子 #67: S-[2-({N-[(2S)-2-hydroxy-3,3-dimethyl-4-(phosphonooxy)butanoyl]-b...
+分子 #68: FE (III) ION
+分子 #69: ZINC ION
+分子 #70: IRON/SULFUR CLUSTER
+分子 #71: FE2/S2 (INORGANIC) CLUSTER
+分子 #72: FLAVIN MONONUCLEOTIDE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.55 mg/mL |
|---|---|
| 緩衝液 | pH: 7.7 詳細: 0.2% digitonin, 30 mM HEPES pH 7.7, 150 mM potassium acetate, 1 mM EDTA, 0.002% PMSF |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: LEICA EM GP / 詳細: 4 ul, 20 seconds pre-blot, blot 4 seconds. |
| 詳細 | Digitonin-extracted, amphipol (A8-35)stabilized |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 25712 / 平均露光時間: 3.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8e73: |
 ムービー
ムービー コントローラー
コントローラー






































 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)